Figure 1
Download original image
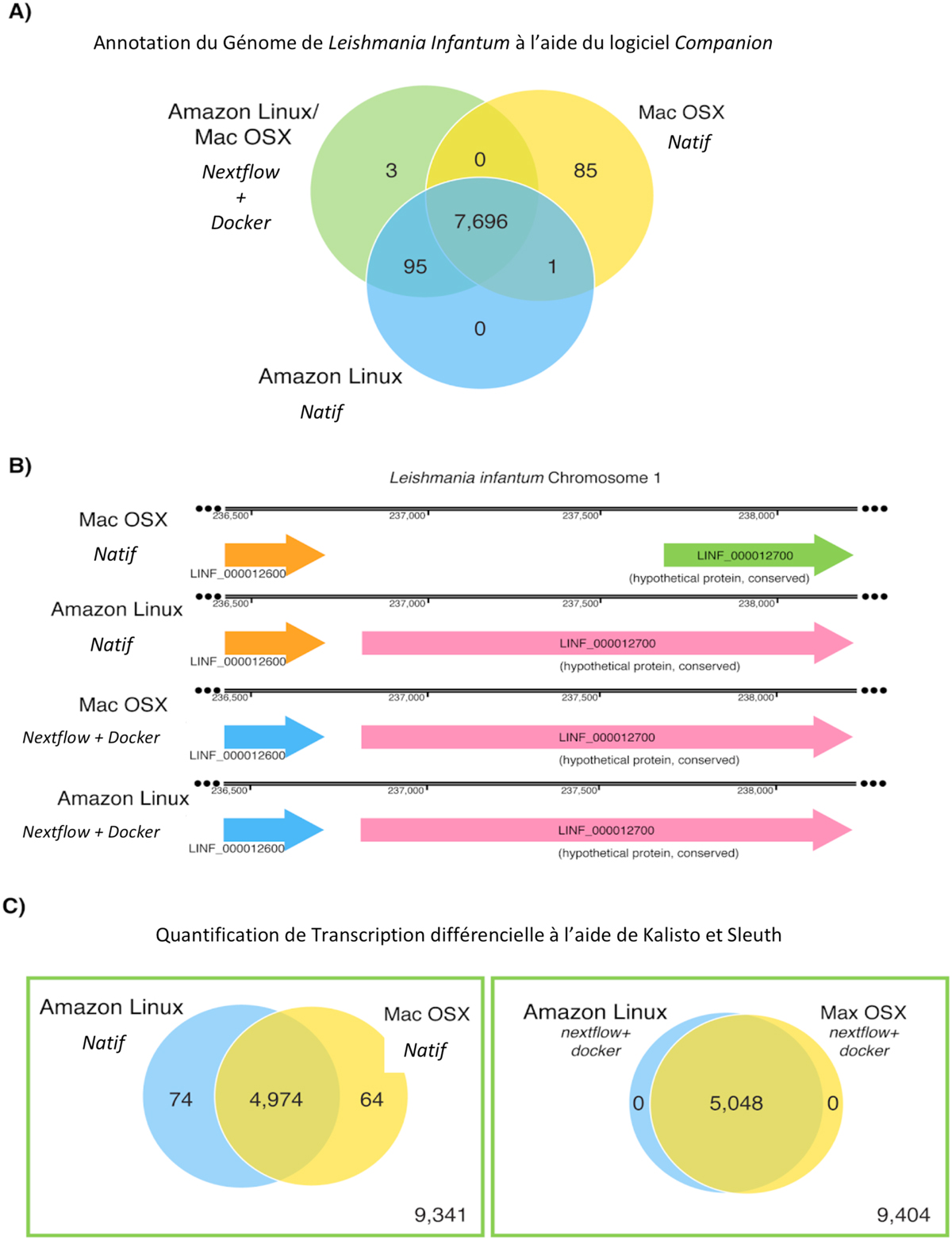
Nextflow produit des résultats stables sur des plates-formes UNIX différentes. (A) L’annotation du génome de Leishmania infantum JPCM5 a été prédite à l’aide d’une version native et dockerisée (Debian Linux) du pipeline d’annotation eucaryote Companion. Les versions natives et dockerisées étaient exécutées sur les plates-formes Mac OSX et Amazon Linux. Le diagramme de Venn montre l’existence de différences minimes mais significatives lors de la comparaison des coordonnées génomiques des gènes codants prédits et des ARN non codants. (B) Certaines de ces disparités incluent des gènes entiers. Au contraire, la version incluse dans un conteneur docker donne le même résultat sur toutes les plates-formes. (C) Une comparaison similaire a été réalisée sur un pipeline Kallisto/Sleuth lors de la recherche de gènes différentiellement exprimés (valeur q < 0,01) dans une expérience ARN-seq (fibroblastes pulmonaires humains). L’analyse réalisée sur des plates-formes Mac OSX et Amazon Linux montre des disparités minimes mais significatives qui disparaissent lorsque la version dockérisée est déployée.
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.


