| Numéro |
Biologie Aujourd’hui
Volume 216, Numéro 1-2, 2022
|
|
|---|---|---|
| Page(s) | 41 - 47 | |
| DOI | https://doi.org/10.1051/jbio/2022008 | |
| Publié en ligne | 25 juillet 2022 | |
Article
L’évolution à petite échelle
Small scale evolution
Sorbonne Université, CNRS, Institut de Biologie Paris-Seine (IBPS), Laboratoire de Biologie du Développement, UMR 7622, 9 quai St-Bernard 75005 Paris, France
* Auteur correspondant: jean-michel.gibert@sorbonne-universite.fr
Reçu :
29
Avril
2022
L’évolution à petite échelle ou microévolution concerne l’évolution au niveau intra-spécifique ou entre espèces proches. Au niveau intra-spécifique, elle permet d’analyser les forces évolutives en action : mutation, dérive génétique, migration et sélection. De plus, en raison de ce temps évolutif court, il est plus facile d’identifier les bases génétiques des différences phénotypiques observées. La plupart des études porte sur des populations actuelles mais de plus en plus de travaux analysent l’ADN ancien. Ces derniers apportent non seulement des informations importantes pour retracer l’histoire des populations mais permettent également de reconstituer les phénotypes d’individus disparus depuis plusieurs milliers d’années. Dans cette courte revue, je présente des travaux montrant comment se mettent en place des barrières pré-zygotiques ou post-zygotiques impliquées dans la formation d’espèces, avec l’exemple de la barrière géographique due à la formation de l’isthme de Panama et celui de la divergence de l’hétérochromatine chez les drosophilidés. Par ailleurs, à propos de cas bien établis, je décris les différentes approches qui ont été utilisées pour identifier les bases génétiques de variations phénotypiques : approche gène-candidat pour ce qui concerne le mélanisme chez les félins, cartographie QTL (Quantitative trait loci) pour la variation du nombre de plaques osseuses latérales chez les épinoches, étude d’association pour la pigmentation chez la coccinelle asiatique. Enfin, j’illustre le rôle de la sélection naturelle avec l’exemple iconique de l’évolution du bec des pinsons des Galapagos et l’implication de certains gènes du développement dans sa diversification morphologique.
Abstract
Small-scale evolution or microevolution concerns evolution at the intra-specific level or between closely related species. At the intra-specific level, it allows the analysis of the evolutionary forces at work: mutation, genetic drift, migration and selection. Moreover, because of the short evolutionary time, it is easier to identify the genetic basis of observed phenotypic differences. Most studies focus on current populations but more and more analyses are performed on ancient DNA. This provides important information for tracing the history of populations and also allows the reconstruction of phenotypes of individuals that disappeared several thousand years ago. In this short review, I present studies showing how pre-zygotic or post-zygotic barriers involved in species formation are set up using the example of the geographical barrier due to the formation of the Isthmus of Panama and that of the heterochromatin divergence in Drosophilidae. I also describe the different approaches that have been used to identify the genetic basis of well known phenotypic variations: candidate gene approach (about melanism in felines), QTL mapping (variation in the number of lateral bone plates in sticklebacks), association study (pigmentation in the Asian ladybird). Finally, I illustrate the key impact of natural selection with the iconic example of the evolution of the beak of Galapagos finches, and the role of certain developmental genes in its morphological diversification.
Mots clés : évolution à petite échelle / microévolution / espèce / génétique / adaptation
Key words: small scale evolution / microevolution / species / genetics / adaptation
© Société de Biologie, 2022
Introduction
Depuis l’apparition de la vie sur terre il y a 3, 5 milliards d’années, l’évolution a produit une remarquable diversité d’organismes. L’évolution à petite échelle ou microévolution s’intéresse à l’évolution intra-spécifique ou entre espèces proches. L’analyse des forces influençant la variation permet d’analyser l’évolution en action : effets des mutations, de la dérive génétique, de la migration et de la sélection naturelle. C’est le domaine de la génétique des populations. Par ailleurs, le temps évolutif court qui caractérise la microévolution permet d’identifier plus facilement les bases génétiques des changements phénotypiques observés car il y a moins de modifications génétiques qu’entre espèces éloignées. Au niveau intra-spécifique, il est possible de réaliser des études d’association ou de faire des croisements pour des analyses par Quantitative trait loci (QTL) (Colosimo et al., 2004, 2005 ; Gautier et al., 2018). Certaines paires d’espèces proches peuvent produire des hybrides fertiles pour au moins l’un des sexes que l’on peut croiser avec l’une des espèces parentales. Ceci est très utile pour identifier les bases génétiques de différences phénotypiques entre ces paires d’espèces (Hagen et al., 2021). Lorsque l’on combine des approches visant à identifier les bases génétiques d’une différence phénotypique et la génétique des populations, on peut reconstituer l’histoire d’un allèle et comment, par exemple, la sélection naturelle a fait augmenter en fréquence un allèle responsable d’une différence phénotypique majeure au sein d’une espèce, qu’il s’agisse d’une mutation survenue dans cette espèce (Van’t Hof et al., 2016) ou d’un allèle introduit à partir d’une autre espèce par introgression après hybridation (Jones et al., 2018). Dans la plupart des cas, ces études sont faites sur des populations actuelles. Cependant, plus récemment, en grande partie grâce aux progrès des méthodes de séquençage (séquençage à haut débit), des études de plus en plus nombreuses ont utilisé l’ADN ancien préservé dansdes restes fossiles pour reconstituer l’histoire de populations récentes, par exemple l’histoire du chat domestique (Ottoni et al., 2017). De plus, l’ADN ancien permet de phénotyper des individus morts il y a des milliers d’années en identifiant des allèles dont on connaît les conséquences phénotypiques, comme ce fut le cas dans l’étude remarquable de Pruvost et al. (2011) sur la pigmentation des chevaux sauvages du paléolithique. Les représentations de chevaux dans certaines grottes peintes du paléolithique montrent des phénotypes de pigmentation rappelant fortement la coloration de certains chevaux domestiques actuels : bai (la forme ancestrale), noirs ou tachetés. Cependant, on ne savait pas si ces représentations correspondaient à la description précise de phénotypes observés dans les populations sauvages de chevaux de l’époque ou s’il s’agissait de représentations symboliques. Les mutations responsables de ces phénotypes chez les chevaux actuels étant connues, elles ont été recherchées et retrouvées dans des ADN anciens préservés dans des ossements de chevaux du paléolithique. Les allèles responsables de certains phénotypes de pigmentation présents chez les chevaux domestiques actuels (bai, mais également noirs ou tachetés) étaient donc déjà présents dans les populations sauvages de chevaux avant la domestication de l’espèce.
Je présente dans cet article des travaux illustrant comment se forment les espèces, quelles sont les bases génétiques des différences intra- ou inter-spécifiques et quel est le rôle de la sélection naturelle.
Comment se forment les espèces?
La définition biologique de l’espèce proposée par Mayr en 1942 est la suivante :
Une espèce est une population ou un ensemble de populations dont les individus peuvent effectivement ou potentiellement se reproduire entre eux et engendrer une descendance viable et féconde, dans des conditions naturelles ». Pour comprendre comment se forment les espèces, il faut donc analyser les mécanismes qui concourent à la mise en place des barrières reproductives.
Il peut s’agir des barrières pré-zygotiques, qui vont par exemple empêcher mâles et femelles de deux futures espèces de se rencontrer et de s’accoupler. Les deux populations divergent ensuite génétiquement et ne sont plus capables de produire des hybrides fertiles. Un type de barrière reproductive bien connu concerne les barrières géographiques. Ainsi, lors de la formation de l’Isthme de Panama il y a quelques millions d’années, des populations d’organismes marins se sont retrouvées isolées dans les océans Pacifique et Atlantique nouvellement séparés. Ces populations, initialement de la même espèce, ont ensuite divergé pour devenir des espèces différentes, comme c’est le cas pour plusieurs espèces de poissons, de mollusques et de crustacés (Miura et al., 2010 ; Thacker, 2017 ; Hiller & Lessios, 2020).
On parle de barrières post-zygotiques lorsque l’accouplement et la fécondation sont possibles mais que les hybrides meurent au cours du développement ou sont stériles. Drosophila melanogaster et Drosophila simulans sont deux espèces proches de drosophiles (insectes diptères) ayant divergé il y a 2 à 3 millions d’années et qui peuvent encore s’accoupler. Les femelles hybrides issues du croisement de mâles D. melanogaster et de femelles D. simulans montrent des mitoses anormales et meurent au cours de l’embryogenèse. Dans ces mitoses anormales, une région chromosomique de certaines chromatides ne ségrège pas correctement au cours de l’anaphase. Cette région correspond au satellite 359, une séquence répétée présente dans une région hétérochromatique du chromosome X paternel (Figure 1). Ce satellite est absent sur le chromosome X de D. simulans. Si on utilise des mères D. simulans et, comme pères, des mâles D. melanogaster issus d’une lignée dans laquelle le satellite 359 est absent du chromosome X, on n’observe plus de mitoses anormales et les femelles hybrides sont viables. Ce résultat souligne l’importance de la divergence de l’hétérochromatine au cours de la formation de nouvelles espèces.
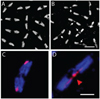 |
Figure 1 La divergence de l’hétérochromatine induit des défauts de ségrégation au cours de la mitose et la létalité des embryons hybrides femelles Drosophila melanogaster / Drosophila simulans (Ferree & Barbash, 2009). Les mitoses sont normales dans un embryon de Drosophila melanogaster (A) tandis que l’on observe des défauts de ségrégation avec des ponts d’ADN (flèches) dans un embryon femelle hybride D. melanogaster / D. simulans (B). Barre d’échelle : 8 µm. L’hybridation avec une sonde marquant le satellite 359-pb (rouge) localisé dans l’héterochromatine du chromosome X de Drosophila melanogaster, mais absent du chromosome X de Drosophila simulans, montre que cette région ségrége normalement au cours des mitoses dans un embryon de Drosophila melanogaster (C) tandis qu’elle est précisément localisée dans les ponts des mitoses anormales chez des embryons hybrides femelles D. melanogaster / D. simulans (D). Barre d’échelle : 5 µm. |
Quelles sont les bases génétiques des différences intra- ou inter-spécifiques ?
La comparaison d’espèces proches montre que certains caractères évoluent particulièrement rapidement. C’est le cas de la taille, en particulier en milieu insulaire où l’on observe que les espèces de grande taille tendent à devenir plus petites, tandis que les espèces de petite taille deviennent souvent plus grandes. Ainsi, il y a quelques milliers d’années existaient des éléphants nains en Sicile et des rats géants aux îles Canaries. L’identification des espèces continentales apparentées a permis de calculer une vitesse accélérée d’évolution de la taille chez ces animaux (Baleka et al., 2021 ; Renom et al., 2021). La coloration est polymorphe chez de nombreuses espèces et évolue également rapidement entre espèces. Un petit nombre de locus, voire un seul, est souvent impliqué dans ces variations de coloration (Theron et al., 2001 ; Eizirik et al., 2003 ; Gautier et al., 2018). Les pièces génitales mâles sont des organes qui évoluent très rapidement, notamment chez les drosophilidés (taille, forme, présence de soies), et des régions génomiques ou même certains gènes impliqués dans ces différences morphologiques générées en quelques centaines de milliers d’années ont été identifiés (Peluffo et al., 2015 ; Tanaka et al., 2015 ; Nagy et al., 2018 ; Hagen et al., 2019, 2021).
Plusieurs approches sont utilisées pour identifier les bases génétiques de différences phénotypiques. Tout d’abord, une approche « gène-candidat », basée sur les résultats obtenus chez des espèces modèles, permet d’analyser de façon ciblée un locus potentiellement impliqué dans le phénotype étudié. Ainsi, il existe chez certaines espèces de félins des formes mélaniques. Des approches gènes-candidats ciblant des gènes ayant un rôle dans la pigmentation, initialement étudiés chez la souris, ont permis de montrer que le mélanisme chez la panthère (Panthera pardus) (Figure 2) est dû à une mutation perte de fonction et récessive dans le gène agouti tandis que chez le jaguar (Panthera onca), le mélanisme est lié à une mutation gain de fonction et dominante au locus MC1R qui rend le récepteur codé par ce gène constitutivement actif (Eizirik et al., 2003 ; Schneider et al., 2012). Le gène agouti code une protéine signal, antagoniste du récepteur MC1R, et ces deux mutations ciblent en réalité la même voie.
Une autre approche utilisée est la cartographie QTL. C’est une approche nécessitant de faire des croisements entre individus montrant des phénotypes différents. C’est donc un type d’étude possible au niveau intra-spécifique ou entre espèce proches, quand les hybrides d’au moins un sexe sont fertiles. Cette approche a été suivie pour identifier les bases génétiques de la différence en nombre de plaques osseuses latérales chez les morphes marins versus les morphes dulcicoles des épinoches (Gasterosteus aculeatus) (Figure 2). Le nombre de plaques latérales est fortement réduit chez les épinoches dulcicoles, et ce changement morphologique a eu lieu de façon récurrente lors de la colonisation par des épinoches marines de lacs d’eau douce formés au moment de la fonte des glaciers il y a 10 000 à 20 000 ans. Une approche par QTL, en croisant des épinoches marines et dulcicoles, a permis d’identifier plusieurs régions génomiques impliquées dans les différences du nombre de plaques osseuses latérales (Colosimo et al., 2004). L’analyse approfondie de la région majoritairement responsable de la variation a permis d’identifier le gène concerné : Ectodysplasin (Eda). La mutation de ce gène associée au nombre réduit de plaques latérales date de plus de 2 millions d’années ; elle était probablement présente à faible fréquence dans les populations marines, et a été recrutée de façon répétée lors de la colonisation du milieu dulcicole (Colosimo et al., 2005). L’allèle dulcicole n’est exprimé qu’à un faible niveau en particulier dans les régions des flancs où se forment des plaques osseuses chez les morphes marins, et où elles sont absentes chez les morphes dulcicoles (O’Brown et al., 2015). Cela a été montré en mesurant l’expression allèle spécifique d’Eda dans les tissus d’un individu F1 issu du croisement d’un morphe marin et d’un morphe dulcicole. La différence d’expression implique des changements dans les séquences cis-régulatrices d’Eda puisque, dans les cellules de l’individu F1, les deux allèles d’Eda sont en présence des mêmes facteurs transrégulateurs. La comparaison de nombreux allèles d’Eda marins et dulcicoles a permis d’identifier un changement de nucléotide unique présent dans tous les allèles dulcicoles mais absent de tous les allèles marins (O’Brown et al., 2015). Celui-ci est localisé dans une séquence régulatrice d’Eda qui dirige l’expression de ce gène dans les flancs. L’analyse d’épinoches transgéniques a montré que le changement de nucléotide observé dans les allèles dulcicoles diminue l’activité de cette séquence régulatrice (O’Brown et al., 2015).
Une dernière approche utilisée est l’étude d’association. Pour cela, il faut disposer d’une population polymorphe, au sein de laquelle ségrége la variation génétique affectant le phénotype donné. En effet, la comparaison de nombreux génomes va permettre d’identifier les régions associées aux différents phénotypes tandis que les autres régions sont homogénéisées entre les différents morphes par la recombinaison. Cette approche a été utilisée avec succès pour identifier les bases génétiques d’un polymorphisme important pour la pigmentation chez la coccinelle asiatique (Harmonia axyridis), une espèce invasive présente en Europe (Gautier et al., 2018). Chez cette espèce, il existe de nombreux morphes (rouges, rouges à taches noires, noires à taches rouges) qui correspondent à une série allélique au même locus (Figure 2). L’étude d’association a identifié une région liée à ce phénotype, contenant deux gènes : GATAe et pannier (pnr). L’inactivation de chacun de ces gènes par ARN interférence a montré que GATAe n’était pas impliqué dans la pigmentation, mais que pnr était requis pour la production de mélanine. De plus, pnr s’exprime dans les élytres au cours du développement au niveau des zones qui seront noires pour tous les morphes analysés. Ce sont donc des changements dans les séquences cis-régulatrices de pnr qui sont impliqués dans ces différents motifs de pigmentation. De façon intéressante, des inversions ont été identifiées dans cette région (Ando et al., 2018 ; Gautier et al., 2018). Elles empêchent la recombinaison et maintiennent une variation génétique importante entre les allèles de pnr des différents morphes. D’autres études chez des oiseaux ou des papillons, par exemple, ont également identifié des inversions, couvrant parfois un grand nombre de gènes, et maintenant une variation génétique importante entre allèles responsables de variation phénotypique (Joron et al., 2011 ; Poelstra et al., 2014 ; Küpper et al., 2016 ; Lamichhaney et al., 2016).
Ces différentes approches ont permis d’identifier un grand nombre de locus impliqués dans des variations phénotypiques au niveau intra- ou inter-spécifique. Un site web remarquable répertorie une grande partie de ces données. Il se nomme Gephebase et peut être consulté à l’adresse suivante : https://www.gephebase.org (Courtier-Orgogozo et al., 2020). L’analyse de toutes ces données permet de caractériser les différents types de mutations impliquées dans les variations phénotypiques étudiées. Il ressort de ces analyses que certains gènes sont des hotspots évolutifs et sont utilisés de façon récurrente au cours de l’évolution (Martin & Orgogozo, 2013). De plus, certains locus à large effet contiennent plusieurs mutations avec des effets faibles. Les variations de morphologie impliquent plus fréquemment des changements dans les séquences régulatrices, tandis que des modifications dans les séquences codantes sont plus souvent impliquées dans des cas d’évolutions physiologiques (Stern & Orgogozo, 2008). Un exemple de mutation dans une séquence régulatrice impliquée dans une différence morphologique concerne le gène Eda chez l’épinoche précédemment présenté (O’Brown et al., 2015). Les variations de ce type de séquences régulatrices n’affectent que certains rôles d’Eda, car ces séquences régulatrices sont modulaires, alors qu’une modification dans la séquence protéique d’Eda aurait des effets pléiotropes. Des exemples de changement de séquences protéiques modulant des processus physiologiques concernent des modifications dans la séquence protéique de l’hémoglobine impliquées dans l’adaptation à l’hypoxie pour des populations ou des espèces supportant l’altitude (Storz et al., 2007 ; Natarajan et al., 2018).
 |
Figure 2 Exemples de phénotypes dont les bases génétiques ont été identifiées par différentes approches. (A) Une approche gène-candidat a permis de montrer que le mélanisme chez la panthère Panthera pardus (photo : J-M Gibert) est dû à une mutation perte de fonction récessive au niveau du locus agouti (Schneider et al., 2012). (B) Les morphes marins et dulcicoles de l’épinoche Gasterosteus aculeatus diffèrent notamment par le nombre de plaques osseuses latérales, ici colorées au rouge Alizarine (O’Brown et al., 2015). Une approche de cartographie QTL a identifié plusieurs régions génomiques impliquées dans cette différence dont celle portant le gène Ectodysplasin. (C) La coccinelle asiatique Harmonia axyridis (photo : J-M Gibert) montre un polymorphisme remarquable pour la pigmentation des élytres. Une étude d’association a montré qu’elle était due à une série allélique au locus pannier, dont l’expression spécifie les zones noires (Gautier et al., 2018). |
Quel est le rôle de la sélection naturelle ?
Darwin a découvert la sélection naturelle en analysant les pinsons des îles Galapagos. Il s’agit d’un complexe d’espèces étroitement apparentées, diversifiées au cours des 2 à 3 derniers millions d’années sur cet archipel et adaptées à différents régimes alimentaires (graines de tailles variées, sang d’oiseaux de mer, nectar de fleurs, insectes) via des changements de taille et de morphologie du bec (Al-Mosleh et al., 2021). Ces populations d’oiseaux ont été l’objet de nombreuses études en vue de comprendre leurs réponses adaptatives aux évènements climatiques. Rosemary et Peter Grant ont ainsi montré qu’après la sécheresse de 1977 la taille des graines disponibles a augmenté et que, pour l’espèce Geospiza fortis, seuls les individus au bec le plus gros ont survécu et se sont reproduits (Grant & Grant, 2003). En conséquence, à la génération suivante, la taille moyenne du bec dans la population a nettement augmenté, car les petits ont hérité de leurs parents des allèles responsables de ce trait morphologique.
D’autres études ont cherché à comprendre comment la taille et la forme du bec pouvaient évoluer et générer cette diversité remarquable observée entre espèces de pinsons. L’étude du morphogène BMP4 a montré qu’il était plus fortement exprimé au cours du développement du bec chez les espèces au bec le plus grand, et la manipulation de ce morphogène dans le bec d’embryons de poulets a indiqué un rôle causal sur la taille du bec (Abzhanov et al., 2004). Une étude récente, basée sur le séquençage des génomes de toutes les espèces de pinsons des Galapagos, a montré que l’hybridation était fréquente entre espèces et pouvait contribuer à l’adaptation via l’introgression de certains locus (Lamichhaney et al., 2015). Une quinzaine de régions du génome associées à des différences de taille et de forme du bec ont été identifiées. Le locus BMP4 n’a pas été retrouvé dans cette analyse, ce qui laisse à penser que la différence d’expression précédemment montrée pourrait dépendre de changements affectant des facteurs trans-régulateurs. Enfin, il a pu être établi qu’un nouveau facteur de transcription impliqué dans le développement crânio-facial, ALX1, est associé à des différences de morphologies du bec entre espèces et à l’intérieur de l’espèce Geospiza fortis.
Conclusion
Ces différentes études sur l’évolution à petite échelle permettent de comprendre comment les espèces s’établissent, pourquoi elles se modifient et comment elles le font. Le grand nombre d’espèces analysées et l’accès à leur génome permettent désormais d’identifier de façon non biaisée les types de locus impliqués (contrairement à l’approche gène-candidat). Il ressort, entre autres, que l’hybridation et l’introgression adaptative sont beaucoup plus fréquentes qu’on ne le pensait précédemment. Des inversions maintiennent parfois une variation génétique importante entre allèles différents. Enfin, les changements de morphologie impliquent plus souvent des modifications dans les séquences cis-régulatrices tandis que des variations physiologiques sont plus souvent associées à des modifications des séquences protéiques.
Références
- Abzhanov, A., Protas, M., Grant, B.R., Grant, P.R., Tabin, C.J. (2004). Bmp4 and morphological variation of beaks in Darwin’s finches. Science, 305, 1462-1465. [CrossRef] [PubMed] [Google Scholar]
- Al-Mosleh, S., Choi, G.P.T., Abzhanov, A., Mahadevan, L. (2021). Geometry and dynamics link form, function, and evolution of finch beaks. Proc Natl Acad Sci USA, 118, e2105957118. [CrossRef] [PubMed] [Google Scholar]
- Ando, T., Matsuda, T., Goto, K., Hara, K., Ito, A., Hirata, J., Yatomi, J., Kajitani, R., Okuno, M., Yamaguchi, K., Kobayashi, M., Takano, T., Minakuchi, Y., Seki, M., Suzuki, Y., Yano, K., Itoh, T., Shigenobu, S., Toyoda, A., Niimi, T. (2018). Repeated inversions within a pannier intron drive diversification of intraspecific colour patterns of ladybird beetles. Nat Commun, 9, 3843. [CrossRef] [PubMed] [Google Scholar]
- Baleka, S., Herridge, V.L., Catalano, G., Lister, A.M., Dickinson, M.R., Di Patti, C., Barlow, A., Penkman, K.E.H., Hofreiter, M., Paijmans, J.L.A. (2021). Estimating the dwarfing rate of an extinct Sicilian elephant. Curr Biol, 31, 3606-3612.e7. [CrossRef] [PubMed] [Google Scholar]
- Colosimo, P.F., Peichel, C.L., Nereng, K., Blackman, B.K., Shapiro, M.D., Schluter, D., Kingsley, D.M. (2004). The genetic architecture of parallel armor plate reduction in threespine sticklebacks. PLoS Biol, 2, E109. [Google Scholar]
- Colosimo, P.F., Hosemann, K.E., Balabhadra, S., Villarreal, G., Jr., Dickson, M., Grimwood, J., Schmutz, J., Myers, R.M., Schluter, D., Kingsley, D.M. (2005). Widespread parallel evolution in sticklebacks by repeated fixation of Ectodysplasin alleles. Science, 307, 1928-1933. [CrossRef] [PubMed] [Google Scholar]
- Courtier-Orgogozo, V., Arnoult, L., Prigent, S.R., Wiltgen, S., Martin, A. (2020). Gephebase, a database of genotype-phenotype relationships for natural and domesticated variation in Eukaryotes. Nucl Acids Res, 48, D696-D703. [CrossRef] [PubMed] [Google Scholar]
- Eizirik, E., Yuhki, N., Johnson, W.E., Menotti-Raymond, M., Hannah, S.S., O’Brien, S.J. (2003). Molecular genetics and evolution of melanism in the cat family. Curr Biol, 13, 448-453. [CrossRef] [PubMed] [Google Scholar]
- Ferree, P.M., Barbash, D.A. (2009). Species-specific heterochromatin prevents mitotic chromosome segregation to cause hybrid lethality in Drosophila. PLoS Biol, 7, e1000234. [CrossRef] [Google Scholar]
- Gautier, M., Yamaguchi, J., Foucaud, J., Loiseau, A., Ausset, A., Facon, B., Gschloessl, B., Lagnel, J., Loire, E., Parrinello, H., Severac, D., Lopez-Roques, C., Donnadieu, C., Manno, M., Berges, H., Gharbi, K., Lawson-Handley, L., Zang, L.S., Vogel, H., Estoup, A., Prudhomme, B. (2018). The genomic basis of color pattern polymorphism in the Harlequin ladybird. Curr Biol, 28, 3296-3302.e7. [CrossRef] [PubMed] [Google Scholar]
- Grant, R.B., Grant, P.R. (2003). What Darwin’s finches can teach us about the evolutionary origin and regulation of biodiversity. BioScience, 53, 965-975. [CrossRef] [Google Scholar]
- Hagen, J.F.D., Mendes, C.C., Blogg, A., Payne, A., Tanaka, K.M., Gaspar, P., Figueras Jimenez, J., Kittelmann, M., McGregor, A.P., Nunes, M.D.S. (2019). Tartan underlies the evolution of Drosophila male genital morphology. Proc Natl Acad Sci USA, 116, 19025-19030. [CrossRef] [PubMed] [Google Scholar]
- Hagen, J.F.D., Mendes, C.C., Booth, S.R., Figueras Jimenez, J., Tanaka, K.M., Franke, F.A., Baudouin-Gonzalez, L., Ridgway, A.M., Arif, S., Nunes, M.D.S., McGregor, P.A. (2021). Unraveling the genetic basis for the rapid diversification of male genitalia between Drosophila species. Mol Biol Evol, 38, 437-448. [CrossRef] [PubMed] [Google Scholar]
- Hiller, A., Lessios, H.A. (2020). Marine species formation along the rise of Central America: The anomuran crab Megalobrachium. Mol Ecol, 29, 413-428. [CrossRef] [PubMed] [Google Scholar]
- Jones, M.R., Mills, L.S., Alves, P.C., Callahan, C.M., Alves, J.M., Lafferty, D.J.R., Jiggins, F.M., Jensen, J.D., Melo-Ferreira, J., Good, J.M. (2018). Adaptive introgression underlies polymorphic seasonal camouflage in snowshoe hares. Science, 360, 1355-1358. [CrossRef] [PubMed] [Google Scholar]
- Joron, M., Frezal, L., Jones, R.T., Chamberlain, N.L., Lee, S.F., Haag, C.R., Whibley, A., Becuwe, M., Baxter, S.W., Ferguson, L., Wilkinson, P.A., Salazar, C., Davidson, C., Clark, R., Quail, M.A., Beasley, H., Glithero, R., Lloyd, C., Sims, S., Jones, M.C., Rogers, J., Jiggins, C.D., ffrench-Constant, R.H. (2011). Chromosomal rearrangements maintain a polymorphic supergene controlling butterfly mimicry. Nature, 477, 203-206. [CrossRef] [PubMed] [Google Scholar]
- Küpper, C., Stocks, M., Risse, J.E., Dos Remedios, N., Farrell, L.L., McRae, S.B., Morgan, T.C., Karlionova, N., Pinchuk, P., Verkuil, Y.I., Kitaysky, A.S., Wingfield, J.C., Piersma, T., Zeng, K., Slate, J., Blaxter, M., Lank, D.B., Burke, T. (2016). A supergene determines highly divergent male reproductive morphs in the ruff. Nat Genet, 48, 79-83. [CrossRef] [PubMed] [Google Scholar]
- Lamichhaney, S., Berglund, J., Almén, M.S., Maqbool, K., Grabherr, M., Martinez Barrio, A., Promerová, M., Rubin, C.-J., Wang, C., Zamani, N., Grant, B.R., Grant, P.R., Webster, M.T., Andersson, L. (2015). Evolution of Darwin’s finches and their beaks revealed by genome sequencing. Nature, 518, 371-375. [CrossRef] [PubMed] [Google Scholar]
- Lamichhaney, S., Fan, G., Widemo, F., Gunnarsson, U., Thalmann, D.S., Hoeppner, M.P., Kerje, S., Gustafson, U., Shi, C., Zhang, H., Chen, W., Liang, X., Huang, L., Wang, J., Liang, E., Wu, Q., Lee, S.M., Xu, X., Höglund, J., Liu, X., Andersson, L. (2016). Structural genomic changes underlie alternative reproductive strategies in the ruff (Philomachus pugnax). Nat Genet, 48, 84-88. [CrossRef] [PubMed] [Google Scholar]
- Martin, A., Orgogozo, V. (2013). The Loci of repeated evolution: a catalog of genetic hotspots of phenotypic variation. Evolution, 67, 1235-1250. [Google Scholar]
- Mayr, E., Systematics and the origin of species from the viewpoint of a zoologist, Columbia Univ Press, New York, 1942 [Google Scholar]
- Miura, O., Torchin, M.E., Bermingham, E. (2010). Molecular phylogenetics reveals differential divergence of coastal snails separated by the Isthmus of Panama. Mol Phylogenet Evol, 56, 40-48. [CrossRef] [PubMed] [Google Scholar]
- Nagy, O., Nuez, I., Savisaar, R., Peluffo, A.E., Yassin, A., Lang, M., Stern, D.L., Matute, D.R., David, J.R., Courtier-Orgogozo, V. (2018). Correlated evolution of two copulatory organs via a single cis-regulatory nucleotide change. Curr Biol, 28, 3450-3457.e13. [CrossRef] [PubMed] [Google Scholar]
- Natarajan, C., Jendroszek, A., Kumar, A., Weber, R.E., Tame, J.R.H., Fago, A., Storz, J.F. (2018). Molecular basis of hemoglobin adaptation in the high-flying bar-headed goose. PLoS Genet, 14, e1007331. [CrossRef] [PubMed] [Google Scholar]
- O’Brown, N.M., Summers, B.R., Jones, F.C., Brady, S.D., Kingsley, D.M. (2015). A recurrent regulatory change underlying altered expression and Wnt response of the stickleback armor plates gene EDA. Elife, 4, e05290. [CrossRef] [PubMed] [Google Scholar]
- Ottoni, C., Van Neer, W., De Cupere, B., Daligault, J., Guimaraes, S., Peters, J., Spassov, N., Prendergast, M.E., Boivin, N., Morales-Muñiz, A., Bălăşescu, A., Becker, C., Benecke, N., Boroneant, A., Buitenhuis, H., Chahoud, J., Crowther, A., Llorente, L., Manaseryan, N., Monchot, H., Onar, V., Osypińska, M., Putelat, O., Quintana Morales, E.M., Studer, J., Wierer, U., Decorte, R., Grange, T., Geigl, E.-M. (2017). The paleogenetics of cat dispersal in the ancient world. Nat Ecol Evol, 1, 1-7. [CrossRef] [PubMed] [Google Scholar]
- Peluffo, A.E., Nuez, I., Debat, V., Savisaar, R., Stern, D.L., Orgogozo, V. (2015). A major locus controls a genital shape difference involved in reproductive isolation between Drosophila yakuba and Drosophila santomea. G3 (Bethesda), 5, 2893-2901. [Google Scholar]
- Poelstra, J.W., Vijay, N., Bossu, C.M., Lantz, H., Ryll, B., Müller, I., Baglione, V., Unneberg, P., Wikelski, M., Grabherr, M.G., Wolf, J.B. (2014). The genomic landscape underlying phenotypic integrity in the face of gene flow in crows. Science, 344, 1410-1414. [CrossRef] [PubMed] [Google Scholar]
- Pruvost, M., Bellone, R., Benecke, N., Sandoval-Castellanos, E., Cieslak, M., Kuznetsova, T., Morales-Muñiz, A., O’Connor, T., Reissmann, M., Hofreiter, M., Ludwig, A. (2011). Genotypes of predomestic horses match phenotypes painted in Paleolithic works of cave art. Proc Natl Acad Sci USA, 108, 18626-18630. [CrossRef] [PubMed] [Google Scholar]
- Renom, P., de-Dios, T., Civit, S., Llovera, L., Sánchez-Gracia, A., Lizano, E., Rando, J.C., Marquès-Bonet, T., Kergoat, G.J., Casanovas-Vilar, I., Lalueza-Fox, C. (2021). Genetic data from the extinct giant rat from Tenerife (Canary Islands) points to a recent divergence from mainland relatives. Biol Lett, 17, 20210533. [CrossRef] [PubMed] [Google Scholar]
- Schneider, A., David, V.A., Johnson, W.E., O’Brien, S.J., Barsh, G.S., Menotti-Raymond, M., Eizirik, E. (2012). How the leopard hides its spots: ASIP mutations and melanism in wild cats. PLoS ONE, 7, e50386. [CrossRef] [PubMed] [Google Scholar]
- Stern, D.L., Orgogozo, V. (2008). The loci of evolution: how predictable is genetic evolution? Evolution, 62, 2155-2177. [Google Scholar]
- Storz, J.F., Sabatino, S.J., Hoffmann, F.G., Gering, E.J., Moriyama, H., Ferrand, N., Monteiro, B., Nachman, M.W. (2007). The molecular basis of high-altitude adaptation in deer mice. PLoS Genetics, 3, e45. [CrossRef] [PubMed] [Google Scholar]
- Tanaka, K.M., Hopfen, C., Herbert, M.R., Schlötterer, C., Stern, D.L., Masly, J.P., McGregor, A.P., Nunes, M.D.S. (2015). Genetic architecture and functional characterization of genes underlying the rapid diversification of male external genitalia between Drosophila simulans and Drosophila mauritiana. Genetics, 200, 357-369. [CrossRef] [PubMed] [Google Scholar]
- Thacker, C.E. (2017). Patterns of divergence in fish species separated by the Isthmus of Panama. BMC Evol Biol, 17, 111. [CrossRef] [PubMed] [Google Scholar]
- Theron, E., Hawkins, K., Bermingham, E., Ricklefs, R.E., Mundy, N.I. (2001). The molecular basis of an avian plumage polymorphism in the wild: a melanocortin-1-receptor point mutation is perfectly associated with the melanic plumage morph of the bananaquit, Coereba flaveola. Curr Biol, 11, 550-557. [CrossRef] [Google Scholar]
- Van’t Hof, A.E., Campagne, P., Rigden, D.J., Yung, C.J., Lingley, J., Quail, M.A., Hall, N., Darby, A.C., Saccheri, I.J. (2016). The industrial melanism mutation in British peppered moths is a transposable element. Nature, 534, 102-105. [CrossRef] [PubMed] [Google Scholar]
Citation de l’article : Gibert, J.-M. (2022). L’évolution à petite échelle. Biologie Aujourd’hui, 216, 41-47
Liste des figures
 |
Figure 1 La divergence de l’hétérochromatine induit des défauts de ségrégation au cours de la mitose et la létalité des embryons hybrides femelles Drosophila melanogaster / Drosophila simulans (Ferree & Barbash, 2009). Les mitoses sont normales dans un embryon de Drosophila melanogaster (A) tandis que l’on observe des défauts de ségrégation avec des ponts d’ADN (flèches) dans un embryon femelle hybride D. melanogaster / D. simulans (B). Barre d’échelle : 8 µm. L’hybridation avec une sonde marquant le satellite 359-pb (rouge) localisé dans l’héterochromatine du chromosome X de Drosophila melanogaster, mais absent du chromosome X de Drosophila simulans, montre que cette région ségrége normalement au cours des mitoses dans un embryon de Drosophila melanogaster (C) tandis qu’elle est précisément localisée dans les ponts des mitoses anormales chez des embryons hybrides femelles D. melanogaster / D. simulans (D). Barre d’échelle : 5 µm. |
| Dans le texte | |
 |
Figure 2 Exemples de phénotypes dont les bases génétiques ont été identifiées par différentes approches. (A) Une approche gène-candidat a permis de montrer que le mélanisme chez la panthère Panthera pardus (photo : J-M Gibert) est dû à une mutation perte de fonction récessive au niveau du locus agouti (Schneider et al., 2012). (B) Les morphes marins et dulcicoles de l’épinoche Gasterosteus aculeatus diffèrent notamment par le nombre de plaques osseuses latérales, ici colorées au rouge Alizarine (O’Brown et al., 2015). Une approche de cartographie QTL a identifié plusieurs régions génomiques impliquées dans cette différence dont celle portant le gène Ectodysplasin. (C) La coccinelle asiatique Harmonia axyridis (photo : J-M Gibert) montre un polymorphisme remarquable pour la pigmentation des élytres. Une étude d’association a montré qu’elle était due à une série allélique au locus pannier, dont l’expression spécifie les zones noires (Gautier et al., 2018). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.


