| Numéro |
Biologie Aujourd'hui
Volume 212, Numéro 3-4, 2018
|
|
|---|---|---|
| Page(s) | 55 - 59 | |
| DOI | https://doi.org/10.1051/jbio/2018025 | |
| Publié en ligne | 11 avril 2019 | |
Article
Protéomique circadienne
Circadian proteomics
1
Institut de Bioingénierie, Faculté des Sciences de la Vie, École Polytechnique Fédérale de Lausanne,
1015
Lausanne, Suisse
2
Département de Pharmacologie et Toxicologie, Université de Lausanne,
1005
Lausanne, Suisse
* Auteur correspondant: frederic.gachon@epfl.ch
Reçu :
2
Octobre
2018
Les progrès récents des techniques de protéomique offrent de nouvelles perspectives pour la biologie circadienne, et en particulier la possibilité d’étudier des modifications post-traductionnelles telles que la phosphorylation et l’acétylation. En utilisant la protéomique in vivo sur des extraits totaux de foie de souris ou des extraits nucléaires, nous avons pu caractériser le protéome rythmique du foie avec une résolution sans précédent, et ainsi révéler de nouveaux processus rythmiques tels que la sécrétion des protéines, la synthèse des ribosomes, la réparation de l’ADN ou la polyploïdie. De plus, l’analyse des modifications post-traductionnelles a permis de mettre en évidence les voies de signalisation impliquées et les conséquences sur le métabolisme hépatique.
Abstract
Recent development in the proteomic technologies offers new perspectives in circadian biology and in particular the possibility to study post-translational modifications such as phosphorylation and acetylation. Applying in vivo proteomics on whole liver or on nuclear extracts, we were able to characterize the rhythmic liver proteome with unprecedented coverage. It allows the characterization of new rhythmic processes such as protein secretion, ribosome biogenesis, DNA repair, and polyploidy. In addition, the analysis of rhythmic post-translational modifications helps to understand the signal pathways involved and their consequences on hepatic metabolism.
Mots clés : horloge circadienne / physiologie hépatique / protéomique / phosphorylation / acétylation
Key words: circadian clock / liver physiology / proteomic / phosphorylation / acetylation
© Société de Biologie, 2019
L’horloge circadienne et sa fonction
La plupart des organismes vivants sur la terre ont acquis durant l’évolution une horloge circadienne, du latin circa diem (environ un jour), pour leur permettre d’anticiper les changements environnementaux dus à l’alternance des périodes de jour et de nuit causée par la rotation de la terre. Chez les mammifères, cette horloge synchronise la plupart des aspects de la physiologie et du comportement des animaux. Elle est organisée de manière hiérarchique, avec une horloge centrale localisée dans les noyaux suprachiasmatiques (NSC) de l’hypothalamus. Ces NSC synchronisent les oscillateurs périphériques présents dans la quasi totalité des cellules de l’organisme via la sécrétion de neuropeptides ou grâce à des connexions nerveuses directes (Panda, 2016 ; Hastings et al., 2018). Au niveau moléculaire, les rythmes circadiens d’expression des gènes sont générés par des boucles interconnectées de rétrocontrôle de la transcription et de la traduction, impliquant des régulations temporelles au niveau transcriptionnel, post-transcriptionnel et post-traductionnel (Takahashi, 2016).
La particularité de cette horloge circadienne est d’être capable de générer des rythmes d’environ 24 h avec une robustesse remarquable, mais d’être également synchronisable par des facteurs environnementaux. Ainsi, la lumière ou les rythmes alimentaires peuvent synchroniser respectivement l’horloge centrale des NSC ou les horloges périphériques pour s’adapter à l’environnement (Atger et al., 2017). Dans ce contexte, nourrir les animaux durant leur phase de repos (c’est-à-dire le jour pour la souris, animal nocturne) inverse la phase de l’expression des gènes dans leurs tissus périphériques (Damiola et al., 2000).
Régulation du protéome circadien
Si le rôle de l’horloge circadienne et des rythmes alimentaires dans la régulation temporelle de l’expression des produits des gènes a été intensivement étudié au niveau transcriptionnel, très peu d’information existe quant à leur impact sur la régulation rythmique de la traduction et de l’accumulation des protéines. Dans ce contexte, nous avons pu mettre récemment en évidence une régulation traductionnelle des protéines, principalement réglée par les rythmes alimentaires (Jouffe et al., 2013 ; Atger et al., 2015). Cette traduction temporelle concerne essentiellement les protéines ribosomales avec pour conséquence une synthèse rythmique des ribosomes se manifestant par une fluctuation de la taille du foie chez la souris lorsque les rythmes alimentaires et circadiens sont synchronisés (Jouffe et al., 2013 ; Sinturel et al., 2017).
Cependant, les conséquences de ces transcriptions et traductions rythmiques sur la dynamique du protéome du foie sont encore largement inconnues. Des études pionnières utilisant la technologie de l’électrophorèse 2D avaient permis de quantifier quelques dizaines de protéines rythmiques au niveau hépatique, dont une grande partie est codée par des ARN messagers (ARNm) non rythmiques, soulignant l’implication de mécanismes post-transcriptionnels dans leur régulation (Reddy et al., 2006).
La protéomique circadienne à grande échelle
Profitant des progrès technologiques de l’analyse du protéome par spectrométrie de masse quantitative à haute résolution, incluant un marquage in vivo par des isotopes non radioactifs (SILAC), nous avons pu augmenter fortement le niveau de résolution de l’étude du protéome rythmique et découvrir de nouvelles fonctions biologiques régulées par l’horloge biologique ou les rythmes alimentaires chez la souris. Pas moins de 5000 protéines du foie ont pu être ainsi quantifiées et parmi celles-ci 6 % présentaient un profil d’accumulation rythmique. En comparant les rythmes des ARNm correspondant à ce protéome rythmique, nous avons pu montrer que seule la moitié de celui-ci était effectivement codée par des ARNm rythmiques, confirmant la forte implication des mécanismes post-transcriptionnels. L’analyse des fonctions exercées par ce premier groupe de protéines a révélé une intervention dans les fonctions du foie telles que le métabolisme lipidique (phase diurne) mais aussi celui du glucose et des xénobiotiques (phase nocturne). L’autre moitié du protéome rythmique, codée par des ARNm non rythmiques, est fortement enrichie en protéines sécrétées dans la circulation. Ces protéines sécrétées par le foie incluent notamment l’albumine, les facteurs de coagulation du sang, les protéines du complément impliquées dans la réponse immunitaire et une famille d’inhibiteurs de protéases appelées serpines. Toutes ces protéines présentent un rythme d’accumulation synchrone dans le plasma, rythme qui est conservé chez l’homme. Le rythme de sécrétion de ces protéines persiste chez les souris déficientes pour l’horloge circadienne nourries uniquement en phase nocturne, et disparaît pendant le jeûne. Ceci suggère que des signaux liés à l’alimentation influencent le rythme de sécrétion de ces protéines et, plus généralement, des facteurs circulant dans le plasma (Mauvoisin et al., 2014).
Développement de la protéomique circadienne au niveau subcellulaire
L’analyse d’extraits protéiques complexes provenant de cellules ou de tissus entiers reste difficile malgré les progrès technologiques. Il est ainsi compliqué de quantifier des protéines faiblement exprimées comme les facteurs de transcription. Pour contourner cette limite technique, nous avons effectué une analyse de protéomique et de phosphoprotéomique quantitative sur des noyaux de cellules du foie purifiés. Nous avons combiné l’approche in vivo SILAC précédemment utilisée et l’enrichissement de noyaux de manière à quantifier plus de 4000 protéines nucléaires. Le taux de détection des protéines nucléaires dans le foie est sans précédent puisque 70 % des protéines prédites pour avoir une localisation nucléaire sont représentées dans nos données, incluant 90 % des composants des compartiments nucléaires expérimentalement caractérisés.
Une analyse de rythmicité très sélective de ce protéome nucléaire révèle qu’environ 10 % de ces protéines présentent une accumulation rythmique. Cependant, ce taux de protéines rythmiques atteint 45 % dès lors que nous utilisons les mêmes critères de sélection que ceux de l’étude précédente (Mauvoisin et al., 2014), suggérant une régulation diurne extrêmement importante au niveau nucléaire. Ces protéines rythmiques présentent des maxima d’accumulation en fin de journée et de nuit. En comparant les profils d’accumulation des ARNm correspondant à ces protéines, on s’aperçoit que seulement le quart de celles-ci sont codées par des ARNm rythmiques, suggérant une importante régulation au niveau post-transcriptionnel, voire post-traductionnel (Wang et al., 2017).
Les protéines s’accumulent dans le noyau sous forme de complexes impliqués dans la régulation temporelle de processus cellulaires fondamentaux
La plupart des protéines nucléaires détectées appartiennent à des complexes protéiques précédemment caractérisés. Nous observons que beaucoup de sous-unités de complexes connus, rythmiques ou non, présentent des accumulations synchrones dans le noyau. Ceci indique que la régulation diurne du transport nucléaire de ces complexes est certainement un élément important de la régulation du protéome nucléaire rythmique. L’analyse de la rythmicité de ces complexes montre que ceux présents dans le noyau le jour sont plutôt impliqués dans la réparation de l’ADN et la régulation transcriptionnelle, tandis que les complexes présents la nuit interviennent dans la régulation de l’organisation du cytosquelette, le transport protéique et la protéolyse. Représentant environ 20 % du protéome nucléaire rythmique, la majorité des protéines engagées dans les différentes étapes de la biogénèse des ribosomes s’accumulent de façon séquentielle, ce qui est en accord avec les travaux précédents portant notamment sur la synthèse rythmique des protéines et des ARN ribosomaux (ARNr) (Jouffe et al., 2013 ; Atger et al., 2015 ; Sinturel et al., 2017). Ces résultats constituent un exemple supplémentaire du rôle fondamental de l’horloge biologique dans la régulation temporelle de processus biologiques énergivores tels que la synthèse des ribosomes (Wang et al., 2017).
Phosphoprotéome rythmique et activités kinases associées
Pour obtenir plus d’informations sur la dynamique du protéome nucléaire rythmique, nous avons analysé en parallèle sa régulation au niveau post-traductionnel. Dans ce but, nous avons quantifié le phosphoprotéome rythmique et analysé les activités kinases associées. Nous avons quantifié près de 1500 sites phosphorylés dont 10 % présentaient un profil rythmique. Deux tiers de ces sites étaient situés sur des protéines nucléaires non rythmiques et montraient une répartition d’accumulation en fin de journée et de nuit. Ces données nous ont permis de déduire la rythmicité de 39 motifs consensus de kinases dont une grande partie est liée à des kinases impliquées dans la régulation du cycle cellulaire (Wang et al., 2017).
Orchestration rythmique de la transcription au niveau nucléaire
Parmi les protéines et les motifs phosphorylés rythmiques au niveau nucléaire, nous avons identifié de nombreux facteurs de transcription. Nos données montrent, en effet, que 99 facteurs de transcription et 80 co-régulateurs de transcription sont rythmiques au niveau nucléaire. Sur ces facteurs, 16 facteurs et 17 co-régulateurs de transcription conservent leur rythme chez des souris dépourvues d’horloge circadienne, indiquant, comme c’est le cas pour la protéine SREBP1 (Sterol Regulatory Element Binding Protein 1), impliquée dans la régulation du métabolisme des lipides, qu’ils sont principalement régulés par des signaux d’entraînement indépendants de l’horloge moléculaire tels que les rythmes alimentaires (Wang et al., 2017).
La polyploïdie hépatique orchestrée de manière rythmique
Les différentes analyses réalisées ont permis de mettre en évidence des événements relatifs au cycle cellulaire tels que l’activité de nombreuses kinases et de facteurs de transcription. Ceci est en accord avec des travaux précédents portant sur le lien entre le cycle cellulaire et l’horloge circadienne (Matsuo et al., 2003). Ainsi, il semblerait que les cycles cellulaires et circadiens soient alignés bien que le foie soit un organe principalement quiescent. Le foie étant un tissu sujet à la polyploïdie (Gentric & Desdouets, 2014), nous nous sommes demandé si un lien entre les activités liées au cycle cellulaire et la polyploïdie pouvait éventuellement exister. En analysant la quantité d’ADN présente dans les noyaux, nous avons pu observer une proportion antiphasique des noyaux diploïdes comparés aux noyaux tétraploïdes. Indépendamment, nous avons aussi observé par analyse de coupes histologiques la proportion des différentes populations hépatocytaires. La proportion des hépatocytes binucléés diploïdes (2 × 2N) est antiphasique par rapport à celle des hépatocytes mononucléés tétraploïdes (1 × 4N), si bien que cette polyploïdie rythmique pourrait être la conséquence d’un lien entre les cycles cellulaire et diurne (Wang et al., 2017). Ces différents phénomènes sont résumés dans la figure 1.
 |
Figure 1 Orchestration de la physiologie rythmique du foie mise en évidence par protéomique nucléaire quantitative. La combinaison de l’analyse quantitative et temporelle du protéome et du phosphoprotéome nucléaire in vivo nous a permis de mettre en évidence la rythmicité de nombreux processus cellulaires fondamentaux tels que la régulation transcriptionnelle, l’assemblage des ribosomes et la réparation de l’ADN. Les cycles cellulaire et diurne semblent alignés et la polyploïdie rythmique que nous observons semblerait être une conséquence de ce lien. © Med Sci (Paris) 2017, 33, 573-576. |
Orchestration de l’acétylation rythmique des protéines par l’horloge circadienne et les rythmes alimentaires
De manière à obtenir des informations complémentaires sur l’activité de ces protéines, nous nous sommes intéressés à leurs modifications post-traductionnelles. En plus de la phosphorylation décrite précédemment, nous avons caractérisé l’acétylation rythmique des protéines dans des extraits totaux et nucléaires. De manière surprenante, nous avons pu mettre en évidence que les protéines de la mitochondrie étaient essentiellement acétylées pendant le jour, soit la période de jeûne chez la souris, alors que les protéines nucléaires étaient acétylées durant la nuit, quand les animaux se nourrissent (Mauvoisin et al., 2017). Ces fluctuations de l’acétylation en fonction du compartiment cellulaire correspondent à celles de la synthèse dans ces compartiments de l’acétyl-Coenzyme A (acétyl-CoA), substrat nécessaire à l’acétylation des protéines. Cette synthèse rythmique de l’acétyl-CoA dépend essentiellement des rythmes alimentaires (Shi & Tu, 2015). Cependant, cette acétylation rythmique résulte également de la déacétylation des protéines par les déacétylases de la famille des sirtuines, et notamment SIRT3. L’activité de ces enzymes dépendantes du Nicotinamide Adénine Dinucléotide (NAD+) est également rythmique et contrôlée par l’horloge circadienne (Peek et al., 2013). De ce fait, les animaux déficients pour l’horloge circadienne présentent un niveau anormal de NAD+, et donc d’acétylation des protéines. Il en résulte une dérégulation de l’activité rythmique de ces protéines acétylées, impactant le métabolisme de l’urée et des acides aminées (Mauvoisin et al., 2017) (Figure 2).
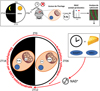 |
Figure 2 L’acétylation rythmique des protéines présente une phase différente selon la localisation subcellulaire des protéines. Les rythmes alimentaires et l’horloge circadienne contrôlent cette acétylation, respectivement en régulant la synthèse d’acétly-CoA et du NAD+ dont dépend l’activité de la déacétylase SIRT3. © Cell Rep 2017, 20, 1729-1743. |
Conclusion
La combinaison de l’analyse quantitative et temporelle du protéome, du phosphoprotéome, et de l’acétylome nucléaire in vivo constitue un outil puissant pour découvrir de nouvelles fonctions biologiques rythmiquement orchestrées et non identifiables par des techniques de génomique ou de protéomique réalisées sur des extraits cellulaires totaux. Les données obtenues constituent une étape importante vers l’identification de nouvelles fonctions biologiques dirigées par l’horloge circadienne et/ou les rythmes alimentaires. La purification d’organelles associées à la protéomique quantitative in vivo constitue une nouvelle voie prometteuse non seulement pour l’étude de la biologie circadienne, mais également pour l’étude de nombreuses fonctions dans différents modèles et systèmes où les mécanismes moléculaires sont encore peu et mal décrits.
Références
- Atger, F., Gobet, C., Marquis, J., Martin, E., Wang, J., Weger, B., Lefebvre, G., Descombes, P., Naef, F., Gachon, F. (2015). Circadian and feeding rhythms differentially affect rhythmic mRNA transcription and translation in mouse liver. Proc Natl Acad Sci USA, 112, E6579-E6588. [CrossRef] [Google Scholar]
- Atger, F., Mauvoisin, D., Weger, B., Gobet, C., Gachon, F. (2017). Regulation of mammalian physiology by interconnected circadian and feeding rhythms. Front Endocrinol, 8, 42. [CrossRef] [Google Scholar]
- Damiola, F., Le Minh, N., Preitner, N., Kornmann, B., Fleury-Olela, F., Schibler, U. (2000). Restricted feeding uncouples circadian oscillators in peripheral tissues from the central pacemaker in the suprachiasmatic nucleus. Genes Dev, 14, 2950-2961. [CrossRef] [PubMed] [Google Scholar]
- Gentric, G., Desdouets, C. (2014). Polyploidization in liver tissue. Am J Pathol, 184, 322-331. [CrossRef] [PubMed] [Google Scholar]
- Hastings, M.H., Maywood, E.S., Brancaccio, M. (2018). Generation of circadian rhythms in the suprachiasmatic nucleus. Nat Rev Neurosci, 19, 453-469. [CrossRef] [Google Scholar]
- Jouffe, C., Cretenet, G., Symul, L., Martin, E., Atger, F., Naef, F., Gachon, F. (2013). The circadian clock coordinates ribosome biogenesis. PLoS Biol, 11, e1001455. [CrossRef] [PubMed] [Google Scholar]
- Matsuo, T., Yamaguchi, S., Mitsui, S., Emi, A., Shimoda, F., Okamura, H. (2003). Control mechanism of the circadian clock for timing of cell division in vivo. Science, 302, 255-259. [CrossRef] [PubMed] [Google Scholar]
- Mauvoisin, D., Wang, J., Jouffe, C., Martin, E., Tger, F., Waridel, P., Quadroni, M., Gachon, F., Naef, F. (2014). Circadian clock-dependent and -independent rhythmic proteomes implement distinct diurnal functions in mouse liver. Proc Natl Acad Sci USA, 111, 167-172. [CrossRef] [Google Scholar]
- Mauvoisin, D., Atger, F., Dayon, L., Núñez Galindo, A., Wang, J., Martin, E., Da Silva, L., Montoliu, I., Collino, S., Martin, F.-P., Ratajczak, J., Cantó, C., Kussmann, M., Naef, F., Gachon, F. (2017). Circadian and feeding rhythms orchestrate the diurnal liver acetylome. Cell Rep, 20, 1729-1743. [CrossRef] [Google Scholar]
- Panda, S. (2016). Circadian physiology of metabolism. Science, 354, 1008-1015. [CrossRef] [PubMed] [Google Scholar]
- Peek, C.B., Affinati, A.H., Ramsey, K.M., Kuo, H.-Y., Yu, W., Sena, L.A., Ilkayeva, O., Marcheva, B., Kobayashi, Y., Omura, C., Levine, D.C., Bacsik, D.J., Gius, D., Newgard, C.B., Goetzman, E., Chandel, N.S., Denu, J.M., Mrksich, M., Bass, J. (2013). Circadian clock NAD+ cycle drives mitochondrial oxidative metabolism in mice. Science, 342, 6158. [CrossRef] [Google Scholar]
- Reddy, A.B., Karp, N.A, Maywood, E.S., Sage, E.A., Deery, M., O’Neill, J.S., Wong, G.K.Y., Chesham, J., Odell, M., Lilley, K.S., Kyriacou, C.P., Hastings, M.H. (2006). Circadian orchestration of the hepatic proteome. Curr Biol, 16, 1107-1115. [CrossRef] [PubMed] [Google Scholar]
- Shi, L., Tu, B.P. (2015). Acetyl-CoA and the regulation of metabolism: mechanisms and consequences. Curr Opin Cell Biol, 33, 125-131. [CrossRef] [Google Scholar]
- Sinturel, F., Gerber, A., Mauvoisin, D., Wang, J., Gatfield, D., Stubblefield, J.J., Green, C.B., Gachon, F., Schibler, U. (2017). Diurnal oscillations in liver mass and cell size accompany ribosome assembly cycles. Cell, 169, 651-663.e614. [CrossRef] [Google Scholar]
- Takahashi, J.S. (2016). Transcriptional architecture of the mammalian circadian clock. Nat Rev Gen, 18, 164-179. [CrossRef] [Google Scholar]
- Wang, J., Mauvoisin, D., Martin, E., Atger, F., Galindo, A.N., Dayon, L., Sizzano, F., Palini, A., Kussmann, M., Waridel, P., Quadroni, M., Dulić, V., Naef, F., Gachon, F. (2017). Nuclear proteomics uncovers diurnal regulatory landscapes in mouse liver. Cell Metab, 25, 102-117. [CrossRef] [PubMed] [Google Scholar]
Citation de l’article : Gachon, F. (2018) Protéomique circadienne. Biologie Aujourd'hui, 212, 55-59
Liste des figures
 |
Figure 1 Orchestration de la physiologie rythmique du foie mise en évidence par protéomique nucléaire quantitative. La combinaison de l’analyse quantitative et temporelle du protéome et du phosphoprotéome nucléaire in vivo nous a permis de mettre en évidence la rythmicité de nombreux processus cellulaires fondamentaux tels que la régulation transcriptionnelle, l’assemblage des ribosomes et la réparation de l’ADN. Les cycles cellulaire et diurne semblent alignés et la polyploïdie rythmique que nous observons semblerait être une conséquence de ce lien. © Med Sci (Paris) 2017, 33, 573-576. |
| Dans le texte | |
 |
Figure 2 L’acétylation rythmique des protéines présente une phase différente selon la localisation subcellulaire des protéines. Les rythmes alimentaires et l’horloge circadienne contrôlent cette acétylation, respectivement en régulant la synthèse d’acétly-CoA et du NAD+ dont dépend l’activité de la déacétylase SIRT3. © Cell Rep 2017, 20, 1729-1743. |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.


