| Numéro |
Biologie Aujourd'hui
Volume 211, Numéro 4, 2017
|
|
|---|---|---|
| Page(s) | 255 - 264 | |
| Section | CRISPR : d’un système immunitaire procaryote à une révolution technologique (Journée Claude Bernard) | |
| DOI | https://doi.org/10.1051/jbio/2018005 | |
| Publié en ligne | 29 juin 2018 | |
Article
Incontournables et pourtant rares : les déterminants de la distribution des systèmes CRISPR-Cas dans les génomes bactériens
Why so rare if so essentiel: the determinants of the sparse distribution of CRISPR-Cas systems in bacterial genomes
1
Synthetic Biology Group, Institut Pasteur,
25–28 rue Dr. Roux,
75015
Paris, France
2
Microbial Evolutionary Genomics, Institut Pasteur,
25–28 rue Dr Roux,
75015
Paris, France
3
AgroParisTech,
75005
Paris, France
* Auteur correspondant : aude.bernheim@gmail.com
Reçu :
1
Mars
2018
Les systèmes CRISPR-Cas (Cluster of Regularly Interspaced Short Palindromic Repeats) confèrent aux bactéries une immunité adaptative contre les éléments génétiques mobiles, jouant ainsi un rôle important dans l’évolution bactérienne. Cependant, malgré la protection qu’ils apportent, moins de la moitié des génomes bactériens encodent des systèmes CRISPR-Cas. En comparaison, 90 % des archées ont des systèmes CRISPR-Cas et un génome bactérien code pour en moyenne deux systèmes de restriction-modification. Cette faible prévalence est d’autant plus intrigante que les systèmes CRISPR-Cas sont massivement transférés horizontalement. Cette revue s’attache à décrire la distribution des systèmes CRISPR-Cas dans les génomes bactériens et à explorer les différentes hypothèses pouvant expliquer pourquoi les systèmes CRISPR-Cas sont présents dans un nombre relativement limité de ces génomes. Les mécanismes d’évasion des phages, les facteurs écologiques tels que la diversité et l’abondance des phages ou les coûts intrinsèques liés à la maintenance du système ou aux phénomènes d’auto-immunité seront notamment discutés. Comprendre les inconvénients à encoder des systèmes CRISPR-Cas est essentiel pour appréhender leurs dynamiques évolutives et leurs succès relatifs dans certains environnements ou clades.
Abstract
CRISPR-Cas (Cluster of Regularly Interspaced Short Palindromic Repeats) systems confer bacteria and archaea an adaptative immunity against phages and other invading genetic elements playing an important role in bacterial evolution. However, despite the protection they generate and high rate of horizontal transfer, less than 50% of bacterial genomes harbor a CRISPR-Cas system. As a comparison, 90% of archaea encode a CRISPR-Cas system and a bacterial genome codes for two restriction modification systems on average. This review describes CRISPR-Cas systems distribution in bacterial genomes and then details the different hypotheses put forward to explain the relative scarcity of CRISPR-Cas systems. More specifically, phage escape mechanisms, ecological factors such as phage diversity and abundance and intrinsic costs, such as maintenance or autoimmunity, are discussed. Overall, a better understanding of the downsides of encoding CRISPR-Cas systems is essential to explain their evolutionary dynamics and their relative success in different environments and clades.
Mots clés : CRISPR-Cas / distribution CRISPR-Cas / transfert horizontal / auto-immunité / bactériophages
Key words: CRISPR-Cas / CRISPR-Cas distribution / horizontal transfer / autoimmunity / bacteriophages
© Société de Biologie, 2018
Les systèmes CRISPR-Cas sont des systèmes immunitaires adaptatifs des bactéries
Au cours des dix dernières années, les systèmes CRISPR-Cas ont passionné la communauté scientifique à la fois en raison de leur rôle de système immunitaire adaptatif dans les bactéries et de leur utilisation dans de nombreuses applications biotechnologiques, en particulier pour éditer le génome. Alors que de nombreux efforts ont été entrepris pour comprendre en détail la biologie complexe et les mécanismes moléculaires de ces systèmes, les questions d’évolution restent multiples.
Les systèmes CRISPR-Cas constituent un processus immunitaire adaptatif des bactéries et des archées ciblant les éléments génétiques mobiles (MGE) tels que les phages ou les plasmides (Barrangou et al., 2007 ; Brouns et al., 2008 ; Marraffini et Sontheimer, 2008). Ils sont composés d’une série de courtes séquences répétées appelées CRISPR (courtes répétitions palindromiques groupées et régulièrement espacées) et d’un groupe de gènes cas (CRISPR associated genes). Les CRISPR comprennent deux types de séquences : les répétitions et les spacers. Les répétitions sont des séquences courtes (typiquement 20–40 pb) identiques dans un CRISPR donné. Ces systèmes sont entrecoupés par des spacers, séquences courtes et diverses (typiquement 20–40 pb), qui correspondent souvent à des séquences d’éléments génétiques mobiles. Le second élément des systèmes CRISPR-Cas consiste en un ensemble de gènes cas nécessaires pour réaliser l’immunité (Bolotin et al., 2005 ; Mojica et al., 2005 ; Pourcel et al., 2005 ; Makarova et al., 2006).
L’immunité CRISPR-Cas fonctionne en deux étapes : immunité et immunisation aussi appelée adaptation (Marraffini, 2015) (Figure 1). L’immunité peut être scindée en deux phases : 1) l’expression et la maturation du crRNA, 2) l’interférence. Le CRISPR est transcrit puis scindé en ARN plus petits, composés chacun d’une répétition et d’un seul spacer appelé crRNA (CRISPR RNA). Chacun de ces ARN sert alors de guide pour un complexe de protéines Cas. Si la séquence d’un guide est identique à une autre séquence d’ADN dans la cellule, comme par exemple l’ADN d’un phage, le complexe activera une réponse immunitaire, la plupart du temps en coupant l’ADN envahisseur qui sera ensuite dégradé. Pendant l’adaptation, un complexe de protéines Cas génère puis incorpore un nouveau spacer dans le locus CRISPR. Un aspect important du système est la capacité à distinguer son propre ADN d’un ADN exogène. Dans de nombreux cas, une courte séquence appelée PAM (Protospacer Adjacent Motif) est essentielle pour ce processus. Présente à proximité d’un protosopacer (séquence d’un spacer sur un élément génétique mobile) mais absente dans le locus CRISPR, la PAM est nécessaire pour que l’interférence ait lieu (Briner et al., 2015).
 |
Figure 1 Vue d’ensemble du mécanisme CRISPR-Cas. Un locus CRISPR-Cas est organisé autour de deux éléments principaux : un groupe de gènes cas et un CRISPR composé de spacers et de répétitions (en noir). L’immunité CRISPR-Cas fonctionne en deux phases principales : l’adaptation où le complexe de protéines Cas permet l’acquisition de nouveaux spacers et l’immunité ciblée. a. Pendant la phase d’adaptation, les protéines Cas forment un complexe (1) capable de générer un court fragment d’ADN à partir de l’ADN étranger présent dans la cellule (2) qui est ensuite intégré dans le CRISPR comme nouveau spacer (3). b. Pendant la phase d’immunité, le CRISPR est transcrit (4) et scindé en petits ARN (5) qui servent de guides pour la reconnaissance de la cible pour les protéines Cas (6), qui dégradent l’ADN (7). Figure adaptée de Bernheim (2017). |
La diversité des systèmes CRISPR-Cas
Les systèmes CRISPR-Cas sont extrêmement divers. Ils sont distingués en deux classes, six types (I à VI) et 21 sous-types (Makarova et al., 2015) (Figure 2). De nouveaux sous-types sont découverts chaque année, menant à une classification évoluant régulièrement (Burstein et al., 2016a, b ; Shmakov et al., 2017). La classification des systèmes CRISPR-Cas est fondée sur l’architecture des loci et sur le contenu des clusters Cas, plus spécifiquement sur les protéines signatures de chaque type (Makarova et al., 2015) : Cas3 pour le type I, Cas9 pour le type II, Cas10 pour le type III, Csf4 pour le type IV, Cas12 pour le type V et Cas13 pour le type VI. Un aspect clé de l’organisation des systèmes CRISPR-Cas est leur modularité (Mohanraju et al., 2016). Les divers systèmes CRISPR-Cas sont organisés autour de quatre modules : traitement de l’ARNr, reconnaissance de la cible, clivage de la cible et adaptation. Alors que le module d’adaptation est largement similaire dans tous les systèmes CRISPR-Cas et implique Cas1 et Cas2, les autres modules peuvent englober un très petit ou un grand nombre de protéines (Makarova et al., 2015 ; Mohanraju et al., 2016).
Les systèmes CRISPR-Cas de Classe 1 sont divisés en trois types et 12 sous-types (Makarova et al., 2015). Les systèmes de Classe 1 sont définis par l’utilisation d’effecteurs multi-protéiques, c’est-à-dire que la phase d’interférence est réalisée par un complexe de protéines multiples. Ils sont de loin les plus abondants car ils représentent 90 % des systèmes CRISPR-Cas (Makarova et al., 2015). D’autre part, les systèmes CRISPR-Cas de Classe 2 ne représentent que 10 % des systèmes CRISPR-Cas et sont presque complètement absents chez les archées (Makarova et al., 2015). Leur effecteur est constitué d’une seule protéine multi-domaines. Les technologies CRISPR-Cas sont dérivées de sous-types appartenant à cette classe. La diversité des protéines Cas reflète la diversité des mécanismes moléculaires par lesquels les systèmes CRISPR-Cas mettent en œuvre l’immunité adaptative.
Selon une hypothèse actuelle, les systèmes CRISPR-Cas auraient émergé à la suite de l’insertion d’un casposon à proximité d’une protéine analogue à Cas10 (Koonin et Krupovic, 2014). Les casposons constituent une classe de transposons auto-synthétisants qui codent pour un homologue de Cas1 (Krupovic et al., 2014). Les systèmes de classe 1 étant les plus répandus (Makarova et al., 2013), les effecteurs de classe 1 sont considérés comme la forme ancestrale. Les variants de classe 2 auraient évolué par le remplacement du locus effecteur par des protéines uniques provenant de différents éléments génétiques mobiles. Les systèmes de type II dériveraient des protéines des transposons IscB (Kapitonov et al., 2015), tandis que les systèmes de type V seraient liés aux transposons TnpB (Shmakov et al., 2017). Enfin, les ancêtres du type VI n’ont pas encore été déterminés avec précision, mais dériveraient des protéines Cas contenant des domaines HEPN (Koonin et Makarova, 2017).
 |
Figure 2 La diversité des systèmes CRISPR-Cas. Classification des systèmes CRISPR-Cas. Les systèmes CRISPR-Cas sont divisés en deux classes, six types (I à VI) et 21 sous-types. La classification est basée sur des protéines signatures et l’architecture des loci. Figure adaptée de Bernheim (2017). |
Une distribution éclatée des systèmes CRISPR-Cas malgré des transferts horizontaux constants
Les systèmes CRISPR-Cas sont présents dans plus de 90 % des archées et dans environ 50 % des bactéries (Makarova et al., 2015). De nombreuses études de métagénomique ont révélé la présence de systèmes CRISPR-Cas dans des environnements aussi divers que l’intestin, le rumen, la neige en Antarctique ou des sources chaudes (Heidelberg et al., 2009 ; Stern et al., 2012 ; Rho et al., 2012 ; Lopatina et al., 2016).
Cette large distribution peut en partie s’expliquer par l’observation selon laquelle les systèmes CRISPR-Cas sont massivement transférés horizontalement. Plusieurs études phylogénétiques ont par exemple signalé l’incongruence entre l’arbre phylogénétique des gènes cas et celui des espèces (Godde et Bickerton, 2006 ; Chakraborty et al., 2010). De plus, des systèmes CRISPR-Cas ont été détectés sur divers éléments génétiques mobiles. Ainsi, on trouve des systèmes CRISPR-Cas sur des mégaplasmides dans diverses bactéries dont Treponema denticola, Vibrio vulnificus, Yersinia pestis (Godde et Bickerton, 2006). Des systèmes CRISPR-Cas portés par des plasmides ont aussi été caractérisés expérimentalement (Guo et al., 2011 ; Millen et al., 2012 ; Scholz et al., 2013). Les phages codent également pour des systèmes CRISPR-Cas. Par exemple, des CRISPR ont été détectés dans des prophages de Clostridium (Soutourina et al., 2013). Expérimentalement, l’exemple du phage ICP1 de Vibrio cholera qui code pour un système fonctionnel CRISPR-Cas de Type I-F a permis de montrer un mécanisme de piratage de ce système de défense pour le retourner contre la bactérie (Seed et al., 2013). Enfin, il a été récemment démontré que les transposons de type Tn7 encodent des systèmes CRISPR-Cas minimaux de type I-F et de type I-B (Peters et al., 2017).
Cependant, la présence des systèmes CRISPR-Cas est potentiellement surestimée. Une récente étude sur les filtrats d’eaux souterraines a révélé que dans cet environnement seulement 10 % des bactéries codent pour des systèmes CRISPR-Cas (Burstein et al., 2016a, b). Plus étonnant, certains phylums étaient complètement dépourvus de systèmes CRISPR-Cas (Burstein et al., 2016a, b). L’étude de ces phylums dans d’autres environnements a confirmé que l’absence de systèmes CRISPR-Cas n’était pas limitée à un environnement spécifique (Burstein et al., 2016a, b). L’écart entre ces détections peut s’expliquer par les génomes étudiés. L’abondance de certains clades dans les filtrats d’eau souterraine diffère de l’abondance dans la base de données de génomes entièrement séquencés et pourrait suggérer que les systèmes CRISPR-Cas sont moins avantageux dans cet environnement spécifique. Au total, leur prévalence semble assez faible par rapport à d’autres systèmes de défense tels que la modification de restriction, dont on trouve en moyenne deux copies par génome (Oliveira et al., 2016). Même à l’intérieur d’une espèce, les souches diffèrent en termes de présence de systèmes CRISPR-Cas (Westra et al., 2016). Cette relative rareté soulève des questions relatives aux coûts potentiels des systèmes CRISPR-Cas.
Plusieurs études ont également signalé que les systèmes CRISPR-Cas ne sont pas répartis uniformément dans différents environnements. Plus spécifiquement, les environnements à haute température englobent plus d’organismes codant pour des systèmes CRISPR-Cas (Makarova et al., 2006 ; Anderson et al., 2011 ; Weinberger et al., 2012). Un enrichissement en systèmes CRISPR-Cas a également été rapporté dans les métagénomes microbiens associés aux éponges marines (Horn et al., 2016) alors que les filtrats des eaux souterraines ont montré une prévalence réduite des systèmes CRISPR-Cas (Burstein et al., 2016a, b). Cette distribution dispersée souligne l’impact des facteurs écologiques sur la distribution des systèmes CRISPR-Cas.
Pour expliquer la variation de présence, du nombre et de la fonction des loci CRISPR-Cas dans et entre les espèces bactériennes, plusieurs études plaident pour une dynamique de perte fréquente des systèmes CRISPR-Cas qui sont ensuite regagnés par transfert horizontal de gènes (Takeuchi et al., 2012 ; Jiang et al., 2013 ; Berezovskaya et al., 2014). Les gènes cas évoluent dans le cadre d’une sélection négative qui est généralement beaucoup plus faible que la force médiane de la sélection négative, ce qui suggère qu’ils peuvent être facilement perdus (Takeuchi et al., 2012). Différentes études expérimentales chez Staphylococcus epidermidis, Thermoanaerobacter et Mycoplasma gallisepticum ont rapporté des pertes fréquentes des systèmes CRISPR-Cas (Delaney et al., 2012 ; Jiang et al., 2013 ; Mai et al., 2016). Chez Staphylococcus epidermidis, la perte de systèmes CRISPR-Cas, même par de grandes suppressions, a un coût faible voire inexistant (Jiang et al., 2013). Les preuves d’un transfert fréquent et de pertes répétées ont conduit à supposer que les systèmes CRISPR-Cas sont en état continu de flux, étant perdus quand ils supportent un coût ou lorsqu’ils sont neutres, puis réacquis par transfert horizontal (Jiang et al., 2013).
Une efficacité limitée par des mécanismes d’évasion des bactériophages
Les bactéries et les phages sont engagés dans une course aux armements constante. Comme avec n’importe quel système de défense, les phages ont trouvé des moyens d’échapper à l’immunité CRISPR-Cas en utilisant une variété de mécanismes qui impliquent la modification du génome ou des protéines spécifiques anti-CRISPR (Figure 3).
Les phages ont un taux de mutation élevé qui leur permet de s’adapter rapidement (Drake, 1991). L’hypothèse a été émise puis confirmée, selon laquelle les phages pouvaient échapper aux systèmes CRISPR-Cas par des mutations simples. Plusieurs expériences de co-évolution entre des bactéries encodant un système CRISPR-Cas fonctionnel et un phage ont été menées chez Streptococcus thermophilus (Deveau et al., 2008 ; Sun et al., 2013 ; Paez-Espino et al., 2015) et chez Synechococcus (Heidelberg et al., 2009). Les études rapportent l’accumulation de mutations uniques chez les phages dans les régions ciblées par CRISPR. Plus spécifiquement, la séquence PAM est plus fréquemment mutée que le reste du protospacer. Pour certains sous-types CRISPR-Cas, il existe une tolérance pour la spécificité de séquence, les mutations dans la PAM assurent l’abolition complète de l’immunité CRISPR-Cas.
Des expériences de co-évolution ont également conduit à l’observation de réarrangements génomiques (Paez-Espino et al., 2015). Il a été rapporté que la présence de phages divers augmentait leur persistance. L’analyse des génomes a révélé que les événements de recombinaison se produisaient entre les phages, générant ainsi des phages chimériques qui ne contenaient pas de régions ciblées par CRISPR (Paez-Espino et al., 2015). Des observations similaires ont été faites lors de l’étude d’écosystèmes naturels (Andersson et Banfield, 2008).
Les phages peuvent également modifier leurs génomes par l’attachement de groupes chimiques à leur ADN (Bryson et al., 2015). Ces formes de modifications de l’ADN sont utilisées pour échapper à d’autres systèmes immunitaires bactériens comme les systèmes de restriction-modification (Warren, 1980). Ainsi, un phage T4 contenant une modification covalente de l’ADN où la cytosine est remplacée par la glucosyl-hydroxyméthylcytosine est résistant à l’interférence CRISPR-Cas9 (Bryson et al., 2015). Cependant, les modifications du génome ne sont pas le seul moyen par lequel la course aux armements opère.
Les phages codent pour de petites protéines qui inhibent les systèmes CRISPR-Cas et sont nommées anti-CRISPR (Borges et al., 2017 ; Sontheimer et Davidson 2017). La découverte des anti-CRISPR provient de l’observation inattendue selon laquelle des souches lysogènes de P. aeruginosa encodant certains prophages spécifiques étaient sensibles à certains phages lytiques alors que les non lysogènes étaient résistantes (Bondy-Denomy et al., 2013). Ce phénotype s’explique par la présence de protéines codées par les prophages inhibant les systèmes CRISPR-Cas de Type I-F, autrement actifs et conférant une résistance aux phages lytiques. Depuis, des anti-CRISPR ont été découverts contre les systèmes CRISPR-Cas de type I-E, I-F, II-A et II-C (Pawluk et al., 2014, Pawluk et al., 2016 ; Rauch et al., 2016 ; Hynes et al., 2017). Ils sont encodés par des phages tempérés et lytiques (Borges et al., 2017 ; Hynes et al., 2017). Leurs mécanismes d’actions varient. Ils peuvent empêcher la reconnaissance de la cible en se liant au complexe effecteur ou à la nucléase dans les systèmes de type I (Wang J. et al., 2016 ; Wang X. et al., 2016 ; Chowdhury et al., 2017). Dans les systèmes de type II, ils peuvent entraver la liaison à l’ADN guidé (Dong et al., 2017 ; Yang & Patel, 2017 ; Shin et al., 2017). Les anti-CRISPR sont répandus ; 64 % des 449 CRISPR-Cas type I-F de P. aeruginosa, 53 % de 81 CRISPR-Cas type I-F de P. aeruginosa, et 50 % CRISPR-Cas type II-A de L. monocytogenes sont sensibles à des anti-CRISPR (Bondy-Denomy et al., 2015 ; Rauch et al., 2016). Les anti-CRISPR ont suscité beaucoup d’intérêt pour leur potentielle utilisation biotechnologique. Ils pourraient en effet être utilisés pour réguler des protéines telles que Cas9, ce qui est essentiel pour certaines applications spécifiques.
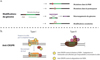 |
Figure 3 Les mécanismes de résistance des phages à l’immunité CRISPR-Cas. a. Des modifications du génome telles que des mutations dans la séquence PAM, ou dans les séquences cibles, des réarrangements de génomes ou des modifications covalentes de l’ADN permettent aux phages d’échapper à l’immunité CRISPR-Cas. b. Les protéines anti-CRISPR ou Acr (polygones jaunes ou roses) inhibent les systèmes CRISPR-Cas de manière dépendante du type et même du sous-type, en se liant à différents domaines des protéines Cas impliquées dans la reconnaissance de la cible ou le clivage de l’ADN. Figure adaptée de Bernheim (2017). |
Des facteurs écologiques viennent expliquer cette distribution clairsemée
Les bactéries possèdent un arsenal de divers systèmes immunitaires pour contrer les phages. Étant donné la faible abondance des systèmes CRISPR-Cas par rapport à certains mécanismes de défense, il est essentiel de comprendre comment ces différents systèmes de défense interagissent et dans quelles conditions certains systèmes spécifiques seront favorisés par rapport aux autres. Une étude dans S. thermophilus a démontré que les systèmes CRISPR-Cas et de restriction-modification pouvaient être compatibles et même fonctionner de manière synergique, augmentant ainsi la résistance aux phages (Dupuis et al., 2013). Au contraire, un autre système expérimental permet à une souche de P. aeruginosa de développer une résistance face au phage DMS3 soit par CRISPR-Cas, soit par une modification de surface, montrant que les systèmes de défense ne fonctionnent pas toujours en synergie (Westra et al., 2015).
Un facteur clé pour l’émergence des systèmes CRISPR-Cas est la faible diversité de phages (Weinberger et al., 2012 ; Iranzo et al., 2013). Deux modèles différents ont prédit que les avantages des systèmes CRISPR-Cas diminueraient avec l’augmentation de la diversité génétique des virus (Weinberger et al., 2012 ; Iranzo et al., 2013). Au-dessus d’une valeur seuil de diversité virale, le système CRISPR-Cas deviendrait inefficace car il ne serait pas capable de coder suffisamment de spacers pour cibler la population de phages et serait donc à terme perdu. Comme la diversité génétique dépend entre autres déterminants des taux de mutation et de la taille de la population, ces deux facteurs auront un impact sur le maintien des systèmes CRISPR-Cas dans une population. L’importante prévalence des systèmes CRISPR-Cas chez les thermophiles pourrait s’expliquer par des taux de mutation plus faibles chez les thermophiles que chez les mésophiles (Weinberger et al., 2012). La taille de la population pourrait aussi expliquer cette prévalence élevée (Iranzo et al., 2013). Comme les populations de mésophiles sont plus importantes que celle des thermophiles, une plus grande diversité de phages émergerait dans une population large. Les systèmes CRISPR-Cas deviendraient inefficaces dans ce contexte (Iranzo et al., 2013). Enfin, la diversité virale peut également permettre l’émergence de nouveaux mécanismes d’échappement tels que la recombinaison entre les phages sensibles à un spacer spécifique limitant encore plus l’immunité CRISPR-Cas (Paez-Espino et al., 2015).
Un deuxième facteur clé pour l’émergence des systèmes CRISPR-Cas est la faible abondance des phages. Les principales conclusions sur l’importance de la force d’infection, c’est-à-dire l’abondance de phages, proviennent d’une étude expérimentale chez P. aeruginosa (Westra et al., 2015). Dans cette étude, P. aeruginosa peut développer soit une résistance due à une modification de surface, soit un système CRISPR-Cas pour échapper au phage DMS3. Lorsque l’expérience de co-évolution est réalisée dans un milieu à ressources élevées tel que le LB (Lysogeny Broth, milieu de culture riche), elle conduit à l’émergence de modifications presque exclusivement de surface alors que dans un milieu à ressources faibles, le contraire est observé avec une résistance presque exclusivement CRISPR-Cas. Ces observations s’expliquent par un paramètre : l’abondance des phages. Face à des infections rares, un système inductible comme CRISPR-Cas sera favorisé, tandis que des infections constantes favoriseront un type de défense constitutive.
Coûts intrinsèques à coder pour un système CRISPR-Cas
Au-delà des facteurs écologiques, la rareté relative et la perte fréquente des systèmes CRISPR-Cas suggèrent qu’il existe des coûts intrinsèques à encoder des systèmes CRISPR-Cas, tels que les coûts de maintenance et d’expression du système, ceux liés aux phénomènes d’auto-immunité et ceux de limitation du transfert horizontal de gènes (Figure 4).
Il est assez difficile de déterminer le coût d’expression d’un système CRISPR-Cas car les systèmes sont très diversifiés aussi bien en termes de protéines que de régulation. Le coût de l’expression des protéines Cas du type II-A et de l’acquisition de nouveaux spacers a été mesuré chez S. thermophilus. Le coût principal provenait de l’expression des protéines Cas, tandis que l’ajout de spacers supplémentaires n’étaient associés à aucun coût, ce qui a conduit les auteurs à conclure que le coût était principalement dû au maintien du CRISPR-Cas (Vale et al., 2015). Dans une seconde étude sur P. aeruginosa, les systèmes CRISPR-Cas de type I-F se sont avérés avoir un coût dépendant de l’infection mais pas de la maintenance (Westra et al., 2015). Les deux études aboutissant à des conclusions différentes, d’autres travaux sur différentes espèces et types de systèmes CRISPR-Cas pourraient aider à clarifier ce problème.
Un deuxième coût potentiel généré par les systèmes CRISPR-Cas est lié à l’auto-immunité. Des spacers ciblant le propre chromosome de la bactérie ont été détectés dès les premières études postulant la fonction immunitaire des systèmes CRISPR-Cas (Pourcel et al., 2005). Chez Yersinia pestis, sur 36 spacers analysés, 8 spacers correspondaient aux séquences du chromosome Yersinia (Pourcel et al., 2005). Une étude plus générale des CRISPR provenant de 330 organismes a montré qu’un spacer sur 250 cible le chromosome bactérien et que cette auto-immunité se produit dans 18 % de tous les organismes porteurs de CRISPR (Stern et al., 2010). Chez E. coli, une étude a démontré expérimentalement que dans ce contexte, le taux d’incorporation de spacers ciblant le chromosome est de l’ordre de 1 à 1000 (Díez-Villaseñor et al., 2013 ; Levy et al., 2015).
Le résultat le plus commun de ce phénomène d’auto-immunité est la mort cellulaire. Même si la mortalité due à l’auto-immunité est élevée, les bactéries peuvent survivre par plusieurs moyens. La pression de ces phénomènes peut mener au remodelage du génome. Chez Pectobacterium atrosepticum, le système CRISPR-Cas de type I-F possède un spacer ciblant son propre chromosome, plus précisément un îlot de pathogénicité des plantes. La forte toxicité générée par le ciblage du chromosome impose une forte pression de sélection qui peut entraîner des altérations génomiques à grande échelle, y compris le remodelage ou la suppression de l’ensemble des îlots de pathogénicité préexistants (Vercoe et al., 2013). Des résultats similaires ont été rapportés pour le système CRISPR-Cas de type II-A de S. thermophilus. Confrontées à des spacers ciblant le chromosome, 99 % des bactéries sont mortes, mais les survivantes présentaient de grandes délétions des régions ciblées qui étaient le résultat de la recombinaison entre les éléments de séquence d’insertion (IS) (Selle et al., 2015).
Fournir une résistance aux éléments génétiques étrangers, par exemple par l’immunité CRISPR-Cas, peut dans certains cas être délétère. En effet, les bactéries s’adaptent par transfert de gènes horizontal, et la limitation du flux de ce transfert de gènes pourrait empêcher les bactéries d’acquérir des caractères souhaitables. Deux études ont apporté des indices pour soutenir cette affirmation. Premièrement, une étude a examiné l’avantage relatif de coder un système CRISPR-Cas ou d’acquérir un gène de résistance aux antibiotiques par transfert de plasmide chez S. epidermis (Jiang et al., 2013). Les auteurs ont programmé le système CRISPR-Cas pour cibler un plasmide portant une cassette de résistance aux antibiotiques. Après exposition aux antibiotiques, des mutants CRISPR-Cas apparaissaient rapidement dans cette population, favorisant ainsi l’acquisition du plasmide portant la cassette de résistance aux antibiotiques plutôt que le maintien du système CRISPR-Cas (Jiang et al., 2013). Une autre étude a répété l’expérience qui a conduit à la découverte du transfert horizontal de gènes : des souches de S. pneumoniae sans gènes de capsule ont été utilisés pour infecter des souris (Bikard et al., 2012). Sans capsules, l’infection échoue. Cependant, lorsque ces souches sont utilisées pour infecter des souris en présence de Pneumocoques encapsulés et tués par la chaleur, l’infection réussit grâce au transfert de gènes de capsules des Pneumocoques vers les S. pneumoniae. En répétant l’expérience avec des souches codant pour des systèmes CRISPR-Cas ciblant des gènes de capsules, seuls les mutants ayant inactivé le système CRISPR-Cas ont survécu au système immunitaire des souris et ont réussi à générer une infection. Cela suggère que la perte du système CRISPR-Cas était adaptative dans ces conditions (Bikard et al., 2012). Enfin, une analyse de génomes de C. jejuni a montré que certaines souches encodant des systèmes CRISPR-Cas défectueux portaient un prophage ou un plasmide qui conférait de la virulence à la souche. Ces éléments génétiques mobiles étaient absents du génome de deux souches étroitement apparentées avec des systèmes CRISPR-Cas fonctionnels (Louwen et al., 2013). Ces observations soulignent que les avantages liés aux transferts horizontaux de gènes peuvent générer une sélection contre les systèmes CRISPR-Cas.
Certains mécanismes limitent les coûts différents des systèmes CRISPR-Cas décrits ci-dessus. D’abord, la régulation et plus spécifiquement la répression ou l’expression inductible des systèmes CRISPR-Cas peuvent réduire les coûts intrinsèques. Les systèmes CRISPR-Cas sont régulés par divers mécanismes (Patterson et al., 2017). La régulation la plus célèbre des systèmes CRISPR-Cas provient de E. coli, car son système CRISPR-Cas endogène de type I-E n’est pas exprimé dans des conditions de laboratoire (Westra et al., 2010). Le système CRISPR-Cas de type I-E est réprimé par la structuration nucléoïde de type histone (H-NS). Cette inhibition peut être relâchée par l’activateur de transcription LeuO (Westra et al., 2010). Ce type de répression n’est pas spécifique à E. coli mais partagé par Salmonella enterica sérotype Typhi et Klebsiella pneumoniae (Medina-Aparicio et al., 2011 ; Lin et al., 2016).
D’autre part, une adaptation dite « primed » a été proposée comme mécanisme de limitation des effets de l’auto-immunité. Dans certains types de systèmes CRISPR-Cas comme les I-E et les I-F, l’acquisition de nouveaux spacers est beaucoup plus efficace, lorsqu’un spacer cible déjà l’élément. Ce mécanisme permettrait de promouvoir l’acquisition de spacers contre des éléments génétiques mobiles spécifiques et de limiter l’auto-acquisition (Fineran et al., 2014).
À court terme, ces mécanismes limitent les coûts et pourraient favoriser la maintenance des systèmes CRISPR-Cas. Dans les organismes où les systèmes CRISPR-Cas sont régulés, leur perte pourrait donc s’expliquer par une longue période d’absence de sélection plus que par les coûts constitutifs.
En conclusion, les systèmes CRISPR-Cas restent relativement rares pour un système immunitaire. Alors que les facteurs écologiques semblent jouer un rôle majeur dans leur présence inégale dans divers environnements, ils ne suffisent pas à expliquer leur distribution dispersée dans les génomes bactériens. Plusieurs hypothèses ont été avancées pour expliquer les pertes fréquentes observées : les coûts de maintenance, d’auto-immunité et de limitation du transfert horizontal de gènes. Cependant, aucune de ces explications ne résout entièrement cette question. Premièrement, il n’y a pas de raison claire pour laquelle le coût de l’auto-immunité devrait varier entre les clades. Deuxièmement, le coût du maintien des systèmes de défense et de la prévention du transfert horizontal de gènes est général pour tous les systèmes de défense. Cette question reste donc ouverte et de nouvelles hypothèses sont nécessaires pour mieux comprendre la distribution des systèmes CRISPR chez les bactéries.
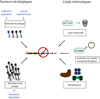 |
Figure 4 les inconvénients à coder pour des systèmes CRISPR-Cas. La relativement faible abondance des systèmes CRISPR-Cas peut s’expliquer à la fois par des facteurs écologiques, notamment la diversité et l’abondance des phages, et par des coûts intrinsèques, y compris le coût d’expression, la limitation du transfert horizontal de gènes et les phénomènes d’auto-immunité. Figure adaptée de Bernheim (2017). |
Références
- Andersson, A.F., Banfield J.F. (2008). Virus population dynamics and acquired virus resistance in natural microbial communities. Science, 320, 1047-1050. [CrossRef] [PubMed] [Google Scholar]
- Anderson, R.E., Brazelton, W.J., Baross J.A. (2011). Using CRISPRs as a metagenomic tool to identify microbial hosts of a diffuse flow hydrothermal vent viral assemblage. FEMS Microbiol Ecol, 77, 120-133. [CrossRef] [PubMed] [Google Scholar]
- Barrangou, R., Frémaux, C., Deveau, H., Richards, M., Boyaval, P., Moineau, S., Romero, D.A., Horvath P. (2007). CRISPR provides acquired resistance against viruses in prokaryotes. Science, 315, 1709-1712. [CrossRef] [Google Scholar]
- Berezovskaya, F.S., Wolf, Y.I., Koonin, E.V., Karev, G.P. (2014). Pseudo-chaotic oscillations in CRISPR-virus coevolution predicted by bifurcation analysis. Biology Direct, 9(1), 13. [CrossRef] [PubMed] [Google Scholar]
- Bernheim, A., The distribution of CRISPR-Cas systems is affected by interactions with DNA repair pathways, PhD Thesis, Université Paris-Descartes, 2017. [Google Scholar]
- Bikard, D., Hatoum-Aslan, A., Mucida, D., Marraffini, L.A. (2012). CRISPR interference can prevent natural transformation and virulence acquisition during in vivo bacterial infection. Cell Host Microbe, 12, 177-186. [CrossRef] [PubMed] [Google Scholar]
- Bolotin, A., Quinquis, B., Sorokin, A., Ehrlich, S.D. (2005). Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 151, 2551-2561. [CrossRef] [PubMed] [Google Scholar]
- Bondy-Denomy, J., Pawluk, A., Maxwell, K.L., Davidson, A.R. (2013). Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system. Nature, 493, 429-432. [CrossRef] [PubMed] [Google Scholar]
- Bondy-Denomy, J., Maxwell, K.L., Garcia, B., Rollins, M. (2015). Multiple mechanisms for CRISPR-Cas inhibition by anti-CRISPR proteins. Nature, 526, 136-139. [CrossRef] [PubMed] [Google Scholar]
- Borges, A.L., Davidson, A.R., Bondy-Denomy, J. (2017). The discovery, mechanisms, and evolutionary impact of anti-CRISPRs. Ann Rev Virol, 4. doi: 10.1146/annurev-virology-101416-041616. [Google Scholar]
- Briner, A.E., Lugli, G.A., Milani, C., Duranti, S, Turroni, F., Gueimonde, M., Margolles, A., van Sinderen, D., Ventura, M., Barrangou, R. (2015). Occurrence and diversity of CRISPR-Cas systems in the genus Bifidobacterium. Plos One, 10, e0133661. [CrossRef] [PubMed] [Google Scholar]
- Brouns, S.J.J., Jore, M.M., Lundgren, M., Westra, E.R., Slijkhuis, R.J.H., Snijders, A.P.L., Dickman, M.J., Makarova, K.S., Koonin, E.V., van der Oost, J. (2008). Small CRISPR RNAs guide antiviral defense in Prokaryotes. Science, 321, 960-964. [CrossRef] [Google Scholar]
- Bryson, A., Hwang, Y., Sherrill-Mix, S., Wu, G.D., Lewis, J.D., Black, L., Clark, T.A., Bushman, F.D. (2015). Covalent modification of bacteriophage T4 DNA inhibits CRISPR-Cas9. mBio 6(32), 1-9. [CrossRef] [Google Scholar]
- Burstein, D., Sun, L.C., Brown, C.T., Sharon, I., Anantharaman, K., Probst, A.J., Thomas, B.C., Banfield J.B. (2016a). Major bacterial lineages are essentially devoid of CRISPR-Cas viral defense systems. Nat Commun, 7, 10613. [CrossRef] [PubMed] [Google Scholar]
- Burstein, D., Harrington, L.B., Strutt, S.C., Probst, A.J. (2016b). New CRISPR-Cas systems from uncultivated microbes. Nature, 542, 237-241. [Google Scholar]
- Chakraborty, S., Snijders, A.P., Chakravorty, R., Ahmed, M., Tarek, A.M, Hossain, M.A. (2010). Comparative network clustering of Direct Repeats (DRs) and Cas Genes confirms the possibility of the horizontal transfer of CRISPR Locus among Bacteria. Mol Phylogenet Evol, 56, 878-887. [CrossRef] [Google Scholar]
- Chowdhury, S., Carter, J., Rollins, M.F., Golden S.M., Jackson, N.R., Hoffmann, C., Nosaka, L., Bondy-Denomy, J., Maxwell, K.L., Davidson, A.R., Fischer, E.R., Lander, G.C., Wiedenheft, B. (2017). Structure reveals mechanisms of viral suppressors that intercept a CRISPR RNA-guided surveillance complex. Cell, 169, 47-57, e11. [CrossRef] [PubMed] [Google Scholar]
- Delaney, N.F., Balenger, S., Bonneaud, C., Marx, C.J., Hill, G.E., Ferguson-Noel, N., Tsai, P., Rodrigo, A., Edwards, S.V. (2012). Ultrafast evolution and loss of CRISPRs following a host shift in a novel wildlife pathogen, Mycoplasma gallisepticum. PLoS Genet, 8, e1002511. [CrossRef] [PubMed] [Google Scholar]
- Deveau, H, Barrangou, R., Garneau, J.E., Labonté, J., Frémaux, C., Boyaval, P., Romero, D.A., Horvath, P., Moineau, S. (2008). Phage response to CRISPR-encoded resistance in Streptococcus thermophilus. J Bacteriol, 190, 1390-1400. [CrossRef] [PubMed] [Google Scholar]
- Díez-Villaseñor, C., Guzmán, N.M., Almendros, C., García-Martínez J., Mojica, F.J.M. (2013). CRISPR-spacer integration reporter plasmids reveal distinct genuine acquisition specificities among CRISPR-Cas I-E variants of Escherichia coli. RNA Biol, 10, 792-802. [CrossRef] [Google Scholar]
- Dong, D., Guo, M., Wang, S., Zhu, Y., Wang, S., Xiong, Z., Yang, J., Xu, Z., Huang, Z. (2017). Structural basis of CRISPR SpyCas9 inhibition by an anti-CRISPR protein. Nature, 546, 436-439. [CrossRef] [PubMed] [Google Scholar]
- Drake, J.W. (1991). A constant rate of spontaneous mutation in DNA-based microbes. Proc Natl Acad Sci USA, 88, 7160-7164. [CrossRef] [Google Scholar]
- Dupuis, M.E., Villion, M., Magadán, A.H., Moineau, S. (2013). CRISPR-Cas and restriction-modification systems are compatible and increase phage resistance. Nat Commun, 4, 2087. [CrossRef] [PubMed] [Google Scholar]
- Fineran, P.C., Gerritzen, M.J.H., Suárez-Diez, M., Künne, T., Boekhorst, J., van Hijum, S.A., Staals, R.H., Brouns S.J. (2014). Degenerate target sites mediate rapid primed CRISPR adaptation. Proc Natl Acad Sci USA, 111, E1629-1638. [CrossRef] [Google Scholar]
- Godde, J.S, Bickerton, A. (2006). The repetitive DNA elements called CRISPRs and their associated genes: Evidence of horizontal transfer among Prokaryotes. J Mol Evol, 62, 718-729. [CrossRef] [PubMed] [Google Scholar]
- Guo, P., Cheng, Q., Xie, P., Fan, Y., Jiang, W., Qin, Z. (2011). Characterization of the multiple CRISPR loci on Streptomyces linear plasmid pSHK1. Acta Biochim Biophys Sin, 43, 630-639. [CrossRef] [Google Scholar]
- Heidelberg, J.F., Nelson, W.C., Schoenfeld, T., Bhaya, D. (2009). Germ warfare in a microbial mat community: CRISPRs provide insights into the co-evolution of host and viral genomes. PLoS One 4, e4169. [CrossRef] [PubMed] [Google Scholar]
- Horn, H., Slaby, B., Jahn, M., Bayer, K., Moitinho-Silva, L., Förster, F., Abdelmohsen, U.R., Hentschel, U. (2016). An enrichment of CRISPR and other defense-related features in marine sponge-associated microbial metagenomes. Front Microbiol, 7, 1751. [PubMed] [Google Scholar]
- Hynes, A.P., Rousseau, G.M., Lemay, M.L., Horvath, P., Romero, D.A., Frémaux, C., Moineau, S. (2017). An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus Pyogenes Cas9. Nat Microbiol, 2, 1374-1380. [CrossRef] [PubMed] [Google Scholar]
- Iranzo, J., Lobkovsky, A.E., Wolf, Y.I., Koonin, E.V. (2013). Evolutionary dynamics of the prokaryotic adaptive immunity system CRISPR-Cas in an explicit ecological context. J Bacteriol, 195, 3834-3844. [CrossRef] [PubMed] [Google Scholar]
- Jiang, W., Maniv, I., Arain, F., Wang, Y., Levin, B.R., Marraffini, L.A. (2013). Dealing with the evolutionary downside of CRISPR immunity: Bacteria and beneficial plasmids. PLoS Genet, 9, e1003844. [CrossRef] [PubMed] [Google Scholar]
- Kapitonov, V.V., Makarova, K.S., Koonin, E.V. (2015). ISC, a novel group of bacterial and archaeal DNA transposons that encode Cas9 homologs. J Bacteriol, 198, 797-807. [CrossRef] [PubMed] [Google Scholar]
- Koonin, E.V., Krupovic, M. (2014). Evolution of adaptive immunity from transposable elements combined with innate immune systems. Nat Rev Genet, 16, 184-192. [CrossRef] [PubMed] [Google Scholar]
- Koonin, E.V., Makarova, K.S. (2017). Mobile genetic elements and evolution of CRISPR-Cas systems: All the way there and back. Genome Biol Evol, 9, 28-25. [CrossRef] [Google Scholar]
- Krupovic, M., Makarova, K.S., Forterre, P., Prangishvili, D., Koonin, E.V. (2014). Casposons: A new superfamily of self-synthesizing DNA transposons at the origin of prokaryotic CRISPR-Cas immunity. BMC Biol, 12, 36. [CrossRef] [PubMed] [Google Scholar]
- Levy, A., Goren, M.G., Yosef, I., Auster, O., Manor, M., Amitai, G., Edgar, R., Qimron, U., Sorek, R. (2015). CRISPR adaptation biases explain preference for acquisition of foreign DNA. Nature, 520, 505-510. [CrossRef] [PubMed] [Google Scholar]
- Lin, T.L., Pan Y.J., Hsieh, P.F., Hsu, C.R., Wu, M.C., Wang, J.T. (2016). Imipenem represses CRISPR-Cas interference of DNA acquisition through H-NS stimulation in Klebsiella pneumoniae. Sci Rep, 6, 31644. [CrossRef] [PubMed] [Google Scholar]
- Lopatina, A., Medvedeva, S., Shmakov, S., Logacheva, M.D., Krylenkov, V., Severinov, K. (2016). Metagenomic analysis of bacterial communities of antarctic surface snow. Front Microbiol, 7, 1-13. [Google Scholar]
- Louwen, R., Horst-Kreft, D., De Boer, A.G., Van Der Graaf, L., De Knegt, G., Hamersma, M., Heikema, A.P., Timms, A.R., Jacobs, B.C., Wagenaar, J.A., Endtz, H.P., van der Oost, J., Wells J.M., Nieuwenhuis E.E., van Vliet A.H., Willemsen P.T., van Baarlen P., van Belkum, A. (2013). A novel link between Campylobacter jejuni bacteriophage defence, virulence and Guillain-Barré syndrome. Eur J Clin Microbiol Infect Dis, 32, 207-226. [CrossRef] [PubMed] [Google Scholar]
- Mai, G., Ge, R., Sun, G., Meng, Q., Zhou, F. (2016). A comprehensive curation shows the dynamic evolutionary patterns of prokaryotic CRISPRs. BioMed Res Int. doi: 10.1155/2016/7237053. [Google Scholar]
- Makarova, K.S., Grishin, N.V., Shabalina, S.A., Wolf, Y.I., Koonin, E.V. (2006). A putative RNA-interference-based immune system in Prokaryotes: Computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. Biology Direct, 16, 1-7. [Google Scholar]
- Makarova, K.S., Wolf, Y.I., Koonin, E.V. (2013). The basic building blocks and evolution of CRISPR-Cas systems. Biochem Soc Trans, 41, 1392-1400. [CrossRef] [PubMed] [Google Scholar]
- Makarova, K.S., Wolf, Y.I., Alkhnbashi, O.S., Costa, F., Shah, S.A., Saunders, S.J., Barrangou, R., Brouns, S.J., Charpentier, E., Haft, D.H., Horvath, P., Moineau, S., Mojica, F.J., Terns, R.M. Terns, M.P., White, M.F., Yakunin, A.F., Garrett, R.A., van der Oost, J., Backofen, R., Koonin, E.V. (2015). An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol 13, 722-736. [CrossRef] [PubMed] [Google Scholar]
- Marraffini, L.A. (2015). CRISPR-Cas immunity in Prokaryotes. Nature, 526, 55-61. [CrossRef] [PubMed] [Google Scholar]
- Marraffini, L.A., Sontheimer, E.J. (2008). CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA. Science, 322, 1843-1845. [CrossRef] [Google Scholar]
- Medina-Aparicio, L., Rebollar-Flores J.E., Gallego-Hernández, A.L., Vázquez, A., Olvera, L., Gutiérrez-Ríos, R.M., Calva, E., Hernández-Lucas, I. (2011). The CRISPR/Cas immune system is an operon regulated by LeuO, H-NS, and leucine-responsive regulatory rrotein in Salmonella enterica serovar Typhi. J Bacteriol, 193, 2396-2407. [CrossRef] [PubMed] [Google Scholar]
- Millen, A.M., Horvath, P., Boyaval, P., Romero, D.A. (2012). Mobile CRISPR/Cas-mediated bacteriophage resistance in Lactococcus lactis. PLoS One, 7, e51663. [CrossRef] [PubMed] [Google Scholar]
- Mohanraju, P., Makarova, K.S., Zetsche, B., Zhang, F., Koonin, E.V., van der Oost J. (2016). Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science, 353, aad5147. [CrossRef] [Google Scholar]
- Mojica, F.J.M., Díez-Villaseñor, C., García-Martínez, J., Soria, E. (2005). Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol, 60, 174-182. [CrossRef] [PubMed] [Google Scholar]
- Oliveira, P.H., Touchon, M., Rocha E.P.C. (2016). Regulation of genetic flux between bacteria by restriction modification systems. Proc Natl Acad Sci USA, 113, 5658-5663. [CrossRef] [Google Scholar]
- Paez-Espino, D., Sharon, I., Morovic, W., Stahl, B., Thomas, B.C., Barrangou, R., Banfield, F. (2015). CRISPR immunity drives rapid phage genome evolution in Streptococcus thermophilus. mBio, 6(2), 1-9. [CrossRef] [PubMed] [Google Scholar]
- Patterson, A.G., Yevstigneyeva, M.S., Peter, C. (2017). Regulation of CRISPR-Cas adaptive immune Systems. Curr Opin Microbiol, 37, 1-7. [CrossRef] [PubMed] [Google Scholar]
- Pawluk, A., Bondy-Denomy, J., Cheung, Cheung, V.H.W, Maxwell, K.L., Davidson, A.R. (2014). A new group of phage anti-CRISPR genes inhibits the type I-E CRISPR-Cas system of Pseudomonas aeruginosa. mBio 5(2): 1-7. [CrossRef] [PubMed] [Google Scholar]
- Pawluk, A., Amrani, N., Zhang, Y., Garcia, B., Hidalgo-Reyes, Y., Lee, J., Edraki, A., Shah, M., Sontheimer, E.J., Maxwell, K.L., Davidson, A.R. (2016). Naturally occurring off-switches for CRISPR-Cas9. Cell, 167, 1829-1838. [CrossRef] [PubMed] [Google Scholar]
- Peters, J.E., Makarova, K.S., Shmakov, S., Koonin, E.V. (2017). Recruitment of CRISPR-Cas systems 73by Tn7-like transposons. Proc Natl Acad Sci USA, 114, E7358-E7366. [CrossRef] [Google Scholar]
- Pourcel, C., Salvignol, G., Vergnaud, G. (2005). CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology, 151, 653-663. [CrossRef] [PubMed] [Google Scholar]
- Rauch, B.J., Silvis, M.R., Hultquist, J.F., Waters, C., McGregor, M.J., Krogan, N.J., Bondy-Denomy, J. (2016). Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell, 168, 150-158. [Google Scholar]
- Rho, M., Wu, Y.W., Tang, H., Doak, T.G., Ye, Y. (2012). Diverse CRISPRs evolving in human microbiomes. PLoS Genet, 8, e1002441. [CrossRef] [PubMed] [Google Scholar]
- Scholz, I., Lange, S.J., Hein, S., Hess, W.R., Backofen, R. (2013). CRISPR-Cas systems in the Cyanobacterium synechocystis Sp. PCC6803 exhibit distinct processing pathways involving at least two Cas6 and a Cmr2 protein. PLoS One 8, e56470. [CrossRef] [PubMed] [Google Scholar]
- Seed, K.D., Lazinski, D.W., Calderwood, S.B., Camilli, A. (2013). A bacteriophage encodes its own CRISPR/Cas adaptive response to evade host innate immunity. Nature, 494, 489-491. [CrossRef] [PubMed] [Google Scholar]
- Selle, K., Klaenhammer, T.R., Barrangou, R. (2015). CRISPR-based screening of genomic island excision events in Bacteria. Proc Natl Acad Sci USA, 112, 8076-8081. [CrossRef] [Google Scholar]
- Shin, J., Jiang, F., Liu, J.J., Bray, N.L., Rauch, B.J., Baik, S.H., Nogales, E., Bondy-Denomy, J., Corn, J.E., Doudna, J.A. (2017). Disabling Cas9 by an anti-CRISPR DNA mimic. Sci Adv, 3, e1701620. [CrossRef] [PubMed] [Google Scholar]
- Shmakov, S., Smargon, A., Scott, D., Cox D., Pyzocha, N., Yan, W., Abudayyeh, O.O., Gootenberg, J.S., Makarova, K.S., Wolf, Y.I., Severinov, K., Zhang, F., Koonin, E.V. (2017). Diversity and evolution of class 2 CRISPR Cas systems. Nat Rev Microbiol. doi: 10.1038/nrmicro.2016.184. [Google Scholar]
- Sontheimer, E.J., Davidson A.R. (2017). Inhibition of CRISPR-Cas systems by mobile genetic elements. Curr Opin Microbiol, 37, 120-127. [CrossRef] [PubMed] [Google Scholar]
- Soutourina, O.A., Monot, M., Boudry, P., Saujet, L., Pichon, C., Sismeiro, O. Semenova, E., Severinov, K., Le Bouguenec, C., Coppée, J.Y., Dupuy, B., Martin-Verstraete, I. (2013). Genome-wide identification of regulatory RNAs in the human pathogen Clostridium difficile. PLoS Genet, 9(5), e1003493. [CrossRef] [PubMed] [Google Scholar]
- Stern, A., Keren, L., Wurtzel, O., Amitai, G., Sorek, R. (2010). Self-targeting by CRISPR: Gene regulation or autoimmunity? Trends Genet, 26, 335-340. [CrossRef] [PubMed] [Google Scholar]
- Stern, A., Mick, E., Tirosh, I., Sagy, O., Sorek, R. (2012). CRISPR targeting reveals a reservoir of common phages associated with the human gut microbiome. Genome Res, 22, 1985-1994. [CrossRef] [PubMed] [Google Scholar]
- Sun, C.L., Barrangou, R., Thomas, B.C., Horvath, P., Frémaux, C., Banfield, J.F. (2013). Phage mutations in response to CRISPR diversification in a bacterial population. Environ Microbiol, 15, 463-470. [CrossRef] [PubMed] [Google Scholar]
- Takeuchi, N., Wolf Y.I., Makarova, K.S., Koonin, E.V. (2012). Nature and intensity of selection pressure on CRISPR-associated genes. J Bacteriol, 194, 1216-1225. [CrossRef] [PubMed] [Google Scholar]
- Vale, P.F., Lafforgue, G., Gatchitch, F., Gardan, R., Moineau, S., Gandon, S. (2015). Costs of CRISPR-Cas-mediated resistance in Streptococcus thermophilus. Proc Biol Sci, 282, 1812. [Google Scholar]
- Vercoe, R.B., Chang, J.T., Dy, R.L., Taylor, C., Gristwood, T., Clulow, J.S., Richter, C., Przybilski, R., Pitman, A.R., Fineran, P.C. (2013). Cytotoxic chromosomal targeting by CRISPR/Cas systems can reshape bacterial genomes and expel or remodel pathogenicity islands. PLoS Genet, 9(4): e1003454. [CrossRef] [PubMed] [Google Scholar]
- Wang, J., Ma, J, Zhi, C., Xu, M., Lilan, Y., Min, W., Xinzheng, Z., Yanli, W. (2016). A CRISPR evolutionary arms race: structural insights into viral Anti-CRISPR/Cas responses. Cell Res, 26, 1165-1168. [CrossRef] [PubMed] [Google Scholar]
- Wang, X., Yao D., Xu, J.G., Li, A.R., Xu, J., Fu, P., Zhou, Y., Zhu, Y. (2016). Structural basis of Cas3 inhibition by the bacteriophage protein AcrF3. Nat Struct Mol Biol, 23, 868-871. [CrossRef] [PubMed] [Google Scholar]
- Warren, R.A.J. (1980). Modified bases in bacteriophage DNAs. Ann Rev Microbiol, 34, 137-158. [CrossRef] [Google Scholar]
- Weinberger, A.D., Wolf, Y.I., Lobkovsky, A.E., Gilmore, M.S., Koonin E.V. (2012). Viral diversity threshold for adaptive immunity in Prokaryotes. mBio 3(6): 1-10. [CrossRef] [Google Scholar]
- Westra, E.R., Pul, U., Heidrich, N., Jore, M.M., Lundgren, M., Stratmann, T., Wurm, R., Raine, A., Mescher, M., Van Heereveld, L., Mastop, M., Wagner, E.G., Schnetz, K., van Der Oost, J., Wagner, R., Brouns, S.J. (2010). H-NS-mediated repression of CRISPR-based immunity in Escherichia coli K12 can be relieved by the transcription activator LeuO. Mol Microbiol, 77, 1380-1393. [CrossRef] [PubMed] [Google Scholar]
- Westra, E.R., van Houte, S., Oyesiku-Blakemore, S., Makin, B., Broniewski, J.M., Best, A., Bondy-Denomy, J., Davidson, A., Boots, M., Buckling, A. (2015). Parasite exposure drives selective evolution of constitutive versus inducible defense. Curr Biol, 25, 1043-1049. [CrossRef] [PubMed] [Google Scholar]
- Westra, E.R, Dowling, A.J., Broniewski, J.M., van Houte, S. (2016). Evolution and ecology of CRISPR. Ann Rev Ecol Evol Syst, 47, 307-331. [CrossRef] [Google Scholar]
- Yang, H., Patel, D.J. (2017). Inhibition mechanism of an anti-CRISPR suppressor AcrIIA4 targeting SpyCas9. Mol Cell, 67, 117-127. [CrossRef] [PubMed] [Google Scholar]
Citation de l’article : Bernheim, A. (2017). Incontournables et pourtant rares : les déterminants de la distribution des systèmes CRISPR-Cas dans les génomes bactériens. Biologie Aujourd'hui, 211, 255-264
Liste des figures
 |
Figure 1 Vue d’ensemble du mécanisme CRISPR-Cas. Un locus CRISPR-Cas est organisé autour de deux éléments principaux : un groupe de gènes cas et un CRISPR composé de spacers et de répétitions (en noir). L’immunité CRISPR-Cas fonctionne en deux phases principales : l’adaptation où le complexe de protéines Cas permet l’acquisition de nouveaux spacers et l’immunité ciblée. a. Pendant la phase d’adaptation, les protéines Cas forment un complexe (1) capable de générer un court fragment d’ADN à partir de l’ADN étranger présent dans la cellule (2) qui est ensuite intégré dans le CRISPR comme nouveau spacer (3). b. Pendant la phase d’immunité, le CRISPR est transcrit (4) et scindé en petits ARN (5) qui servent de guides pour la reconnaissance de la cible pour les protéines Cas (6), qui dégradent l’ADN (7). Figure adaptée de Bernheim (2017). |
| Dans le texte | |
 |
Figure 2 La diversité des systèmes CRISPR-Cas. Classification des systèmes CRISPR-Cas. Les systèmes CRISPR-Cas sont divisés en deux classes, six types (I à VI) et 21 sous-types. La classification est basée sur des protéines signatures et l’architecture des loci. Figure adaptée de Bernheim (2017). |
| Dans le texte | |
 |
Figure 3 Les mécanismes de résistance des phages à l’immunité CRISPR-Cas. a. Des modifications du génome telles que des mutations dans la séquence PAM, ou dans les séquences cibles, des réarrangements de génomes ou des modifications covalentes de l’ADN permettent aux phages d’échapper à l’immunité CRISPR-Cas. b. Les protéines anti-CRISPR ou Acr (polygones jaunes ou roses) inhibent les systèmes CRISPR-Cas de manière dépendante du type et même du sous-type, en se liant à différents domaines des protéines Cas impliquées dans la reconnaissance de la cible ou le clivage de l’ADN. Figure adaptée de Bernheim (2017). |
| Dans le texte | |
 |
Figure 4 les inconvénients à coder pour des systèmes CRISPR-Cas. La relativement faible abondance des systèmes CRISPR-Cas peut s’expliquer à la fois par des facteurs écologiques, notamment la diversité et l’abondance des phages, et par des coûts intrinsèques, y compris le coût d’expression, la limitation du transfert horizontal de gènes et les phénomènes d’auto-immunité. Figure adaptée de Bernheim (2017). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.


