| Numéro |
Biologie Aujourd’hui
Volume 215, Numéro 3-4, 2021
|
|
|---|---|---|
| Page(s) | 85 - 94 | |
| DOI | https://doi.org/10.1051/jbio/2021013 | |
| Publié en ligne | 11 mars 2022 | |
Article
Les avancées récentes dans le domaine de la biologie structurale des récepteurs couplés aux protéines G de la classe C : Le récepteur métabotropique du glutamate 5
Recent advances in the structural biology of the class C G protein-coupled receptors: The metabotropic Glutamate receptor 5
1
IGF, Université de Montpellier, CNRS, INSERM,
34094
Montpellier, France
2
MRC Laboratory of Molecular Biology,
Cambridge CB2 0QH, UK
3
National Centre for Biological Sciences TIFR,
GKVK Post, Bellary Road,
Bangalore
560065, India
* Auteur correspondant : guillaume.lebon@igf.cnrs.fr
Reçu :
7
Décembre
2021
La classe C des Récepteurs Couplés aux Protéines G (RCPG) comprend plusieurs membres aux fonctions physiologiques importantes comme par exemple les récepteurs des principaux neurotransmetteurs excitateurs (glutamate) et inhibiteurs (GABA) du système nerveux, les récepteurs des goûts umami et sucré et les récepteurs sensibles au calcium. Ces récepteurs possèdent une architecture moléculaire particulière, caractérisée par la présence d’un large domaine extracellulaire (ECD) relié à un domaine membranaire composé de 7 hélices transmembranaires (7TM). De plus, ils forment tous des dimères obligatoires, la dimérisation étant fondamentale pour leur fonction. La fixation d’agoniste dans l’ECD induit l’activation du récepteur. L’activité des agonistes peut être modulée de manière allostérique par des modulateurs positifs (PAM) ou négatifs (NAM), se liant au domaine 7TM. Il est important de comprendre comment les changements de conformation induits par la liaison des agonistes au sein du domaine extracellulaire sont transmis au domaine transmembranaire mais aussi de comprendre les bases structurales et moléculaires de la régulation allostérique des récepteurs de la classe C. Les progrès récents de la microscopie électronique en conditions cryogéniques (cryoEM) ont permis des avancées sans précédent dans le décryptage des bases structurelles et moléculaires des mécanismes d’activation des RCPG de classe C, et notamment du récepteur métabotropique du glutamate de type 5 (mGlu5). Le glutamate entraîne une fermeture et un changement d’orientation des domaines extracellulaires qui induit un mouvement important entre les sous-unités, rapprochant les 7TM et stabilisant la conformation active du récepteur. La diversité de conformations inactives pour les récepteurs de la classe C était inattendue mais propice à une activation possible par des PAM. Ces derniers stabilisent une conformation active des 7TM, indépendante des changements conformationnels induits par les agonistes, représentant un mode alternatif d’activation des récepteurs mGlu. Nous présentons et discutons ici les caractérisations structurales récentes des récepteurs de classe C, en soulignant les résultats qui rendent cette famille de récepteurs unique. La compréhension de la base structurelle de la signalisation des dimères de mGlu représente une réalisation historique et ouvre la voie à l’analyse de la signalisation des dimères de RCPG en général. Ces analyses structurales devraient également ouvrir de nouvelles voies pour la conception de médicaments ciblant cette famille de récepteurs qui sont aussi des cibles thérapeutiques.
Abstract
Class C GPCRs, that include metabotropic glutamate receptors (mGlu), taste receptors, GABAB receptor and Calcium-sensing receptor, are unusual in terms of their molecular architecture and allosteric regulation. They all form obligatory dimers, dimerization being fundamental for their function. More specifically, the mGlu are activated by the main excitatory neurotransmitter, L-glutamate. mGlu activation by glutamate binding in the venus flytrap domain (VFT) triggers conformational changes that are transmitted, through the Cystein-Rich Domain (CRD), to the conserved fold of 7 transmembrane helices (7TM), that couples to intracellular G protein. mGlu activity can also be allosterically modulated by positive (PAM) or negative (NAM) allosteric modulators binding to the 7TM. Recent progress in cryo-electron microscopy (cryoEM) has allowed unprecedented advances in deciphering the structural and molecular basis of their activation mechanism. The agonist induces a large movement between the subunits, bringing the 7TMs together and stabilizing a 7TM conformation structurally similar to the inactive state. The diversity of inactive conformations for the class C was unexpected but allows PAM stabilising a 7TM active conformation independent of the conformational changes induced by agonists, representing an alternative mode of mGlu activation. Here we present and discuss recent structural characterisation of mGlu receptors, highlighting findings that make the class C of GPCR unique. Understanding the structural basis of mGlu dimer signaling represents a landmark achievement and paves the way for structural investigation of GPCR dimer signaling in general. Structural information will open new avenues for structure-based drug design.
Mots clés : récepteur métabotropique du glutamate / allostérie / cryo-microscopie électronique / neuroscience / récepteurs couplés aux protéines G
Key words: metabotropic glutamate receptors / allostery / cryo-electron microscopy / neuroscience / G protein-coupled receptors
© Société de Biologie, 2022
Le cerveau humain est composé de milliards de neurones interconnectés par des synapses. Les réseaux de communication neuronaux ainsi formés sont fondamentaux pour les fonctions cérébrales telles que les processus d’apprentissage ou la mémoire. Les synapses sont le lieu d’échanges des molécules de la communication entre les neurones, appelées neurotransmetteurs, tels que le glutamate. Le glutamate, libéré dans la synapse, est capté par des récepteurs membranaires qui participent à la modulation de l’activité neuronale. Parmi ceux-ci, les récepteurs couplés aux protéines G (RCPG) de la classe C jouent un rôle clé dans le fonctionnement synaptique (Gregory & Goudet, 2021). Leur dysfonctionnement est associé à plusieurs pathologies du système nerveux central comme l’autisme, les douleurs chroniques, la maladie de Parkinson, la schizophrénie et bien d’autres encore (Gregory & Goudet, 2021). Comprendre leurs mécanismes d’activation à l’échelle atomique est essentiel pour mettre au point de nouveaux médicaments pour traiter ces maladies.
La famille des récepteurs de la classe C se compose de 22 récepteurs différents parmi lesquels les récepteurs métabotropiques du glutamate (mGlu) et les récepteurs de l’acide gamma aminobutyrique de type GABAB. Ces deux derniers types de récepteurs sont des récepteurs clés de la neurotransmission, caractéristiques des synapses excitatrices et inhibitrices respectivement (Pin & Bettler, 2016; Kooistra et al., 2021) (Figure 1). Ces récepteurs forment des homo- ou hétéro-dimères obligatoires indispensables pour leurs fonctions biologiques (El Moustaine et al., 2012). Ils sont composés d’un large domaine extracellulaire bilobé appelé Venus Fly Trap (VFT), dans lequel se fixe le GABA ou le glutamate (Pin & Bettler, 2016). Ces domaines VFT sont de plus responsables de la dimérisation du récepteur via une interface de dimérisation entre les lobes I. Le dimère est aussi stabilisé par la formation d’un pont disulfure entre les deux domaines N-terminaux. Chez les mGlu, le VFT est relié au 7TM par un domaine rigide et riche en cystéines (CRD). Enchâssé dans la membrane, le 7TM est une caractéristique conservée et partagée par tous les RCPG. Ce domaine est responsable du couplage des RCPG avec les effecteurs intracellulaires que sont les protéines G et les β-arrestines. Il est aussi le lieu de liaison de modulateurs allostériques positifs (PAM) qui augmentent la transduction du signal de l’extérieur vers l’intérieur de la cellule, ou négatifs (NAM) qui la diminuent (Gregory & Goudet, 2021).
L’analyse structurale des récepteurs de la classe C a longtemps été limitée au domaine extracellulaire (ECD), et plus particulièrement aux VFT composés des Lobes I et II qui constituent le site de liaison du glutamate (Kunishima et al., 2000 ; Pin & Bettler, 2016). La structure cristallographique de plusieurs VFT a été résolue dans les années 2000, démontrant pour la première fois le changement conformationnel et la fermeture du VFT induits par la fixation du glutamate, mais aussi décrivant l’interface de dimérisation entre les deux Lobes I. La fermeture des VFT est accompagnée par leur rotation l’un par rapport à l’autre, suggérant une réorganisation relative des domaines transmembranaires (Doumazane et al., 2013). La réorganisation de la structure quaternaire du récepteur associée à son activation représente un réel challenge pour une analyse structurale. Une première difficulté réside dans la purification d’un récepteur correctement replié et fonctionnel. Un deuxième challenge est représenté par la multitude de conformations que ces récepteurs peuvent adopter de par leurs propriétés dynamiques. En effet, de manière générale, les RCPG existent à l’équilibre entre leurs états actifs et inactifs (Venkatakrishnan et al., 2013, 2016). La situation est d’autant plus compliquée pour les récepteurs de classe C puisque ces derniers forment des dimères obligatoires, avec une dynamique conformationnelle associée à la structure quaternaire du dimère.
Le développement de méthodologies basées sur l’ingénierie des protéines a permis de modifier les RCPG en introduisant des mutations thermostabilisantes ou en utilisant des protéines de fusion afin de contrôler à la fois la stabilité des récepteurs extraits de la membrane à l’aide de détergents et la flexibilité de leurs domaines transmembranaires (Rosenbaum et al., 2007 ; Tate & Schertler, 2009). Ces approches d’ingénierie des protéines ont été la clé pour résoudre les structures de récepteurs liés à leurs ligands en utilisant la technique de diffraction des rayons X. Nous disposons maintenant de plus de 300 structures cristallographiques de récepteurs de la classe A principalement, mais aussi de récepteurs des classes B et F ainsi que des domaines transmembranaires des récepteurs mGlu1 et mGlu5 de la classe C. Ces structures ont permis de décrire le mode de liaison des ligands à leurs récepteurs ainsi que les changements conformationnels associés (Hauser et al., 2021).
Dans le cas du récepteur mGlu5, les études cristallographiques ont révélé le site de fixation de NAM ainsi que les résidus clés dans la modulation de l’activation des 7TM (Doré et al., 2014). Ainsi la structure du 7TM du récepteur mGlu5 lié au NAM mavoglurant [(3aR,4S,7aR)-4-hydroxy-4-[(3-méthylphényl)éthynyl]octahydro-1H-indole-1-carboxylate] a été établie avec une résolution de 2,6Å, révélant le site de fixation allostérique localisé dans les TM2, TM3, TM6 et TM7 (Doré et al., 2014). Ces travaux ont permis de caractériser le site de liaison pour les NAM et ainsi de faciliter l’optimisation et le développement de nouvelles molécules d’intérêt thérapeutique (Christopher et al., 2019).
Analyse structurale des récepteurs de la classe C par cryo-microscopie électronique
L’avènement de la technique de microscopie en conditions cryogéniques (cryoEM) a permis d’analyser la structure des récepteurs liés à leurs ligands ainsi qu’au sein des complexes avec leurs effecteurs de signalisation intracellulaire tels que les protéines G ou, dans de plus rares cas, les arrestines (García-Nafría & Tate, 2020). Cette méthodologie a aussi été le point de départ pour l’analyse structurale et la compréhension de la structure quaternaire des récepteurs dimériques de la classe C. En effet, la cryoEM a permis d’accéder à la structure de récepteurs de la classe C qui étaient réfractaires à la cristallographie des rayons X (García-Nafría & Tate, 2021). Nous disposons maintenant de plus de cinquante structures de récepteurs de la classe C liés à des activateurs ou à des inhibiteurs mais aussi au sein de complexes avec les protéines G. Ces études par cryoEM des récepteurs mGlu2 (Lin et al., 2021 ; Seven et al., 2021), mGlu4 (Lin et al., 2021), mGlu5 (Koehl et al., 2019 ; Nasrallah et al., 2021), GABAB (Papasergi-Scott et al., 2020 ; Park et al., 2020 ; Shen et al., 2021), et du CaSR (Gao et al., 2021 ; Wen et al., 2021), combinées à des analyses biophysiques et fonctionnelles, ont permis de comprendre les bases structurales et moléculaires de la signalisation des récepteurs de la classe C (Figure 1).
Le récepteur mGlu5 est le premier récepteur pour lequel la structure de la forme dimérique a été résolue (Koehl et al., 2019). En plus de révéler l’organisation moléculaire du dimère mGlu5, les changements conformationnels induits par la fixation de l’agoniste quisqualate dans le VFT ont été identifiés. La fermeture des VFT et leurs rotations relatives induisent le rapprochement des deux CRD qui vont établir des contacts moléculaires et ainsi contribuer à stabiliser la conformation active du récepteur (Koehl et al., 2019). Le CRD établit aussi des contacts moléculaires avec l’ECL2 du 7TM, qui sont importants et nécessaires pour la transmission du signal. Cette interaction entre le CRD et la pointe de l’ECL2 est indispensable à la réorganisation des 7TM dans le dimère. Les TM sont séparés dans la conformation inactive Apo. Lors de l’activation, ils sont soumis à un mouvement de rotation et de translation pour, in fine, être repositionnés à une distance suffisante pour établir des contacts moléculaires au niveau de la partie extracellulaire des hélices 6 (Koehl et al., 2019). Cette interaction est nécessaire à la stabilisation de la conformation active du récepteur. Les structures établies récemment pour les récepteurs mGlu2, mGlu4, GABAB et CaSR décrivent un mouvement similaire (Mao et al., 2020 ; Papasergi-Scott et al., 2020 ; Park et al., 2020 ; Gao et al., 2021 ; Lin et al., 2021 ; Seven et al., 2021 ; Shen et al., 2021 ; Wen et al., 2021).
À l’opposé, l’interaction d’un antagoniste maintient le VFT ouvert et prévient la réorganisation des domaines transmembranaires. Dans la conformation inactive, les 7TM sont physiquement distants (Koehl et al., 2019 ; Nasrallah et al., 2021). L’absence d’interactions moléculaires entre les 7TM est caractéristique mais pas spécifique au récepteur mGlu5 (Du et al., 2021). En effet, alors que leur mécanisme d’activation semble conservé, il existe une diversité de conformations inactives parmi les récepteurs de la classe C. Ainsi, l’absence de contacts moléculaires entre les 7TM est observée pour mGlu5 (Koehl et al., 2019 ; Nasrallah et al., 2021) et mGlu7 (Du et al., 2021) par exemple, alors que pour GABAB (Mao et al., 2020 ; Papasergi-Scott et al., 2020 ; Park et al., 2020) et mGlu2 (Du et al., 2021 ; Lin et al., 2021), les 7TM forment une interface moléculaire qui stabilise le dimère dans sa conformation inactive. Cette diversité de conformations peut significativement impacter le rôle physiologique de chaque récepteur.
Dans ce contexte, nous avons récemment résolu les structures du récepteur humain mGlu5 avec un antagoniste orthostérique LY341494 lié dans les VFT et un NAM, le composé MPEP, lié dans les 7TM (Nasrallah et al., 2021). En parallèle, nous avons aussi résolu la structure du récepteur lié à un agoniste orthostérique, le quisqualate, et à un PAM, le composé VU0424465 (Nasrallah et al., 2021). En combinant des études pharmacologiques et structurales, nous avons analysé l’impact des changements conformationnels induits par la fixation des agonistes et des modulateurs allostériques sur la transduction du signal, permettant ainsi de mieux comprendre les mécanismes d’activation du récepteur mGlu5.
Thermostabilisation du récepteur mGlu5
Nous avons précédemment décrit l’expression et la purification d’une version tronquée (mGlu5-Δ856) du récepteur mGlu5 humain (Nasrallah et al., 2018). Le rendement de ces procédures était faible et surtout, le récepteur était plutôt instable dans le détergent n-Dodécyl-β-D-Maltopyranoside couramment utilisé pour la caractérisation structurale des RCPG. Pour améliorer à la fois le rendement de production de mGlu5 solubilisé et correctement replié, nous avons augmenté la stabilité thermique du récepteur en détergent, aussi appelée thermostabilité. Ainsi, la conformation du récepteur mGlu5 sous sa forme dimérique liée au NAM MPEP a été thermostabilisée en introduisant cinq mutations (T742A5.42, S753A5.53, T777A6.42, I799A7.29, A813L7.43) dans le 7TM du récepteur (Nasrallah et al., 2021) (Figure 2). Les mutants thermostabilisants ont été identifiés en combinant un test de liaison basé sur l’utilisation d’un radioligand ([3H]-MPEP) et la mutagénèse dirigée (Lebon et al., 2011 ; Magnani et al., 2016). Dans le cadre de cette étude, nous avons criblé 288 mutants simples du domaine 7TM de mGlu5. Onze mutants ont d’abord été identifiés et sélectionnés pour leur effet thermostabilisant. Le meilleur mutant simple thermostable A813L (mGlu5-1M) affiche un Tm de 28 °C, comparé au récepteur WT qui a un Tm de 20 °C (Figure 2). A813L a ensuite été combiné avec quatre autres mutants thermostabilisants, T742A5.42 (TM5), S753A5.53 (TM5), T777A6.42 (TM6), et I799A7.29 (TM7) en fonction de leurs propriétés additives, pour donner la construction nommée mGlu5-5M (Figure 2). Cette construction mGlu5-5M présente une stabilité thermique améliorée de ∼ 20 °C dans les détergents couramment utilisés pour les études structurales tels que MNG3 (Tm apparent = 41 °C) (Figure 2) ou DDM lorsqu’elle est liée au [3H]-MPEP (Nasrallah et al., 2021). Le récepteur mGlu5-5M conserve sa pleine capacité à activer la protéine G par rapport au récepteur de type sauvage (WT). Le récepteur mGlu5-5M thermostabilisé a été exprimé et purifié jusqu’à homogénéité. Nous avons observé des particules isolées dans la glace amorphe nécessaire à la réalisation de l’analyse structurale en utilisant la cryoEM (Figure 3).
Le processus d’identification des mutations thermostabilisantes nous a aussi permis d’évaluer l’effet du domaine extracellulaire ECD sur la stabilité du domaine transmembranaire. L’ECD est composé du VFT et du CRD qui interagit avec des résidus de l’ECL2. Cette interaction a été précédemment montrée comme étant importante pour la médiation de la transduction du signal (Koehl et al., 2019). L’interaction CRD-ECL2 pourrait également contribuer à la thermostabilité du récepteur complet. Nous avons mesuré la thermostabilité du 7TM-5M seul (Nasrallah et al., 2021). Cette dernière est fortement altérée par l’absence de l’ECD (Nasrallah et al., 2021). Cette observation conforte la contribution du domaine extracellulaire non seulement pour la médiation de la transduction du signal mais aussi pour le maintien de l’intégrité structurale du domaine 7TM nécessaire à sa fonction.
 |
Figure 2 Thermostabilisation du récepteur mGlu5 dimérique, mGlu5-5M. Cinq mutations ont été combinées pour thermostabiliser la conformation liée au NAM MPEP du dimère du récepteur mGlu5 humain. Le graphique représente les courbes de liaison du 3H-MPEP en fonction de la température pour le récepteur mGlu5 WT ainsi que pour le mutant simple mGlu5-1M, double (mGlu5-2M), triple (mGlu5-3M), quadruple (mGlu5-4M) et quintuple (mGlu5-5M) thermostabilisés du récepteur mGlu5. WT : type « sauvage » du récepteur mGlu5. Figure adaptée d’après Nasrallah et al. (2021). |
 |
Figure 3 Structures cryoEM du récepteur mGlu5 thermostabilisé. (A et E) Microphotographies du récepteur mGlu5 lié à des inhibiteurs ou activateurs respectivement, immobilisé dans la glace amorphe sur des grilles de cryo-microscopie électronique (Barre = 250 Å). (B) Carte de densité cryoEM et modèle (C) du récepteur lié à l’antagoniste orthostérique LY341495 et au NAM MPEP. (D) Densité pour le LY341495 et les résidus environnants dans le VFT. (F) Carte de densité cryoEM et modèle (G) du récepteur lié à l’agoniste orthostérique quisqualate et au PAM VU0424465. (H) Densité pour le quisqualate et les résidus environnants. Un des monomères du récepteur dans les panneaux B et F est coloré en bleu et l’autre en gris. Les ligands orthostériques sont représentés en rouge. Dans les modèles C et G, les VFT sont colorés en vert, les CRD en bleu et les 7TM en magenta. Figure adaptée d’après Nasrallah et al. (2021). |
Analyse structurale du récepteur mGlu5 par la technique de cryoEM
Sur la base de la construction décrite ci-dessus, la structure cryoEM du récepteur mGlu5-5M lié à l’antagoniste orthostérique LY341495 et au NAM MPEP a été déterminée à une résolution globale de 4,0 Å (Nasrallah et al., 2021) (Figure 3). Nous avons aussi déterminé la structure du mGlu5-5M lié à l’agoniste quisqualate et à l’ago-PAM de haute affinité VU0424465 à une résolution globale de 3,8 Å (Figure 3). Dans ces deux structures, nous avons obtenu une meilleure résolution pour les VFT que pour les CRD ou encore les 7TM. La résolution plus faible dans les 7TM ne permet pas d’identifier clairement le mode de liaison des modulateurs allostériques (Nasrallah et al., 2021).
Dans le cas de la structure liée à l’antagoniste, nous observons une densité supplémentaire dans chaque VFT, située à la charnière du Lobe I et du Lobe II, qui est modélisée comme le ligand antagoniste LY341495 (Figures 3 et 4) (Nasrallah et al., 2021). Les interactions entre le LY341495 et le récepteur sont médiées à la fois par le Lobe I (70 % des contacts de résidus) et le Lobe II (30 % des contacts de résidus). Ser152 et Thr175 dans le Lobe I établissent des liaisons hydrogène avec les groupes amino- et carboxylique substitués sur le carbone-2 du LY341495. Dans le Lobe II, l’interaction directe du LY341495 avec la chaîne latérale de Tyr223 stabilise une conformation ouverte du Lobe II par rapport au Lobe I qui est encore plus grande que celle observée dans l’état Apo dans laquelle aucun ligand n’est lié dans le VFT (Nasrallah et al., 2021). La liaison simultanée d’un antagoniste dans le site de liaison orthostérique et du MPEP dans les 7TM abolit la signalisation de la protéine G et l’activité basale de mGlu5. Cette structure représente donc la conformation R totalement inactive du récepteur (Figure 3) (Nasrallah et al., 2021).
Lorsque nous avons effectué une classification 3D du récepteur lié à l’agoniste, une sous-population de récepteurs a été trouvée dans l’état inactif, très probablement dû à la perte du quisqualate. En raison de la faible résolution de la structure obtenue, l’analyse de cette classe n’a pas été poursuivie (Nasrallah et al., 2021). Dans l’ensemble, la carte obtenue est similaire à la structure du récepteur mGlu5 lié à un nanobody, au quisqualate et à l’ago-PAM CDPPB (Koehl et al., 2019). Au niveau du site de liaison orthostérique, nous avons constaté que l’agoniste quisqualate interagit avec un ensemble de résidus du Lobe I similaires à ceux intéragissant avec l’antagoniste LY341495, notamment Tyr64, Trp100, Ser151, Ser152, Ser173, Ala174 et Thr175. Cependant, la structure planaire du quisqualate permet la stabilisation de la conformation fermée des VFT alors que le LY341495, une molécule plus grande, empêche la fermeture des VFT, conduisant à un état ouvert stable (Nasrallah et al., 2021).
La résolution des 7TM est plus faible que celle des VFT et le modèle dérivé de la structure aux rayons X à 2,5 Å du domaine 7TM du récepteur mGlu5 lié à un NAM photoswitchable, Alloswitch-1, qui inclut les chaînes latérales, a été placé dans la densité correspondant à l’analyse en cryoEM du récepteur complet et sous sa forme dimérique. En effet, la densité obtenue pour les 7TM ne nous autorisait pas à construire un modèle des 7TM (Nasrallah et al., 2021). La résolution plus faible ne donnait pas la possibilité d’effectuer des comparaisons structurelles approfondies, seule une analyse globale a été réalisée. La superposition des structures liées à l’agoniste et de l’état Apo (Koehl et al., 2019) avec la structure de l’état lié au LY341495 (Nasrallah et al., 2021) illustre les différentes dispositions des 7TM par rapport au mouvement du Lobe II. Le mouvement du Lobe II induit par sa fermeture entraîne le repositionnement du domaine 7TM via les CRD (Figure 4). Les 7TM de l’état lié au LY341495 sont encore plus éloignés que dans l’état Apo (Figure 4). Nous avons ainsi confirmé qu’il n’y a pas de contacts moléculaires entre les 7TM du dimère du récepteur mGlu5 dans la conformation inactive (Figure 4), comme pour l’état Apo précédemment décrit (Koehl et al., 2019). Au contraire, des interfaces moléculaires entre les 7TM sont suggérées pour la conformation inactive de mGlu2 (Xue et al., 2015) et rapportées pour le récepteur GABAB (Mao et al., 2020 ; Papasergi-Scott et al., 2020 ; Park et al., 2020 ; Shaye et al., 2020), ce qui peut expliquer les différences dans les propriétés de signalisation des divers récepteurs de la classe C. Dans son état inactif, chacun des 7TM des protomères du récepteur mGlu5 est une entité isolée.
Dans la conformation intermédiaire active, les VFT, les CRD et les 7TM forment un dimère compact du récepteur mGlu5, qui stabilise l’interface active des 7TM via des contacts moléculaires TM6-TM6 au niveau des résidus Ile7916.56 séparés de seulement 6,7 Å, alors que ces mêmes résidus sont distants de 41,8 Å et 43,3 Å (mesure entre les atomes Cα) dans l’état lié au LY341495 et l’état Apo, respectivement (Nasrallah et al., 2021) (Figure 4). Ce large mouvement des 7TM est médié au travers de l’interaction établie par l’ECL2 du 7TM avec le CRD et avec le linker reliant TM1 au CRD. Cette interaction est critique pour traduire les changements conformationnels des VFT vers les 7TM et permettre ainsi l’activation des protéines G intracellulaires (Koehl et al., 2019).
La préparation protéique de mGlu5-5M lié à un agoniste contient également l’ago-PAM VU0424465, associé aux 7TM. Certaines PAM comme VU0424465, appelées ago-PAM, montrent aussi un effet agoniste tout en se liant aux 7TM, à savoir la capacité d’activer le récepteur sans la présence d’agoniste orthostérique tel que le glutamate. Il existe cependant peu d’informations structurales sur le mode de liaison et d’action des PAM dans le domaine transmembranaire du récepteur mGlu5. Nous avons observé une forte densité adjacente aux résidus Ser8097.39, Trp7856.50 et Pro6553.40 correspondant probablement à l’ago-PAM VU0424465. En effet, ces résidus composent en partie la poche de liaison allostérique où les NAM se lient (Doré et al., 2014 ; Christopher et al., 2019). Cependant, la résolution obtenue est insuffisante pour modéliser et résoudre les détails moléculaires de la liaison des PAM car la position du ligand ne peut être déterminée sans ambiguïté. À la surface de TM1 faisant face aux lipides sur le versant extracellulaire, nous avons aussi identifié une densité supplémentaire qui a été modélisée comme étant une molécule de cholestérol hémisuccinate (CHS), utilisé tout au long de la purification. La molécule de CHS modélisée se positionne comme deux des molécules d’acide oléique modélisées dans la structure aux rayons X du domaine 7TM de mGlu5 (Doré et al., 2014). Le cholestérol est connu pour se lier à la surface des récepteurs exposée aux lipides et ainsi stabiliser le repliement des récepteurs. Cette observation suggère que la rigidité de la molécule de cholestérol peut également contribuer à stabiliser l’interaction entre TM1, 2 et 3. En conclusion, l’absence de la protéine G nécessaire pour stabiliser la conformation active complète (Venkatakrishnan et al., 2013) suggère que cette structure est susceptible de représenter un état actif intermédiaire du récepteur mGlu5.
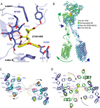 |
Figure 4 Conformation inactive du récepteur mGlu5 lié à l’antagoniste orthostérique LY341495. (A) Site de liaison de l’antagoniste LY341495. (B) Par rapport à l’état apo (cyan ; PDB 6N52), l’agoniste quisqualate induit la fermeture du VFT (vert), tandis que l’antagoniste LY341495 induit une ouverture supplémentaire (bleu). Dans les deux cas, les changements conformationnels sont transmis par les CRD rigides aux 7TM via leur interaction avec la boucle ECL2. (C et D) Repositionnement des 7TM de la conformation liée au LY341495 (C) et au quisqualate (D) par rapport à la conformation apo inactive. Les valeurs représentées (Δ) correspondent à l’augmentation des distances entre les paires d’atomes Cα lors de la transition de l’état inactif à l’état actif. Figure adaptée d’après Nasrallah et al. (2021). |
Agonistes et modulateurs allostériques positifs activent différentes conformations de mGlu5
Enfin, nous nous sommes interrogés sur les possibles conséquences fonctionnelles de l’absence de contact entre les domaines 7TM du récepteur mGlu5 dans sa conformation inactive. En effet, notre étude s’appuyant sur un récepteur mGlu5 thermostabilisé et analysé en détergents confirme les premières observations sur le récepteur mGlu5 reconstitué en nanodisque (Koehl et al., 2019 ; Nasrallah et al., 2021).
Deux interactions moléculaires clés expliquent comment le changement conformationnel du VFT peut conduire à l’activation d’au moins un 7TM. La première est l’interaction entre l’ECL2 et le CRD qui connecte l’ECD à la première hélice transmembranaire du 7TM. L’ECL2 et le CRD forment un pont rigide entre l’ECD et le 7TM. Le changement d’orientation par rapport au plan membranaire du CRD résultant de la réorientation des VFT est important pour la stabilisation de la conformation active. En accord avec cette interaction, la mutation de la région apicale de l’ECL2 affecte largement la capacité des VFT activés à transduire le signal vers les 7TM (Koehl et al., 2019). La deuxième interaction importante est définie par le contact moléculaire entre les TM6 des deux 7TM dans la conformation active du récepteur, permettant la stabilisation d’au moins un 7TM dans son état actif, pour finalement conduire à l’activation de la protéine G. Comme attendu du rôle fonctionnel de cette seconde interaction, Koehl et al. (2019) ont rapporté qu’un récepteur dimérique dans lequel les TM6 sont liées par l’intermédiaire d’un pont disulfure présente une activité constitutive.
Sur cette base, nous avons examiné si le 7TM individuel du dimère du récepteur mGlu5, comme observé dans la conformation inactive du récepteur, peut induire la signalisation de la protéine G lors de l’activation par l’ago-PAM, indépendamment de l’orientation du VFT/CRD par rapport au plan de la membrane et de l’interface du dimère 7TM. Grâce à son activité agoniste intrinsèque, l’ago-PAM VU0424465 peut activer le mGlu5 (Rook et al., 2013). Nous avons démontré que cette réponse ne peut être que partiellement inhibée par des concentrations saturantes de l’antagoniste compétitif LY341495 qui empêche la fermeture du VFT (Nasrallah et al., 2021). Précédemment, il a été rapporté que les VFT ouverts par la liaison d’un antagoniste peuvent toutefois encore se déplacer et adopter une orientation active, comme l’illustre la structure cristalline du dimère VFT mGlu1 avec LY341495 lié (PDB : 3KS9). Nous ne pouvons pas exclure la possibilité que l’ago-PAM VU0424465 induise l’état actif (Kunishima et al., 2000) du dimère VFT. Cependant, cela semble peu probable car le senseur conformationnel du VFT basé sur le signal FRET entre les snap-tags fusionnés à l’extrémité N-terminale et qui permet de suivre les mouvements et l’orientation relative des VFT (Doumazane et al., 2013), n’a pas révélé de changement de conformation inactive vers l’état actif du VFT lors de l’activation par l’ago-PAM VU0424465, en présence de l’antagoniste LY341495 (Nasrallah et al., 2021). Nous avons ensuite confirmé la capacité du récepteur mGlu5 à « signaliser » malgré la conformation R inactive au repos de ses VFT. Pour cela, nous avons mesuré l’activation de la protéine G en utilisant une préparation membranaire obtenue à partir de cellules exprimant le récepteur mGlu5, excluant ainsi la possibilité que le L-glutamate endogène interfère. Comme anticipé, l’ago-PAM VU0424465 déclenche l’activation de la protéine G même si les VFT sont stabilisés à l’état inactif par l’antagoniste LY341495 (Nasrallah et al., 2021). Nos travaux ont donc permis de démontrer que l’ago-PAM VU0424465 peut activer les 7TM même si les VFT sont maintenus dans leur conformation inactive. Cela indique que l’orientation de la liaison rigide CRD-ECL2, nécessaire pour transmettre les changements de conformation des VFT, n’est pas une condition préalable à l’activation des 7TM lors de la stimulation par l’ago-PAM VU0424465. En résumé, l’ago-PAM VU0424465 ne nécessite pas la réorganisation de la structure quaternaire induite par les agonistes orthostériques qui rapproche les 7TM, mais déclenche la transduction du signal à partir du 7TM isolé en l’absence de glutamate (Nasrallah et al., 2021) (Figure 5). Cependant la transduction du signal optimum est obtenue en réponse à la stimulation du récepteur par un agoniste tel que le quisqualate ou le glutamate, qui tous deux vont stabiliser la conformation active du récepteur dimérique.
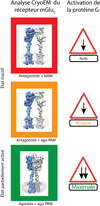 |
Figure 5 Modèle du mécanisme de signalisation des différentes conformations du récepteur mGlu5 pour activer la protéine G. Figure adaptée d’après Nasrallah et al. (2021). |
Mécanisme d’activation des récepteurs de la classe C
Les études récentes sur les structures des récepteurs GABAB (Shen et al., 2021), mGlu2 (Lin et al., 2021 ; Seven et al., 2021) et mGlu4 (Lin et al., 2021), complexés à la protéine G, ont confirmé le mode d’activation asymétrique des récepteurs de la classe C, à savoir qu’un seul protomère peut être lié à une seule protéine G à la fois. De plus, cette asymétrie est illustrée par un seul modulateur allostérique lié dans le 7TM, lui-même couplé à la protéine G. Bien que des changements conformationnels caractéristiques de l’activation soient identifiables, leur amplitude est modeste notamment au niveau de l’hélice 6, dont le mouvement de la partie intracellulaire est caractéristique du mécanisme d’activation des récepteurs de classes A et B. Pour les récepteurs de la classe C, l’hélice 6 est soumise à une légère translation le long de son axe, mais il n’y a pas de large mouvement de son extrémité extracellulaire comme observé pour les récepteurs de classes A et B (Lin et al., 2021 ; Seven et al., 2021). Le peptide C-terminal de la sous-unité alpha de la protéine G vient se loger dans un sillon formé par l’hélice 3 et les boucles intracellulaires 1, 2 et 3 (Lin et al., 2021 ; Seven et al., 2021). Ces différences notables dans le mode de liaison de la protéine G, la conformation active distincte des domaines transmembranaires comparés à la signature moléculaire caractérisée pour les récepteurs des classes A et B ainsi que leur mécanisme d’activation asymétrique font des récepteurs de classe C une famille de récepteurs atypiques. Cela illustre la diversité structurale et fonctionnelle des RCPG. La connaissance des structures d’un nombre croissant de récepteurs de la classe C permettra dans le futur une meilleure compréhension de leurs mécanismes d’activation.
Il aura fallu plus de vingt années pour comprendre les bases structurales de la signalisation des récepteurs de la classe C, depuis la première analyse structurale des VFT isolés jusqu’à l’obtention des premières structures de récepteurs complets et liés à leur protéine G. Ces études décrivent les changements conformationnels nécessaires à la transduction du signal lorsque le glutamate active les récepteurs. Notamment, l’interaction entre la boucle ECL2 et les CRD est critique pour la transmission du signal du VFT vers les 7TM. Aussi, l’interaction entre les domaines 7TM est nécessaire à la stabilisation de l’état actif alors que les mGlu semblent adopter plusieurs conformations, indépendantes d’interactions moléculaires entre les domaines 7TM, à l’état inactif. Cette différence notable est très vraisemblablement source de diversification de la signalisation intracellulaire en conditions physiologiques. Dans le cas du récepteur mGlu5, la conformation active liée à la protéine G, les changements conformationnels associés à l’activation de la protéine G ainsi que le site de liaison des modulateurs allostériques positifs restent indéterminés à ce jour.
Abréviations
7TM : Domaine à 7 hélices transmembranaires
CaSR : Récepteur sensible au calcium
CDPPB : 3-Cyano-N-1,3-diphényl-1H-pyrazol-5-yl benzamide
CHS : Cholestérol hémisuccinate
CRD : Domaine riche en cystéine
cryoEM : Microscopie électronique en conditions cryogéniques
DDM : n-Dodécyl-β-D-Maltopyranoside
ECL2 : Boucle extracellulaire 2
GABAB : Récepteur (type B) de l’acide gamma aminobutyrique
mGlu : Récepteur métabotropique du glutamate
MNG3 : Lauryl maltose-néopentyl glycol
MPEP : 2-Méthyl-6-(phényléthynyl) pyridine
NAM : Modulateur allostérique négatif
PAM : Modulateur allostérique positif
RCPG : Récepteurs Couplés aux Protéines G
VFT : Venus Fly Trap (large domaine extracellulaire bilobé)
Références
- Christopher, J.A., Orgován, Z., Congreve, M., Doré, A.S., Errey, J.C., Marshall, F.H., Mason, J.S., Okrasa, K., Rucktooa, P., Serrano-Vega, M.J., Ferenczy, C.G., Keserù, G.M. (2019). Structure-based optimization strategies for G protein-coupled receptor (GPCR) allosteric modulators: A case study from analyses of new metabotropic glutamate receptor 5 (mGlu5) X-ray structures. J Med Chem, 62, 207-222. [CrossRef] [PubMed] [Google Scholar]
- Doré, A.S., Okrasa, K., Patel, J.C., Serrano-Vega, M., Bennett, K., Cooke, R.M., Errey, J.C., Jazayeri, A., Khan, S., Tehan, B., Weir, M., Wiggin, G.R., Marshall, F.H. (2014). Structure of class C GPCR metabotropic glutamate receptor 5 transmembrane domain. Nature, 511, 557-562. [CrossRef] [PubMed] [Google Scholar]
- Doumazane, E., Scholler, P., Fabre, L., Zwier, J.M., Trinquet, E., Pin, J.-P., Rondard, P. (2013). Illuminating the activation mechanisms and allosteric properties of metabotropic glutamate receptors. Proc Natl Acad Sci USA, 110, E1416-E1425. [CrossRef] [PubMed] [Google Scholar]
- Du, J., Wang, D., Fan, H., Xu, C., Tai, L., Lin, S., Han, S., Tan, Q., Wang, X., Xu, T., Zhang, H., Chu, X., Yi, C., Liu, P., Wang, X., Zhou, Y., Pin, J.P., Rondard, P., Liu, H., Liu, J., Sun, F., Wu, B., Zhao, Q. (2021). Structures of human mGlu2 and mGlu7 homo- and heterodimers. Nature, 594, 589-593. [CrossRef] [PubMed] [Google Scholar]
- El Moustaine, D., Granier, S., Doumazane, E., Scholler, P., Rahmeh, R., Bron, P., Mouillac, B., Banères, J.-L., Rondard, P., Pin, J.-P. (2012). Distinct roles of metabotropic glutamate receptor dimerization in agonist activation and G-protein coupling. Proc Natl Acad Sci USA, 109, 16342-16347. [CrossRef] [PubMed] [Google Scholar]
- Gao, Y., Robertson, M.J., Rahman, S.N., Seven, A.B., Zhang, C., Meyerowitz, J.G., Panova, O., Hannan, F.M., Thakker, R.V., Bräuner-Osborne, H., Mathiesen, J.M., Skiniotis, G. (2021). Asymmetric activation of the calcium-sensing receptor homodimer. Nature, 595, 455-459. [CrossRef] [PubMed] [Google Scholar]
- García-Nafría, J., Tate, C.G. (2020). Cryo-electron microscopy: Moving beyond X-ray crystal structures for drug receptors and drug development. Annu Rev Pharmacol Toxicol, 60, 51-71. [CrossRef] [PubMed] [Google Scholar]
- García-Nafría, J., Tate, C.G. (2021). Structure determination of GPCRs: Cryo-EM compared with X-ray crystallography. Biochem Soc Trans, 49, 2345-2355. [CrossRef] [PubMed] [Google Scholar]
- Gregory, K.J., Goudet, C. (2021). International Union of Basic and Clinical Pharmacology. CXI. Pharmacology, signaling, and physiology of metabotropic glutamate receptors. Pharmacol Rev, 73, 521-569. [CrossRef] [PubMed] [Google Scholar]
- Hauser, A.S., Kooistra, A.J., Munk, C., Heydenreich, F.M., Veprintsev, D.B., Bouvier, M., Babu, M.M., Gloriam, D.E. (2021). GPCR activation mechanisms across classes and macro/microscales. Nat Struct Mol Biol, 28, 879-888. [CrossRef] [PubMed] [Google Scholar]
- Koehl, A., Hu, H., Feng, D., Sun, B., Zhang, Y., Robertson, M.J., Chu, M., Kobilka, T.S., Laeremans, T., Steyaert, J., Tarrasch, J., Dutta, S., Fonseca, R., Weis, W.I., Mathiesen, J.M., Skiniotis, G., Kobilka, B.K. (2019). Structural insights into the activation of metabotropic glutamate receptors. Nature, 566, 79-84. [CrossRef] [PubMed] [Google Scholar]
- Kooistra, A.J., Mordalski, S., Pándy-Szekeres, G., Esguerra, M., Mamyrbekov, A., Munk, C., Keserű, G.M., Gloriam, D.E. (2021). GPCRdb in 2021: Integrating GPCR sequence, structure and function. Nucleic Acids Res, 49, D335- D343. [CrossRef] [PubMed] [Google Scholar]
- Kunishima, N., Shimada, Y., Tsuji, Y., Sato, T., Yamamoto, M., Kumasaka, T., Nakanishi, S., Jingami, H., Morikawa, K. (2000). Structural basis of glutamate recognition by a dimeric metabotropic glutamate receptor. Nature, 407, 971-977. [CrossRef] [PubMed] [Google Scholar]
- Lebon, G., Bennett, K., Jazayeri, A., Tate, C.G. (2011). Thermostabilisation of an agonist-bound conformation of the human adenosine A(2A) receptor. J Mol Biol, 409, 298-310. [CrossRef] [PubMed] [Google Scholar]
- Lin, S., Han, S., Cai, X., Tan, Q., Zhou, K., Wang, D., Wang, X., Du, J., Yi, C., Chu, X., Dai, A., Zhou, Y., Chen, Y., Zhou, Y., Liu, H., Liu, J., Yang, D., Wang, M.W., Zhao, Q., Wu, B. (2021). Structures of Gi-bound metabotropic glutamate receptors mGlu2 and mGlu4. Nature, 594, 583-588. [CrossRef] [PubMed] [Google Scholar]
- Magnani, F., Serrano-Vega, M.J., Shibata, Y., Abdul-Hussein, S., Lebon, G., Miller-Gallacher, J., Singhal, A., Strege, A., Thomas, J.A., Tate, C.G. (2016). A mutagenesis and screening strategy to generate optimally thermostabilized membrane proteins for structural studies. Nat Protoc, 11, 1554-1571. [CrossRef] [PubMed] [Google Scholar]
- Mao, C., Shen, C., Li, C., Shen, D.-D., Xu, C., Zhang, S., Zhou, R., Shen, Q., Chen, L.-N., Jiang, Z., Liu, J., Zhang, Y. (2020). Cryo-EM structures of inactive and active GABAB receptor. Cell Res, 30, 564-573. [CrossRef] [PubMed] [Google Scholar]
- Nasrallah, C., Rottier, K., Marcellin, R., Compan, V., Font, J., Llebaria, A., Pin, J.-P., Banères, J.-L., Lebon, G. (2018). Direct coupling of detergent purified human mGlu5 receptor to the heterotrimeric G proteins Gq and Gs. Sci Rep, 8, 4407. [CrossRef] [PubMed] [Google Scholar]
- Nasrallah, C., Cannone, G., Briot, J., Rottier, K., Berizzi, A.E., Huang, C.-Y., Quast, R.B., Hoh, F., Banères, J.-L., Malhaire, F., Berto, L., Dumazer, A., Font-Ingles, J., Gómez-Santacana, X., Catena, J., Kniazeff, J., Goudet, C., Llebaria, A., Pin, J.P., Vinothkumar, K.R., Lebon, G. (2021). Agonists and allosteric modulators promote signaling from different metabotropic glutamate receptor 5 conformations. Cell Rep, 36, 109648. [CrossRef] [PubMed] [Google Scholar]
- Papasergi-Scott, M.M., Robertson, M.J., Seven, A.B., Panova, O., Mathiesen, J.M., Skiniotis, G. (2020). Structures of metabotropic GABAB receptor. Nature, 584, 310-314. [CrossRef] [PubMed] [Google Scholar]
- Park, J., Fu, Z., Frangaj, A., Liu, J., Mosyak, L., Shen, T., Slavkovich, V.N., Ray, K.M., Taura, J., Cao, B., Geng, Y., Zuo, H., Kou, Y., Grassucci, R., Chen, S., Liu, Z., Lin, X., Williams, J.P., Rice, W.J., Eng, E.T., Huang, R.K., Soni, R.K., Kloss, B., Yu, Z., Javitch, J.A., Hendrickson, W.A., Slesinger, P.A., Quick, M., Graziano, J., Yu, H., Fiehn, O., Clarke, O.B., Frank, J., Fan, Q.R. (2020). Structure of human GABAB receptor in an inactive state. Nature, 584, 304-309. [CrossRef] [PubMed] [Google Scholar]
- Pin, J.-P., Bettler, B. (2016). Organization and functions of mGlu and GABAB receptor complexes. Nature, 540, 60-68. [CrossRef] [PubMed] [Google Scholar]
- Rook, J.M., Noetzel, M.J., Pouliot, W.A., Bridges, T.M., Vinson, P.N., Cho, H.P., Zhou, Y., Gogliotti, R.D., Manka, J.T., Gregory, K.J., Stauffer, S.R., Dudek, F.E., Xiang, Z., Niswender, C.M., Daniels, J.S., Jones, C.K., Lindsley, C.W., Conn, P.J. (2013). Unique signaling profiles of positive allosteric modulators of metabotropic glutamate receptor subtype 5 determine differences in in vivo activity. Biol Psychiatry, 73, 501-509. [CrossRef] [PubMed] [Google Scholar]
- Rosenbaum, D.M., Cherezov, V., Hanson, M.A., Rasmussen, S.G.F., Thian, F.S., Kobilka, T.S., Choi, H.-J., Yao, X.-J., Weis, W.I., Stevens, R.C., Kobilka, B.K. (2007). GPCR engineering yields high-resolution structural insights into beta2-adrenergic receptor function. Science, 318, 1266-1273. [CrossRef] [PubMed] [Google Scholar]
- Seven, A.B., Barros-Álvarez, X., de Lapeyrière, M., Papasergi-Scott, M.M., Robertson, M.J., Zhang, C., Nwokonko, R.M., Gao, Y., Meyerowitz, J.G., Rocher, J.-P., Schelshorn, D., Kobilka, B.K., Mathiesen, J.M., Skiniotis, G. (2021). G-protein activation by a metabotropic glutamate receptor. Nature, 595, 450-454. [CrossRef] [PubMed] [Google Scholar]
- Shaye, H., Ishchenko, A., Lam, J.H., Han, G.W., Xue, L., Rondard, P., Pin, J.-P., Katritch, V., Gati, C., Cherezov, V. (2020). Structural basis of the activation of a metabotropic GABA receptor. Nature, 584, 298-303. [CrossRef] [PubMed] [Google Scholar]
- Shen, C., Mao, C., Xu, C., Jin, N., Zhang, H., Shen, D.-D., Shen, Q., Wang, X., Hou, T., Chen, Z., Rondard, P., Pin, J.P., Zhang, Y., Liu, J. (2021). Structural basis of GABAB receptor-Gi protein coupling. Nature, 594, 594-598. [CrossRef] [PubMed] [Google Scholar]
- Tate, C.G., Schertler, G.F.X. (2009). Engineering G protein-coupled receptors to facilitate their structure determination. Curr Opin Struct Biol, 19, 386-395. [CrossRef] [PubMed] [Google Scholar]
- Venkatakrishnan, A.J., Deupi, X., Lebon, G., Tate, C.G., Schertler, G.F., Babu, M.M. (2013). Molecular signatures of G-protein-coupled receptors. Nature, 494, 185-194. [CrossRef] [PubMed] [Google Scholar]
- Venkatakrishnan, A.J., Deupi, X., Lebon, G., Heydenreich, F.M., Flock, T., Miljus, T., Balaji, S., Bouvier, M., Veprintsev, D.B., Tate, C.G., Schertler, G.F., Babu, M.M. (2016). Diverse activation pathways in class A GPCRs converge near the G-protein-coupling region. Nature, 536, 484-487. [CrossRef] [PubMed] [Google Scholar]
- Wen, T., Wang, Z., Chen, X., Ren, Y., Lu, X., Xing, Y., Lu, J., Chang, S., Zhang, X., Shen, Y., Yang, X. (2021). Structural basis for activation and allosteric modulation of full-length calcium-sensing receptor. Sci Adv, 7, eabg1483. [Google Scholar]
- Xue, L., Rovira, X., Scholler, P., Zhao, H., Liu, J., Pin, J.-P., Rondard, P. (2015). Major ligand-induced rearrangement of the heptahelical domain interface in a GPCR dimer. Nat Chem Biol, 11, 134-140. [CrossRef] [PubMed] [Google Scholar]
Citation de l’article: Berto, L., Dumazer, A., Malhaire, F., Cannone, G., Ragunath, V.K., Goudet, C., et Lebon, G. (2021). Les avancées récentes dans le domaine de la biologie structurale des récepteurs couplés aux protéines G de la classe C : Le récepteur métabotropique du glutamate 5. Biologie Aujourd’hui, 215, 85-94
Liste des figures
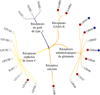 |
Figure 1 Les récepteurs couplés aux protéines G de la classe C. Les points rouges représentent les récepteurs pour lesquels les structures de leurs formes dimériques ont été résolues. Les points bleus représentent les récepteurs pour lesquels la structure du récepteur en complexe avec les protéines G hétérométriques a été établie. Adapté de GPCR db, https://gpcrdb.org/. |
| Dans le texte | |
 |
Figure 2 Thermostabilisation du récepteur mGlu5 dimérique, mGlu5-5M. Cinq mutations ont été combinées pour thermostabiliser la conformation liée au NAM MPEP du dimère du récepteur mGlu5 humain. Le graphique représente les courbes de liaison du 3H-MPEP en fonction de la température pour le récepteur mGlu5 WT ainsi que pour le mutant simple mGlu5-1M, double (mGlu5-2M), triple (mGlu5-3M), quadruple (mGlu5-4M) et quintuple (mGlu5-5M) thermostabilisés du récepteur mGlu5. WT : type « sauvage » du récepteur mGlu5. Figure adaptée d’après Nasrallah et al. (2021). |
| Dans le texte | |
 |
Figure 3 Structures cryoEM du récepteur mGlu5 thermostabilisé. (A et E) Microphotographies du récepteur mGlu5 lié à des inhibiteurs ou activateurs respectivement, immobilisé dans la glace amorphe sur des grilles de cryo-microscopie électronique (Barre = 250 Å). (B) Carte de densité cryoEM et modèle (C) du récepteur lié à l’antagoniste orthostérique LY341495 et au NAM MPEP. (D) Densité pour le LY341495 et les résidus environnants dans le VFT. (F) Carte de densité cryoEM et modèle (G) du récepteur lié à l’agoniste orthostérique quisqualate et au PAM VU0424465. (H) Densité pour le quisqualate et les résidus environnants. Un des monomères du récepteur dans les panneaux B et F est coloré en bleu et l’autre en gris. Les ligands orthostériques sont représentés en rouge. Dans les modèles C et G, les VFT sont colorés en vert, les CRD en bleu et les 7TM en magenta. Figure adaptée d’après Nasrallah et al. (2021). |
| Dans le texte | |
 |
Figure 4 Conformation inactive du récepteur mGlu5 lié à l’antagoniste orthostérique LY341495. (A) Site de liaison de l’antagoniste LY341495. (B) Par rapport à l’état apo (cyan ; PDB 6N52), l’agoniste quisqualate induit la fermeture du VFT (vert), tandis que l’antagoniste LY341495 induit une ouverture supplémentaire (bleu). Dans les deux cas, les changements conformationnels sont transmis par les CRD rigides aux 7TM via leur interaction avec la boucle ECL2. (C et D) Repositionnement des 7TM de la conformation liée au LY341495 (C) et au quisqualate (D) par rapport à la conformation apo inactive. Les valeurs représentées (Δ) correspondent à l’augmentation des distances entre les paires d’atomes Cα lors de la transition de l’état inactif à l’état actif. Figure adaptée d’après Nasrallah et al. (2021). |
| Dans le texte | |
 |
Figure 5 Modèle du mécanisme de signalisation des différentes conformations du récepteur mGlu5 pour activer la protéine G. Figure adaptée d’après Nasrallah et al. (2021). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.


