| Numéro |
Biologie Aujourd’hui
Volume 214, Numéro 3-4, 2020
|
|
|---|---|---|
| Page(s) | 125 - 135 | |
| Section | Centenaire de la Société de Biologie de Strasbourg | |
| DOI | https://doi.org/10.1051/jbio/2020011 | |
| Publié en ligne | 24 décembre 2020 | |
Article
La plasticité développementale chez les plantes : une interaction entre hormones et épigénétique dans les cellules souches méristématiques
Developmental plasticity in plants: an interaction between hormones and epigenetics at the meristem level
Laboratoire de Biologie des Ligneux et des Grandes Cultures (LBLGC), INRAe, Université d’Orléans, EA1207 USC1328,
45067
Orléans, France
* Auteur correspondant : stephane.maury@univ-orleans.fr
Reçu :
16
Septembre
2020
Les plantes sont des organismes fixés dont le développement est continu toute leur vie et qui ont une grande sensibilité aux variations environnementales. Elles réagissent ainsi en manifestant une importante plasticité phénotypique développementale. Cette plasticité est contrôlée pour partie par les (phyto)hormones mais des résultats récents suggèrent également l’implication des mécanismes épigénétiques. Ces deux facteurs interagiraient de manière complexe et notamment dans les cellules souches regroupées au niveau des méristèmes. L’objectif de cette revue est de présenter les arguments actuels concernant cette interaction qui favoriserait la plasticité développementale. Trois points majeurs sont ainsi abordés pour justifier cette interaction entre le contrôle hormonal et l’épigénétique (contrôle au niveau de la chromatine) pour la plasticité développementale des plantes : les arguments en faveur d’un effet des hormones sur la chromatine et vice-versa, les arguments en faveur de leurs rôles sur la plasticité développementale et enfin les arguments en faveur du lieu central de ces interactions, les méristèmes. Diverses perspectives et applications sont discutées.
Abstract
Plants are fixed organisms with continuous development throughout their life and great sensitivity to environmental variations. They react in this way by exhibiting large developmental phenotypic plasticity. This plasticity is partly controlled by (phyto)hormones, but recent studies also suggest the involvement of epigenetic mechanisms. It seems that these two factors may interact in a complex way and especially in the stem cells grouped together in meristems. The objective of this review is to present the current arguments about this interaction which would promote developmental plasticity. Three major points are thus addressed to justify this interaction between hormonal control and epigenetics (control at the chromatin level) for the developmental plasticity of plants: the arguments in favor of an effect of hormones on chromatin and vice versa, the arguments in favor of their roles on developmental plasticity and finally the arguments in favor of the central place of these interactions, the meristems. Various perspectives and applications are discussed.
Mots clés : plantes / épigénétique / plasticité développementale / hormones / méristèmes
Key words: plants / epigenetics / developmental plasticity / hormones / meristems
© Société de Biologie, 2020
Introduction
Parmi les différents règnes du vivant, celui des plantes possède des particularités remarquables concernant leur développement (croissance et morphogenèse). En effet, les plantes, qui sont des organismes fixes, peuvent être annuelles comme la plante modèle Arabidopsis thaliana, bisannuelles comme la betterave sucrière, ou encore pérennes comme les arbres vivant jusqu’à plusieurs milliers d’années. Les plantes ont également un développement dit « continu » c’est-à-dire actif toute leur vie grâce à des zones spécialisées appelées « méristèmes ». Les méristèmes sont des massifs cellulaires totipotents en division active et responsable de la morphogenèse. Les plantes ne possèdent pas de cellules de la lignée germinale « préétablie » comme les animaux, mais celles-ci émergent au cours de la vie de la plante par différenciation de cellules méristématiques. Outre la reproduction sexuée, les plantes peuvent également se multiplier de façon végétative (clonale) en lien direct avec la totipotence de la cellule végétale. Enfin, leur vie fixée est associée à une grande sensibilité vis-à-vis de leur environnement et notamment la lumière, la température, la disponibilité en eau du sol et les micro-organismes pathogènes ou symbiotes. Cette capacité d’acclimatation, quand elle se manifeste par un changement de phénotype, est alors nommée « plasticité phénotypique ». Cela correspond aux différents phénotypes que peut présenter un même génotype (donc sans variation génétique) dans des environnements différents (Pigliucci, 2001 ; West-Eberhard, 2003). La plasticité est dite « stochastique » quand elle répond à une variation environnementale non prévisible par la plante comme une attaque par un agent pathogène. À l’inverse, elle est appelée « prédite » quand elle répond à une variation environnementale anticipée par la plante comme la réponse aux variations saisonnières, telle la floraison (Lachowiec et al., 2016). Enfin, lorsque le changement de phénotype résulte d’une interaction fine entre le développement et l’environnement, on parle alors de « plasticité développementale » (Pigliucci, 1998). À l’inverse, la robustesse est définie comme le maintien du phénotype de la plante dans des environnements contrastés. Ces phénomènes sont contrôlés par des mécanismes moléculaires (Yang & Mackenzie, 2019) permettant notamment un compromis (encore mal connu) pour la plante entre robustesse et plasticité (Lachowiec et al., 2016). Si le rôle des hormones végétales (ou phytohormones) dans ce contrôle du développement et de la réponse à l’environnement est bien documentée depuis des années (Darwin, 1880 ; Gaillochet & Lohmann, 2015), celui de l’épigénétique est bien plus récent (Nicotra et al., 2010 ; Yang & Mackenzie, 2019). Enfin, la signalisation redox en interagissant avec d’autres voies de signalisation, comme les signaux calciques, les cascades de phosphorylation ou de déphosphorylation ou encore avec les voies hormonales, influence également l’expression des gènes et les mécanismes épigénétiques (Figure 1 ; Correia et al., 2016 ; Kumar et al., 2020).
L’objectif de cette revue est de présenter à travers des données récentes l’interconnexion entre le contrôle hormonal et l’épigénétique au niveau des méristèmes qui favoriserait la plasticité développementale (Figure 1). Au-delà de la connaissance des mécanismes fondamentaux, cette question pourrait également être exploitée par l’Homme pour des applications agronomiques ou en biotechnologies végétales.
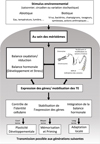 |
Figure 1 La réponse épigénétique aux changements environnementaux au niveau des méristèmes. Les signaux environnementaux sont perçus directement ou indirectement par les méristèmes et pourraient affecter la balance hormonale et l’état redox cellulaire, qui sont alors associés à des changements épigénétiques. Les principaux mécanismes épigénétiques impliqués dans la réponse aux changements environnementaux comprennent le remodelage de la chromatine, la méthylation de l’ADN, les mécanismes des ARN non codants et les modifications et variants d’histones. L’interaction complexe entre les processus de signalisation, l’épigénétique et la génétique se traduit par une variation de l’expression des gènes et/ou la mobilisation d’éléments transposables (TE). Cette interaction participe alors au choix de développement (robustesse vs plasticité) en contrôlant les gènes d’identité cellulaire dans les méristèmes, l’intégration de l’information de la balance hormonale ou encore la stabilisation par la chromatine de l’expression de gènes de réponse aux changements environnementaux. Une réponse physiologique est alors observée ainsi que d’éventuels changements phénotypiques qui permettent aux plantes de s’acclimater aux variations environnementales. La transmission héréditaire de ces changements épigénétiques a été observée dans certains cas chez les plantes (D’après Maury et al., 2019 et Amaral et al., 2020). |
La balance hormonale et l’épigénétique contrôlent le développement des plantes
Les plantes possèdent neuf principales hormones végétales (ou familles de composés) appelées « phytohormones » telles que les auxines, les gibbérellines (GA), les cytokinines, les brassinostéroïdes (BR), les strigolactones, les jasmonates (JA), les salicylates (SA), l’éthylène et l’acide abscissique (ABA). Ces hormones jouent des rôles variés dans la vie des plantes notamment au cours de leur développement (végétatif et reproducteur) et en réponse aux contraintes environnementales (biotiques et abiotiques). Par exemple, la balance entre les auxines, les cytokinines et les GA régule la croissance et la morphogenèse au niveau des méristèmes apicaux caulinaire (MAC) et racinaire. Les JA, SA et l’éthylène sont, en revanche, impliqués dans la réponse aux contraintes biotiques telles que les attaques d’insectes ravageurs ou d’agents pathogènes (champignons, bactéries, virus…).
Ainsi, les hormones végétales agissent en balance et synergie ou antagonisme. Le déterminant majeur de ce contrôle hormonal réside dans un équilibre complexe entre biosynthèse, dégradation, conjugaison, transport, perception et transduction du signal qui peuvent varier d’un organe à l’autre ou bien encore en fonction de l’environnement. C’est notamment au niveau des méristèmes que ce contrôle hormonal s’exerce pour réguler des ensembles de gènes qui déterminent le devenir cellulaire et le développement. En majorité, ces gènes, tels WUSCHEL (WUS) et CLAVATA mais aussi divers gènes homéotiques du célèbre modèle ABCDE de la floraison (AGAMOUS, PISTILLATA, APETALLA, SEPALATTA), codent des facteurs de transcription (FT). Ils permettent l’établissement de gradients moléculaires de différenciation à l’origine de la zonation fonctionnelle dans les méristèmes et au cours de l’organogenèse (Gaillochet & Lohmann, 2015). Par exemple, au sein du MAC, le FT WUS, qui joue un rôle dans le maintien de l’identité des cellules méristématiques (cellules « souches ») (Mayer et al., 1998), est soumis à un contrôle hormonal via les cytokinines (Gaillochet & Lohmann, 2015). À l’inverse, l’émergence d’un primordium foliaire est contrôlée par un transporteur d’efflux d’auxines (PIN1), lui-même régulé par ces mêmes auxines (Blilou et al., 2005). La balance hormonale auxine / cytokinine joue donc un rôle majeur dans le développement des plantes en associant un contrôle hormonal et génétique.
Le contrôle hormonal est également à l’œuvre lors de la plasticité développementale. Par exemple, Rorippa aquatica présente des feuilles de formes différentes selon la température (phénomène appelé « hétérophyllie »). Une modification de la balance hormonale notamment pour l’ABA, l’éthylène et GA a été observée pour une augmentation des températures de 20 à 30 °C (Nakayama et al., 2017). Ces résultats suggèrent que la balance hormonale contrôlerait la forme des feuilles avec des limbes plus ou moins découpés qui s’acclimatent en termes d’évapotranspiration à des températures différentes. Il a été également montré que la méthylation de l’ADN, un mécanisme épigénétique, serait impliqué dans l’hétérophyllie chez Ilex aquifolium (Herrera & Bazaga, 2013).
Ces exemples suggèrent que la plasticité développementale impliquerait une interaction complexe entre la balance hormonale et l’épigénétique. Deux arguments majeurs vont dans ce sens : (i) l’action en balance des hormones avec des synergies et des antagonismes complexes (Robert-Seilaniantz et al., 2007) nécessitant un décodage pour mettre en place l’expression d’un programme génétique « cohérent ». Ce rôle serait joué par la chromatine et les marques épigénétiques ; (ii) le décalage de la cinétique des événements entre le pic hormonal (quelques minutes ou heures) causé par un stress et la manifestation de la plasticité développementale (quelques jours ou semaines). Ce décalage temporel peut devenir important avec notamment la mise en évidence chez les plantes des phénomènes de « vernalisation » (acquisition par une période prolongée au froid en hiver de l’aptitude à fleurir au printemps / été ; voir Baulcombe & Dean, 2014) et de « priming » (stratégie adaptative qui résulte d’une expérience antérieure et améliore la capacité défensive ; Mauch-Mani et al., 2017), voire de transmission dans la descendance d’une plasticité environnementale acquise (Heard & Martienssen, 2014). Tout cela suggère la nécessité d’une mémoire cellulaire, sous le contrôle de processus épigénétiques.
L’épigénétique et la plasticité
L’épigénétique a été définie comme « la branche de la biologie qui étudie les interactions causales entre les gènes et leurs produits qui donnent naissance au phénotype » (Waddington, 1942). Waddington a proposé une image conceptuelle de cette nouvelle discipline avec le « paysage épigénétique », qui représente les interactions entre l’environnement et les gènes conduisant au développement d’un certain phénotype (Waddington, 1940 ; Tronick & Hunter, 2016). La définition de cette nouvelle discipline n’a cessé d’évoluer si bien qu’aujourd’hui c’est un sujet en perpétuel débat (Nicoglou & Merlin, 2017). Ici, nous la définirons donc comme l’étude de tous les processus affectant l’expression des gènes et/ou l’activité des éléments transposables (TE) sans altérer la séquence d’ADN qui peuvent être hérités par mitose (au cours du développement) et/ou par méiose (à travers les générations) (Bossdorf et al., 2007 ; Richards, 2011 ; Ledón-Rettig, 2013). Les TE sont des séquences répétées, présentes dans pratiquement tous les génomes, qui sont capables de se répliquer et de s’insérer dans les chromosomes. Cette activation est sous contrôle épigénétique et peut induire des variations génétiques (Figure 1 ; Marin et al., 2019). L’épigénétique a été proposée comme faisant partie de l’héritabilité manquante puisque l’information codée dans la séquence nucléotidique de l’ADN est souvent insuffisante à elle seule pour expliquer la variation biologique dans toute sa complexité (Maher, 2008 ; Danchin, 2013).
Le principal support moléculaire des mécanismes épigénétiques chez les eucaryotes est la chromatine. Celle-ci est formée par l’association d’ADN enroulé autour de nucléosomes (octamères d’histones). Son compactage se caractérise soit par un état décondensé (chromatine ouverte ou euchromatine) enrichi en gènes et permissif à la transcription, soit par un état condensé (chromatine fermée ou hétérochromatine) enrichi en séquences répétées et en séquences silencieuses. Ces états sont contrôlés par des marques de la chromatine définissant des domaines plus ou moins accessibles aux FT (Bannister & Kouzarides, 2011 ; Roudier et al., 2011) et permettant l’activité de remodelage de la structure de la chromatine (compactée ou décompactée). Ces derniers sont des complexes protéiques modifiant la structure de la chromatine (remodelers) qui permettent alors ou empêchent la transcription des gènes (Becker & Hörz, 2002 ; Harikumar & Meshorer, 2015). Les marques de chromatine ou « marques épigénétiques » (si elles sont transmises par division cellulaire) sont principalement définies par la méthylation des cytosines de l’ADN, les modifications post-traductionnelles d’histones (telles que l’acétylation, la méthylation, la phosphorylation, etc.), les variants d’histones ainsi que les ARN non-codants (ncRNA) (Matzke & Mosher, 2014 ; Jiang & Berger, 2017 ; Zhang et al., 2018 ; Zhao et al., 2019). La compaction de la chromatine, l’expression génique et la mobilisation de TE sont ainsi modulées par des modifications épigénétiques, qui sont déposées par des enzymes spécifiques (appelées writers), puis reconnues par des protéines effectrices (readers) comme les FT. La plupart des marques épigénétiques est également réversibles, et diverses enzymes (erasers) les suppriment (Biswas & Rao, 2017).
Chez les plantes, la méthylation de l’ADN est la marque la plus étudiée car elle est réversible ou transmise lors de la division cellulaire (mitose et méiose) et il est techniquement possible de l’étudier à l’échelle génomique grâce au séquençage à haut-débit (Sow et al., 2018a). La méthylation de l’ADN participe chez les plantes au processus de mémoire cellulaire comme illustré par la vernalisation (Baulcombe & Dean, 2014 ; Hébrard et al., 2016 ; He & Li, 2018). Elle représente aussi une part de l’hérédité car elle est transmise aux générations suivantes (via la méiose) telle que mise en évidence par l’étude d’épivariants naturels (Cubas et al., 1999 ; Manning et al., 2006 ; Miura et al., 2009 ; Lisch, 2012) ou artificiellement induits. C’est par exemple le cas des lignées épigénétiques recombinantes « Epigenetic Recombinant Inbred Lines » (EpiRIL) créées en croisant deux lignées d’Arabidopsis avec des séquences d’ADN similaires mais des profils de méthylation de l’ADN contrastés (Johannes et al., 2009 ; Reinders et al., 2009). Des études sur ces EpiRIL ont montré qu’une partie des variations de méthylation de l’ADN est héritée de manière mendélienne et contribue de façon quantitative à des traits importants pour la plante comme la taille de la racine ou le délai de floraison (Colomé-Tatché et al., 2012 ; Cortijo et al., 2014). De plus, ces EpiRIL montrent une variation phénotypique de leur morphologie ou de leur croissance en réponse à des contraintes biotiques et abiotiques en lien avec la plasticité phénotypique (Latzel et al., 2012 ; Zhang et al., 2013 ; Kooke et al., 2015).
La diversité épigénétique apparaît ainsi comme une nouvelle source de variations phénotypiques qui pourrait favoriser « l’acclimatation » des espèces sauvages aux environnements changeants et être exploitée par les sélectionneurs pour assurer le rendement et la qualité des espèces cultivées dont la diversité génétique a été érodée (Kawakatsu et al., 2016 ; Gallusci et al., 2017 ; Richards et al., 2017 ; Springer & Schmitz, 2017 ; Schmid et al., 2018 ; Marin et al., 2019 ; Herrel et al., 2020). Yona et al. (2015) ont ainsi suggéré que « les organismes s’adaptent en faisant progresser le continuum d’adaptation avec au début des modifications physiologiques et épigénétiques rapides aboutissant ensuite à des variations génétiques (mutations) plus lentes mais durables ». Cela suggère des interactions directes entre l’épigénétique et la génétique (notamment via les TE) mais aussi des interactions entre les réponses physiologiques et l’épigénétique (Alonso et al., 2019 ; Chang et al., 2020). Récemment, il a ainsi été proposé que ces processus impliquent la chromatine et les hormones (Yamamuro et al., 2016 ; Ojolo et al., 2018 ; Yang & Mackenzie, 2019), notamment au niveau des méristèmes, en lien avec la plasticité développementale des plantes (Maury et al., 2019 ; Figure 1).
Les interactions entre le contrôle hormonal et l’épigénétique au niveau des méristèmes : le chaînon manquant pour mieux comprendre la plasticité développementale des plantes ?
Dans la revue de Maury et al. (2019), trois points majeurs sont abordés pour soutenir cette interaction, ou « crosstalk », entre le contrôle hormonal et la chromatine pour la plasticité développementale des plantes : (1) les arguments en faveur d’un effet des hormones sur la chromatine et vice-versa, (2) les arguments en faveur de leurs rôles sur la plasticité développementale ou la robustesse et enfin (3) les arguments en faveur du lieu central de ces interactions, les méristèmes.
(1) Les phytohormones et les marques épigénétiques de la chromatine interagissent à plusieurs niveaux (Maury et al., 2019 ; Figure 1) :
-
Les hormones et leur signalisation affecteraient directement (ou indirectement) l’expression ou l’activité des principaux modificateurs de la chromatine (writers, erasers, readers et remodelers). Ainsi, les composants des voies de signalisation des phytohormones contrôlent directement l’activité des principaux modificateurs de la chromatine tels que les Polycomb Repressive Complex (PRC) 1, avec une activité histone H2A E3 ubiquitine ligase, et PRC2, avec une activité histone H3 lysine27 (H3K27) tri-méthyltransférase, ainsi que d’autres fonctions non-enzymatiques critiques pour le compactage de la chromatine (Yan et al., 2020). PRC1 et 2 jouent un rôle majeur dans la régulation transcriptionnelle au cours du développement des plantes et dans le MAC (Bratzel et al., 2010 ; Chen et al., 2010, 2016 ; Mozgová et al., 2017 ; Yan et al., 2020). Les BR peuvent contrecarrer l’activité H3K27me3 de PRC2 au niveau du répresseur de floraison, Flowering Locus C (FLC), empêchant la transition florale précoce (Yu et al., 2008 ; Li et al., 2018). Enfin, les complexes de chromatine peuvent subir des modifications post-traductionnelles contrôlées par des phytohormones, ce qui influence leur activité. Par exemple, la signalisation de l’ABA induit la phosphorylation de l’ATPase BRAHMA (BRM) modificateur la chromatine (Peirats-Llobet et al., 2016). Cela démontre que l’activité des modificateurs de chromatine peut être modulée par des cascades de signalisation de phytohormones.
-
En réciproque, la chromatine et les marques épigénétiques contrôleraient directement (ou non) l’expression des voies métaboliques, de la conjugaison, du transport, de la perception, de la signalisation des phytohormones ou de leurs gènes cibles. C’est le cas par exemple des variations de la méthylation de l’ADN en réponse à la disponibilité de l’eau chez le peuplier ou les lignées EpiRIL d’Arabidopsis pour lesquelles ont été associés des changements dans les réponses à JA, SA et à l’éthylène (Latzel et al., 2012 ; Lafon-Placette et al., 2018). De même, les plants de riz avec une réduction de la méthylation de H3K27me3 présentent des différences significatives dans la teneur en acide auxine indole-3-acétique, en GA, en ABA, JA et en SA (Liu et al., 2016). Néanmoins, ces effets peuvent être pléiotropes et refléter des états physiologiques généraux modifiés. Ainsi, une preuve plus directe concerne notamment l’activité PRC2 dans le tégument de la graine d’Arabidopsis qui est nécessaire pour réprimer la production de GA avant la fécondation (Figueiredo et al., 2015, 2016 ; Figueiredo & Köhler, 2018).
Ces deux hypothèses ont été confirmées à travers l’étude de différents exemples (Maury et al., 2019), et il est probable que les voies de signalisation des phytohormones et les processus épigénétiques interagissent de manière réciproque à plusieurs niveaux.
(2) Les interconnexions entre la signalisation hormonale et la chromatine participent à la plasticité et à la robustesse. En effet, cette interaction participerait aux processus développementaux de différentes manières (Maury et al., 2019 ; Figure 1) :
-
En contrôlant l’expression de gènes d’identité cellulaire dans les méristèmes. Par exemple, le gène WUS, qui détermine le devenir méristématique d’une cellule, est régulé par la signalisation cytokinine mais aussi par la méthylation de l’ADN, la marque H3K27me et le remodelage de la chromatine (Dodsworth, 2009 ; Cao et al., 2015 ; Liu et al., 2018). Les complexes de modification de la chromatine ciblent également des gènes régulés par des phytohormones qui spécifient l’identité des cellules méristématiques et dont l’expression ectopique peut entraîner une reprogrammation cellulaire (Zuo et al., 2002 ; Galinha et al., 2007). Enfin, la co-expression de FT entraînant une reprogrammation cellulaire peut être déclenchée par des signaux environnementaux et hormonaux ou induite dans des plantes déficientes en certains complexes de la chromatine comme PRC2 (Ikeuchi et al., 2015 ; Mozgová et al., 2017).
-
L’interaction avec la signalisation hormonale interviendrait également en stabilisant l’expression génique au niveau de la chromatine. L’exemple le plus étudié de cette stabilisation dans le temps est l’établissement d’une marque épigénétique, H3K27me3, induite par le froid hivernal sur le facteur de transcription FLC (inhibiteur de la floraison). Ce mécanisme agit pendant la vernalisation et la fixation de cette marque reste stable à travers la mitose ce qui fournit ainsi un système de mémoire de la répression de FLC même lors du retour à une température printanière (Berry et al., 2015 ; Hepworth & Dean, 2015). D’autres marques épigénétiques ou plutôt la persistance de ces marques comme H3K4me2/3, H3/H4ac ou encore l’absence de nucléosome peuvent être retrouvées au niveau des gènes prédisposés aux réponses aux stress biotique et abiotique (Lämke & Bäurle, 2017 ; Bertini et al., 2018 ; Liu et al., 2018), démontrant que la structure de la chromatine contribue également à la mémoire mitotique.
-
Cette interaction, en intégrant les informations des différentes voies de signalisation hormonale au niveau de la chromatine, agit comme un « hub d’intégration ». La modification de la structure de la chromatine induite par les phytohormones peut dépendre de plusieurs modificateurs de chromatine, comme le montrent les modulateurs de la signalisation de l’ABA (Ojolo et al., 2018). Un modificateur de la chromatine unique peut également être impliqué dans les réponses à différentes hormones, comme le montre la BRM (Sarnowska et al., 2016). La chromatine peut ainsi fournir un support robuste intégrant différentes informations tout en potentialisant la persistance des modèles d’expression des gènes pendant les divisions cellulaires mitotiques.
(3) Les méristèmes sont des sites privilégiés des interactions entre hormones et épigénétique. Trois arguments majeurs étayent cette proposition (Maury et al., 2019 ; Figure 1) :
-
Leur rôle central dans la morphogenèse post-embryonnaire, la mémoire et la plasticité. Les méristèmes représentent des sites majeurs de cellules souches chez les plantes (Scheres, 2007 ; Tucker & Laux, 2007 ; Aichinger et al., 2012). Les méristèmes apicaux, ainsi que le méristème secondaire, le cambium, ont la capacité de maintenir et d’auto-renouveler des populations de cellules indifférenciées, permettant le développement continu des organes post-embryonnaires modulé par les conditions environnementales (Gaillochet & Lohmann, 2015). Le MAC est également le lieu de la mémoire épigénétique comme rapporté pour la vernalisation et certains effets de priming (Hepworth & Dean, 2015 ; Lämke & Bäurle, 2017).
-
Leurs particularités pour la signalisation des phytohormones et le remodelage de la chromatine. Les phytohormones et les voies épigénétiques jouent des rôles se chevauchant ou complémentaires dans les fonctions du méristème et la plasticité du développement, posant les bases d’une interaction biologique importante. Les méristèmes se sont également avérés être le lieu du contrôle épigénétique pour la pluripotence, la différenciation et la reprogrammation des cellules souches (Cao et al., 2015 ; Gaillochet & Lohmann, 2015 ; Pi et al., 2015 ; Morao et al., 2016) dont la configuration épigénétique peut différer de celle des tissus environnants (Yadav et al., 2009 ; Baubec et al., 2014). Des preuves majeures de cette interaction phytohormone-chromatine ont été obtenues en utilisant des plants d’Arabidopsis mutants, et en analysant les effets des phytohormones ou des inhibiteurs chimiques de modificateurs de la chromatine sur divers processus de développement (Yamamuro et al., 2016 ; Wong et al., 2017 ; Guo et al., 2018 ; Zheng et al., 2018 ; Genitoni et al., 2020).
-
Les premières études publiées sur cette interaction au niveau du MAC. Par exemple, l’étude de PRC2 réprimant des gènes PIN (transporteurs d’auxines) dans le MAC des mutants d’Arabidopsis clv3, un gène d’identité cellulaire (Lafos et al., 2011). Des études récentes chez la betterave sucrière en état de vernalisation (Hébrard et al., 2016) ou chez le peuplier soumis à la sécheresse ou au froid (Conde et al., 2017 ; Lafon-Placette et al., 2018 ; Le Gac et al., 2018) ont montré que les gènes différentiellement exprimés sous le contrôle de la méthylation de l’ADN dans le MAC correspondent à des réseaux de gènes impliqués dans les voies des phytohormones. Le Gac et al. (2019) ont également pu suivre, au cours de la morphogenèse, la transmission mitotique possible des marques de méthylation du MAC aux jeunes feuilles en formation au cours de la sécheresse. Cette reprogrammation de la méthylation de l’ADN liée aux hormones dans le MAC est également stable plusieurs mois (hiver) après la période de stress estival ce qui suggère l’existence d’une mémoire épigénétique environnementale (Le Gac et al., 2018 ; Sow et al., 2018a). Récemment, l’utilisation de peupliers ddm1 déficients en méthylation de l’ADN (Zhu et al., 2013) a permis de confirmer l’interaction dans le MAC entre la méthylation de l’ADN et les phytohormones cytokinines et SA pour modifier la plasticité développementale de ces arbres en réponse à une sécheresse (Sow et al., 2020).
Conclusions
L’interaction entre les phytohormones et la chromatine permet un contrôle de l’activité méristématique participant à la plasticité développementale des plantes. Néanmoins, la contribution respective de ces deux facteurs et la manière dont ils agissent conjointement ou indépendamment restent encore à préciser (Ojolo et al., 2018 ; Maury et al., 2019). Il est également possible que l’état rédox de la cellule soit un autre élément majeur de cette interaction complexe (Kumar et al., 2020 ; Figure 1). En effet, les espèces réactives de l’oxygène, le monoxyde d’azote et divers antioxydants régulent les activités et l’expression de nombreuses enzymes impliquées dans la méthylation de l’ADN, la méthylation et l’acétylation des histones et le remodelage de la chromatine, contrôlant ainsi la croissance et le développement des plantes en réponse à diverses agressions environnementales (Kumar et al., 2020). L’interaction entre hormones, état redox et mécanismes épigénétiques devra donc faire l’objet d’études plus approfondies chez les plantes et notamment au niveau des méristèmes.
L’interaction entre les phytohormones et la chromatine dans les méristèmes peut intégrer et mémoriser des informations environnementales qui serviront lors d’un futur développement de la plante comme dans le cas du priming (Figure 1). L’effacement de cette signalisation pourrait à l’inverse permettre un ajustement continu de la plante aux nouvelles conditions environnementales. La manière dont l’équilibre entre effacement et mémoire est atteint reste à ce jour énigmatique. La caractérisation moléculaire fine de ces interactions sera certainement plus aisée chez les plantes modèles annuelles comme Arabidopsis, la tomate, le riz ou le maïs mais il est important d’établir aussi des modèles sur des pérennes comme les arbres (exemple des projets ANR EPITREE ANR-17-CE32-0009-01, https://www6.inrae.fr/epitree-project/ et COST Action EPI-CATCH CA19125, https://www.cost.eu/actions/CA19125/) où l’impact de la mémoire épigénétique est important dans le contexte du changement climatique (Sow et al., 2018b ; Amaral et al., 2020).
Il sera également important de quantifier le rôle que jouent les TE dans les changements phénotypiques en réponse à de nouveaux environnements (Marin et al., 2019 ; Figure 1). La régulation des TE étant médiée par des marques épigénétiques, il sera important de dissocier l’épigénétique de la variabilité génétique pour mieux comprendre chaque implication et la causalité des processus. Par exemple, si nous voulons savoir si de nouveaux environnements augmentent la variabilité génétique par la dérégulation épigénétique de l’activité des TE ou exposent simplement la variabilité existante (TE) qui était auparavant masquée (Marin et al., 2019). Le rôle des hormones dans ces mécanismes est encore inconnu. Les méristèmes étant le site de la morphogenèse des organes mais aussi de la production des futures cellules reproductrices (pour le MAC), ces mécanismes joueraient également un rôle dans des phénomènes à plus long terme comme l’adaptation. Au-delà du développement post-embryonnaire assuré par les méristèmes, l’embryon zygotique ou somatique est probablement un autre stade privilégié à étudier pour l’interaction hormones / épigénétique (Maury et al., 2019 ; Wójcikowska et al., 2020). En effet, Wójcikowska et al. (2020) rapportent que la modulation des profils de transcription des gènes pendant l’induction de l’embryogenèse somatique résulte de facteurs épigénétiques qui déterminent également par la suite l’expression de gènes lors du développement embryonnaire. Un exemple concerne des épicéas en Norvège, issus d’embryons somatiques produits à différentes températures, qui ont révélé des différences au niveau de leur phénologie deux ans plus tard, mettant en évidence une mémoire épigénétique acquise au stade embryonnaire (Yakovlev et al., 2011, 2016).
L’exploitation de la variation épigénétique naturelle ou artificiellement induite et le potentiel de modification des plantes à partir de la régénération de méristèmes ou d’embryons somatiques / zygotiques semblent prometteurs pour les sélectionneurs de plantes cultivées (Achour et al., 2017 ; Gallusci et al., 2017 ; Springer & Schmitz, 2017 ; Sow et al., 2018b ; Amaral et al., 2020). À ce sujet, l’édition du génome ou de l’épigénome pour cibler les voies hormonales ou la chromatine semblent également une piste à envisager (Bewg et al., 2018 ; Taagen et al., 2020). Notamment, le potentiel de l’édition d’épigénome pour contrôler la recombinaison pour la sélection végétale est d’un intérêt majeur. Une autre possibilité serait d’exploiter la coordination entre l’épigénétique et le contrôle hormonal pour la production de métabolites spécialisés des plantes comme les alcaloïdes (Dugé de Bernonville et al., 2020). Ces métabolites, indispensables à l’adaptation des plantes à leur environnement, offrent également de nombreuses applications pour l’Homme en pharmacologie et cosmétique. Un enjeu futur serait alors d’améliorer les productions de métabolites secondaires par des biotechnologies végétales notamment in vitro qui cibleraient les marques épigénétiques et les voies hormonales.
Abréviations
EpiRIL : Epigenetic Recombinant Inbred Line
MAC : Méristème Apical Caulinaire
PRC : Polycomb Repressive Complex
Remerciements
Le travail au sein du laboratoire LBLGC est soutenu par des financements récurrents de la part de l’Université d’Orléans et de l’INRAe (département ECODIV). Le projet menant à cette publication a reçu des subventions de l’ANR EPITREE (ANR-17-CE32-0009-01 ; https://www6.inrae.fr/epitree-project/). Nous remercions les GDR 3E (https://rtp-3e.wixsite.com/rt3e), PlasPhen (https://plasticite-phenotypique.cnrs.fr/) et Epiplant (http://www.ibmp.cnrs.fr/creation-dun-groupement-de-recherche-epiplant/) du CNRS ainsi que l’action COST EPI-CATCH (CA19125, https://www.cost.eu/actions/CA19125/) pour leur support et diffusion des travaux de recherche en épigénétique et plasticité phénotypique. Nous remercions J.M. Gibert pour sa relecture et son invitation à la journée « Plasticité Phénotypique » de la Société de Biologie. J.V. perçoit une bourse de thèse du conseil régional Centre Val de Loire. Nous nous excusons auprès de tous les collègues dont le travail n’a pas été cité en raison de limitation de la taille du texte.
Références
- Achour, Z., Archipiano, M., Barneche, F., Baurens, C., Beckert, M. Ben, C. (2017). Epigenetics in plant breeding. Article de positionnement du Groupement d’intérêt scientifique Biotechnologies vertes et de l’Alliance nationale de recherche pour l’environnement. Disponible en ligne à l’adresse http://www.gisbiotechnologiesvertes.com/fr/publications/position-paperepigenetics-in-plant-breeding. [Google Scholar]
- Aichinger, E., Kornet, N., Friedrich, T., Laux, T. (2012). Plant stem cell niches. Annu Rev Plant Biol, 63, 615-636. [CrossRef] [PubMed] [Google Scholar]
- Alonso, C., Ramos-Cruz, D., Becker, C. (2019). The role of plant epigenetics in biotic interactions. New Phytol, 221, 731-737. [CrossRef] [PubMed] [Google Scholar]
- Amaral, J., Ribeyre, Z., Vigneaud, J., Sow, M.D., Fichot, R., Messier, C., Pinto, G., Nolet, P., Maury, S. (2020). Advances and promises of epigenetics for forest trees. Forests, 11, 976. [Google Scholar]
- Bannister, A.J., Kouzarides, T. (2011). Regulation of chromatin by histone modifications. Cell Res, 21, 381-395. [CrossRef] [PubMed] [Google Scholar]
- Baubec, T., Finke, A., Mittelsten Scheid, O., Pecinka, A. (2014). Meristem-specific expression of epigenetic regulators safeguards transposon silencing in Arabidopsis. EMBO Rep, 15, 446-452. [CrossRef] [PubMed] [Google Scholar]
- Baulcombe, D.C., Dean, C. (2014). Epigenetic regulation in plant responses to the environment. Cold Spring Harb Perspect Biol, 6, 19471-19489. [Google Scholar]
- Becker, P.B., Hörz, W. (2002). ATP-dependent nucleosome remodeling. Annu Rev Biochem, 71, 247-273. [CrossRef] [PubMed] [Google Scholar]
- Berry, S., Hartley, M., Olsson, T.S.G., Dean, C., Howard, M. (2015). Local chromatin environment of a Polycomb target gene instructs its own epigenetic inheritance. ELife, 4, 07205. [Google Scholar]
- Bertini, L., Proietti, S., Focaracci, F., Sabatini, B., Caruso, C. (2018). Epigenetic control of defense genes following MeJA-induced priming in rice (O. sativa). J Plant Physiol, 228, 166-177. [CrossRef] [PubMed] [Google Scholar]
- Bewg, W.P., Ci, D., Tsai, C.-J. (2018). Genome editing in trees: from multiple repair pathways to long-term stability. Front Plant Sci, 9, 63-70. [CrossRef] [PubMed] [Google Scholar]
- Biswas, S., Rao, C.M. (2017). Epigenetics in cancer: fundamentals and beyond. Pharmacol Ther, 173, 118-134. [CrossRef] [PubMed] [Google Scholar]
- Blilou, I., Xu, J., Wildwater, M., Willemsen, V., Paponov, I., Friml, J., Heidstra, R., Aida, M., Palme, K., Scheres, B. (2005). The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsis roots. Nature, 433, 39-44. [CrossRef] [PubMed] [Google Scholar]
- Bossdorf, O., Richards, C.L., Pigliucci, M. (2007). Epigenetics for ecologists. Ecol Lett, 11, 106-115. [Google Scholar]
- Bratzel, F., López-Torrejón, G., Koch, M., Del Pozo, J.C., Calonje, M. (2010). Keeping cell identity in Arabidopsis requires PRC1 RING-finger homologs that catalyze H2A monoubiquitination. Curr Biol, 20, 1853-1859. [CrossRef] [PubMed] [Google Scholar]
- Cao, X., He, Z., Guo, L., Liu, X. (2015). Epigenetic mechanisms are critical for the regulation of WUSCHEL expression in floral meristems. Plant Physiol, 168, 1189-1196. [Google Scholar]
- Chang, Y.-N., Zhu, C., Jiang, J., Zhang, H., Zhu, J.-K., Duan, C.-G. (2020). Epigenetic regulation in plant abiotic stress responses. J Integr Plant Biol, 62, 563-580. [Google Scholar]
- Chen, D., Molitor, A., Liu, C., Shen, W.-H. (2010). The Arabidopsis PRC1-like ring-finger proteins are necessary for repression of embryonic traits during vegetative growth. Cell Res, 20, 1332-1344. [CrossRef] [PubMed] [Google Scholar]
- Chen, D., Molitor, A.M., Xu, L., Shen, W.-H. (2016). Arabidopsis PRC1 core component AtRING1 regulates stem cell-determining carpel development mainly through repression of class I KNOX genes. BMC Biol, 14, 112. [CrossRef] [PubMed] [Google Scholar]
- Colomé-Tatché, M., Cortijo, S., Wardenaar, R., Morgado, L., Lahouze, B., Sarazin, A., Etcheverry, M., Martin, A., Feng, S., Duvernois-Berthet, E., Labadie, K., Wincker, P., Jacobsen, S.E., Jansen, R.C., Colot, V., Johannes, F. (2012). Features of the Arabidopsis recombination landscape resulting from the combined loss of sequence variation and DNA methylation. PNAS, 109, 16240-16245. [CrossRef] [Google Scholar]
- Conde, D., Le Gac, A.-L., Perales, M., Dervinis, C., Kirst, M., Maury, S., González-Melendi, P., Allona, I. (2017). Chilling-responsive DEMETER-LIKE DNA demethylase mediates in poplar bud break: role of active DNA demethylase in trees bud break. Plant Cell Environ, 40, 2236-2249. [CrossRef] [PubMed] [Google Scholar]
- Correia, B., Valledor, L., Hancock, R.D., Jesus, C., Amaral, J., Meijón, M., Pinto, G. (2016). Depicting how Eucalyptus globulus survives drought: involvement of redox and DNA methylation events. Funct Plant Biol, 43, 838-850. [CrossRef] [PubMed] [Google Scholar]
- Cortijo, S., Wardenaar, R., Colome-Tatche, M., Gilly, A., Etcheverry, M., Labadie, K., Caillieux, E., Hospital, F., Aury, J.-M., Wincker, P., Roudier, F., Jansen, R.C., Colot, V., Johannes, F. (2014). Mapping the epigenetic basis of complex traits. Science, 343, 1145-1148. [Google Scholar]
- Cubas, P., Vincent, C., Coen, E. (1999). An epigenetic mutation responsible for natural variation in floral symmetry. Nature, 401, 157-161. [CrossRef] [PubMed] [Google Scholar]
- Danchin, E. (2013). Avatars of information: towards an inclusive evolutionary synthesis. Trends Ecol Evol, 28, 351-358. [CrossRef] [PubMed] [Google Scholar]
- Darwin, C., The power of movements in plants, London, John Murray, 1880, 592 p. [Google Scholar]
- Dodsworth, S. (2009). A diverse and intricate signalling network regulates stem cell fate in the shoot apical meristem. Dev Biol, 336, 1-9. [CrossRef] [PubMed] [Google Scholar]
- Dugé de Bernonville, T., Maury, S., Delaunay, A., Daviaud, C., Chaparro, C., Tost, J., O’Connor, S.E., Courdavault, V. (2020). Developmental Methylome of the Medicinal Plant Catharanthus roseus Unravels the tissue-specific control of the monoterpene indole alkaloid pathway by DNA methylation. Int J Mol Sci, 21, 6028. [Google Scholar]
- Figueiredo, D.D., Köhler, C. (2018). Auxin: a molecular trigger of seed development. Genes Dev, 32, 479-490. [CrossRef] [PubMed] [Google Scholar]
- Figueiredo, D.D., Batista, R.A., Roszak, P.J., Köhler, C. (2015). Auxin production couples endosperm development to fertilization. Nat Plants, 1, 15184. [CrossRef] [PubMed] [Google Scholar]
- Figueiredo, D.D., Batista, R.A., Roszak, P.J., Hennig, L., Köhler, C. (2016). Auxin production in the endosperm drives seed coat development in Arabidopsis. ELife, 5, 20542. [Google Scholar]
- Gaillochet, C., Lohmann, J.U. (2015). The never-ending story: from pluripotency to plant developmental plasticity. Development, 142, 2237-2249. [CrossRef] [PubMed] [Google Scholar]
- Galinha, C., Hofhuis, H., Luijten, M., Willemsen, V., Blilou, I., Heidstra, R., Scheres, B. (2007). PLETHORA proteins as dose-dependent master regulators of Arabidopsis root development. Nature, 449, 1053-1057. [CrossRef] [PubMed] [Google Scholar]
- Gallusci, P., Dai, Z., Génard, M., Gauffretau, A., Leblanc-Fournier, N., Richard-Molard, C., Vile, D., Brunel-Muguet, S. (2017). Epigenetics for plant improvement: current knowledge and modeling avenues. Trends Plant Sci, 22, 610-623. [CrossRef] [PubMed] [Google Scholar]
- Genitoni, J., Vassaux, D., Delaunay, A., Citerne, S., Portillo Lemus, L., Etienne, M.-P., Renault, D., Stoeckel, S., Barloy, D., Maury, S. (2020). Hypomethylation of the aquatic invasive plant, Ludwigia grandiflora subsp. hexapetala mimics the adaptive transition into the terrestrial morphotype. Physiol Plant, 170, 280-298. [CrossRef] [PubMed] [Google Scholar]
- Guo, J.-E., Hu, Z., Yu, X., Li, A., Li, F., Wang, Y., Tian, S., Chen, G. (2018). A histone deacetylase gene, SlHDA3, acts as a negative regulator of fruit ripening and carotenoid accumulation. Plant Cell Rep, 37, 125-135. [Google Scholar]
- Harikumar, A., Meshorer, E. (2015). Chromatin remodeling and bivalent histone modifications in embryonic stem cells. EMBO Rep, 16, 1609-1619. [CrossRef] [PubMed] [Google Scholar]
- He, Y., Li, Z. (2018). Epigenetic environmental memories in plants: establishment, maintenance, and reprogramming. Trends Genet, 34, 856-866. [CrossRef] [PubMed] [Google Scholar]
- Heard, E., Martienssen, R.A. (2014). Transgenerational epigenetic inheritance: myths and mechanisms. Cell, 157, 95-109. [CrossRef] [PubMed] [Google Scholar]
- Hébrard, C., Peterson, D.G., Willems, G., Delaunay, A., Jesson, B., Lefèbvre, M., Barnes, S., Maury, S. (2016). Epigenomics and bolting tolerance in sugar beet genotypes. J Exp Bot, 67, 207-225. [CrossRef] [PubMed] [Google Scholar]
- Hepworth, J., Dean, C. (2015). Flowering locus C’s lessons: conserved chromatin switches underpinning developmental timing and adaptation. Plant Physiol, 168, 1237-1245. [Google Scholar]
- Herrel, A., Joly, D., Danchin, E. (2020). Epigenetics in ecology and evolution. Funct Ecol, 34, 381-384. [Google Scholar]
- Herrera, C.M., Bazaga, P. (2013). Epigenetic correlates of plant phenotypic plasticity: DNA methylation differs between prickly and nonprickly leaves in heterophyllous Ilex aquifolium (Aquifoliaceae) trees: Heterophylly and DNA Methylation. Bot J Linnean Soc, 171, 441-452. [CrossRef] [Google Scholar]
- Ikeuchi, M., Iwase, A., Rymen, B., Harashima, H., Shibata, M., Ohnuma, M., Breuer, C., Morao, A.K., de Lucas, M., De Veylder, L., Goodrich, J., Brady, S. M., Roudier, F., Sugimoto, K. (2015). PRC2 represses dedifferentiation of mature somatic cells in Arabidopsis. Nat Plants, 1, 15089. [CrossRef] [PubMed] [Google Scholar]
- Jiang, D., Berger, F. (2017). DNA replication-coupled histone modification maintains Polycomb gene silencing in plants. Science, 357, 1146-1149. [Google Scholar]
- Johannes, F., Porcher, E., Teixeira, F.K., Saliba-Colombani, V., Simon, M., Agier, N., Bulski, A., Albuisson, J., Heredia, F., Audigier, P., Bouchez, D., Dillmann, C., Guerche, P., Hospital, F., Colot, V. (2009). Assessing the impact of transgenerational epigenetic variation on complex traits. PLOS Genet, 5, 1000530. [Google Scholar]
- Kawakatsu, T., Huang, S.C., Jupe, F., Sasaki, E., Schmitz, R.J., Urich, M.A., Castanon, R., Nery, J.R., Barragan, C., He, Y., Chen, H., Dubin, M., Lee, C.-R., Wang, C., Bemm, F., Becker, C., O’Neil, R., O’Malley, R. C., Quarless, D. X., The 1001 Genomes Consortium, Schork, N.J., Weigel, D., Nordborg, M., Ecker, J.R. (2016). Epigenomic diversity in a global collection of Arabidopsis thaliana accessions. Cell, 166, 492-505. [CrossRef] [PubMed] [Google Scholar]
- Kooke, R., Johannes, F., Wardenaar, R., Becker, F., Etcheverry, M., Colot, V., Vreugdenhil, D., Keurentjes, J.J.B. (2015). Epigenetic basis of morphological variation and phenotypic plasticity in Arabidopsis thaliana. Plant Cell, 27, 337-348. [Google Scholar]
- Kumar, S.K., Wang, Y., Zhang, X., Cheng, H., Sun, L., He, S., Hao, F. (2020). Redox components: key regulators of epigenetic modifications in plants. Int J Mol Sci, 21, 1419. [Google Scholar]
- Lachowiec, J., Queitsch, C., Kliebenstein, D.J. (2016). Molecular mechanisms governing differential robustness of development and environmental responses in plants. Ann Bot, 117, 795-809. [CrossRef] [PubMed] [Google Scholar]
- Lafon-Placette, C., Le Gac, A.-L., Chauveau, D., Segura, V., Delaunay, A., Lesage-Descauses, M.-C., Hummel, I., Cohen, D., Jesson, B., Le Thiec, D., Bogeat-Triboulot, M.-B., Brignolas, F., Maury, S. (2018). Changes in the epigenome and transcriptome of the poplar shoot apical meristem in response to water availability affect preferentially hormone pathways. J Exp Bot, 69, 537-551. [CrossRef] [PubMed] [Google Scholar]
- Lafos, M., Kroll, P., Hohenstatt, M.L., Thorpe, F.L., Clarenz, O., Schubert, D. (2011). Dynamic regulation of H3K27 trimethylation during Arabidopsis differentiation. PLOS Genet, 7, 1002040. [Google Scholar]
- Lämke, J., Bäurle, I. (2017). Epigenetic and chromatin-based mechanisms in environmental stress adaptation and stress memory in plants. Genome Biol, 18, 124. [Google Scholar]
- Latzel, V., Zhang, Y., Karlsson Moritz, K., Fischer, M., Bossdorf, O. (2012). Epigenetic variation in plant responses to defence hormones. Ann Bot, 110, 1423-1428. [CrossRef] [PubMed] [Google Scholar]
- Le Gac, A.-L., Lafon-Placette, C., Chauveau, D., Segura, V., Delaunay, A., Fichot, R., Marron, N., Le Jan, I., Berthelot, A., Bodineau, G., Bastien, J.-C., Brignolas, F., Maury, S. (2018). Winter-dormant shoot apical meristem in poplar trees shows environmental epigenetic memory. J Exp Bot, 69, 4821-4837. [CrossRef] [PubMed] [Google Scholar]
- Le Gac, A.-L., Lafon-Placette, C., Delaunay, A., Maury, S. (2019). Developmental, genetic and environmental variations of global DNA methylation in the first leaves emerging from the shoot apical meristem in poplar trees. Plant Signal Behav, 14, 159671. [Google Scholar]
- Ledón-Rettig, C.C. (2013). Ecological epigenetics: an introduction to the symposium. Integr Comp Biol, 53, 307-318. [CrossRef] [PubMed] [Google Scholar]
- Li, Q.-F., Lu, J., Yu, J.-W., Zhang, C.-Q., He, J.-X., Liu, Q.-Q. (2018). The brassinosteroid-regulated transcription factors BZR1/BES1 function as a coordinator in multisignal-regulated plant growth. Biochim Biophys Acta Gene Regul Mech, 1861, 561-571. [CrossRef] [PubMed] [Google Scholar]
- Lisch, D. (2012). How important are transposons for plant evolution? Nat Rev Genet, 14, 49-61. [Google Scholar]
- Liu, H., Lämke, J., Lin, S., Hung, M.-J., Liu, K.-M., Charng, Y., Bäurle, I. (2018). Distinct heat shock factors and chromatin modifications mediate the organ-autonomous transcriptional memory of heat stress. Plant J, 95, 401-413. [CrossRef] [PubMed] [Google Scholar]
- Liu, X., Wei, X., Sheng, Z., Jiao, G., Tang, S., Luo, J., Hu, P. (2016). Polycomb protein OsFIE2 affects plant height and grain yield in rice. PLOS ONE, 11, 0164748. [Google Scholar]
- Maher, B. (2008). Personal genomes: The case of the missing heritability. Nat News, 456, 18-21. [CrossRef] [Google Scholar]
- Manning, K., Tör, M., Poole, M., Hong, Y., Thompson, A.J., King, G.J., Giovannoni, J.J., Seymour, G.B. (2006). A naturally occurring epigenetic mutation in a gene encoding an SBP-circle transcription factor inhibits tomato fruit ripening. Nat Genet, 38, 948-952. [Google Scholar]
- Marin, P., Genitoni, J., Barloy, D., Maury, S., Gibert, P., Ghalambor, C.K., Vieira, C. (2019). Biological invasion: The influence of the hidden side of the (epi)genome. Funct Ecol, 34, 385-400. [Google Scholar]
- Matzke, M.A., Mosher, R.A. (2014). RNA-directed DNA methylation: an epigenetic pathway of increasing complexity. Nat Rev Genet, 15, 394-408. [CrossRef] [PubMed] [Google Scholar]
- Mauch-Mani, B., Baccelli, I., Luna, E., Flors, V. (2017). Defense priming: an adaptive part of induced resistance. Ann Rev Plant Biol, 68, 485-512. [CrossRef] [Google Scholar]
- Maury, S., Sow, M.D., Le Gac, A.-L., Genitoni, J., Lafon-Placette, C., Mozgova, I. (2019). Phytohormone and chromatin crosstalk: the missing link for developmental plasticity? Front Plant Sci, 10, 115-120. [CrossRef] [PubMed] [Google Scholar]
- Mayer, K.F., Schoof, H., Haecker, A., Lenhard, M., Jürgens, G., Laux, T. (1998). Role of WUSCHEL in regulating stem cell fate in the Arabidopsis shoot meristem. Cell, 95, 805-815. [CrossRef] [PubMed] [Google Scholar]
- Miura, K., Okada, Y., Aoi, T., Okada, A., Takahashi, K., Okita, K., Nakagawa, M., Koyanagi, M., Tanabe, K., Ohnuki, M., Ogawa, D., Ikeda, E., Okano, H., Yamanaka, S. (2009). Variation in the safety of induced pluripotent stem cell lines. Nat Biotechnol, 27, 743-745. [CrossRef] [PubMed] [Google Scholar]
- Morao, A.K., Bouyer, D., Roudier, F. (2016). Emerging concepts in chromatin-level regulation of plant cell differentiation: timing, counting, sensing and maintaining. Curr Opin Plant Biol, 34, 27-34. [CrossRef] [PubMed] [Google Scholar]
- Mozgová, I., Muñoz-Viana, R., Hennig, L. (2017). PRC2 Represses Hormone-Induced Somatic Embryogenesis in Vegetative Tissue of Arabidopsis thaliana. PLOS Genet, 13, 1006562. [Google Scholar]
- Nakayama, H., Sinha, N.R., Kimura, S. (2017). How do plants and phytohormones accomplish heterophylly, leaf phenotypic plasticity, in response to environmental cues. Front Plant Sci, 8, 1717. [CrossRef] [PubMed] [Google Scholar]
- Nicoglou, A., Merlin, F. (2017). Epigenetics: A way to bridge the gap between biological fields. Stud Hist Philos Biol Biomed Sci, 66, 73-82. [CrossRef] [PubMed] [Google Scholar]
- Nicotra, A.B., Atkin, O.K., Bonser, S.P., Davidson, A.M., Finnegan, E.J., Mathesius, U., Poot, P., Purugganan, M.D., Richards, C.L., Valladares, F., van Kleunen, M. (2010). Plant phenotypic plasticity in a changing climate. Trends Plant Sci, 15, 684-692. [CrossRef] [PubMed] [Google Scholar]
- Ojolo, S.P., Cao, S., Priyadarshani, S.V.G.N., Li, W., Yan, M., Aslam, M., Zhao, H., Qin, Y. (2018). Regulation of plant growth and development: a review from a chromatin remodeling perspective. Front Plant Sci, 9, 16-28. [CrossRef] [PubMed] [Google Scholar]
- Peirats-Llobet, M., Han, S.-K., Gonzalez-Guzman, M., Jeong, C.W., Rodriguez, L., Belda-Palazon, B., Wagner, D., Rodriguez, P.L. (2016). A direct link between abscisic acid sensing and the chromatin-remodeling ATPase BRAHMA via core ABA signaling pathway components. Mol Plant, 9, 136-147. [CrossRef] [PubMed] [Google Scholar]
- Pi, L., Aichinger, E., van der Graaff, E., Llavata-Peris, C.I., Weijers, D., Hennig, L., Groot, E., Laux, T. (2015). Organizer-derived WOX5 signal maintains root columella stem cells through chromatin-mediated repression of CDF4 expression. Dev Cell, 33, 576-588. [CrossRef] [PubMed] [Google Scholar]
- Pigliucci, M. (1998). Developmental phenotypic plasticity: where internal programming meets the external environment. Curr Opin Plant Biol, 1, 87-91. [CrossRef] [PubMed] [Google Scholar]
- Pigliucci, M., Phenotypic plasticity: beyond nature and nurture, The Johns Hopkins University Press, 2001, 328 p. [Google Scholar]
- Reinders, J., Wulff, B.B.H., Mirouze, M., Marí-Ordóñez, A., Dapp, M., Rozhon, W., Bucher, E., Theiler, G., Paszkowski, J. (2009). Compromised stability of DNA methylation and transposon immobilization in mosaic Arabidopsis epigenomes. Genes Dev, 23, 939-950. [CrossRef] [PubMed] [Google Scholar]
- Richards, E.J. (2011). Natural epigenetic variation in plant species: a view from the field. Curr Opin Plant Biol, 14, 204-209. [CrossRef] [PubMed] [Google Scholar]
- Richards, C.L., Alonso, C., Becker, C., Bossdorf, O., Bucher, E., Colomé-Tatché, M., Durka, W., Engelhardt, J., Gaspar, B., Gogol-Döring, A., Grosse, I., van Gurp, T.P., Heer, K., Kronholm, I., Lampei, C., Latzel, V., Mirouze, M., Opgenoorth, L., Paun, O., Prohaska, S.J., Rensing, S.A., Stadler, P.F., Trucchi, E., Ullrich, K., Verhoeven, K.J.F. (2017). Ecological plant epigenetics: Evidence from model and non-model species, and the way forward. Ecol Lett, 20, 1576-1590. [Google Scholar]
- Robert-Seilaniantz, A., Navarro, L., Bari, R., Jones, J.D.G. (2007). Pathological hormone imbalances. Curr Opin Plant Biol, 10, 372-379. [CrossRef] [PubMed] [Google Scholar]
- Roudier, F., Ahmed, I., Bérard, C., Sarazin, A., Mary-Huard, T., Cortijo, S., Bouyer, D., Caillieux, E., Duvernois-Berthet, E., Al-Shikhley, L., Giraut, L., Després, B., Drevensek, S., Barneche, F., Dèrozier, S., Brunaud, V., Aubourg, S., Schnittger, A., Bowler, C., Martin-Magniette, M.-L., Robin, S., Caboche, M., Colot, V. (2011). Integrative epigenomic mapping defines four main chromatin states in Arabidopsis: Organization of the Arabidopsis epigenome. EMBO J, 30, 1928-1938. [CrossRef] [PubMed] [Google Scholar]
- Sarnowska, E., Gratkowska, D.M., Sacharowski, S.P., Cwiek, P., Tohge, T., Fernie, A.R., Siedlecki, J.A., Koncz, C., Sarnowski, T.J. (2016). The role of SWI/SNF chromatin remodeling complexes in hormone crosstalk. Trends Plant Sci, 21, 594-608. [CrossRef] [PubMed] [Google Scholar]
- Scheres, B. (2007). Stem-cell niches: nursery rhymes across kingdoms. Nat Rev Mol Cell Biol, 8, 345-354. [CrossRef] [PubMed] [Google Scholar]
- Schmid, M.W., Heichinger, C., Coman Schmid, D., Guthörl, D., Gagliardini, V., Bruggmann, R., Aluri, S., Aquino, C., Schmid, B., Turnbull, L.A., Grossniklaus, U. (2018). Contribution of epigenetic variation to adaptation in Arabidopsis. Nat Commun, 9, 4446. [Google Scholar]
- Sow, M.D., Allona, I., Ambroise, C., Conde, D., Fichot, R., Gribkova, S., Jorge, V., Le-Provost, G., Pâques, L., Plomion, C., Salse, J., Sanchez-Rodriguez, L., Segura, V., Tost, J. Maury, S. (2018a). Epigenetics in forest trees: state of the art and potential implications for breeding and management in a context of climate change. Adv Bot Res, 88, 387-453. [Google Scholar]
- Sow, M.D., Segura, V., Chamaillard, S., Jorge, V., Delaunay, A., Lafon-Placette, C., Fichot, R., Faivre-Rampant, P., Villar, M., Brignolas, F., Maury, S. (2018b). Narrow-sense heritability and PST estimates of DNA methylation in three Populus nigra L. populations under contrasting water availability. Tree Genet Genomes, 14, 78. [Google Scholar]
- Sow, M.D., Le Gac, A.-L., Fichot, R., Lanciano, S., Delaunay, A., Le Jan, I., Lesage-Descauses, M.-C., Citerne, S., Caius, J., Brunaud, V., Soubigou-Taconnat, L., Cochard, H., Segura, V., Chaparro, C., Grunau, C., Daviaud, C., Tost, J., Brignolas, F., Strauss, S.H., Mirouze, M. Maury, S. (2020). Hypomethylated poplars show higher tolerance to water deficit and highlight a dual role for DNA methylation in shoot meristem: regulation of stress response and genome integrity. BioRxiv, 045328. [Google Scholar]
- Springer, N.M., Schmitz, R.J. (2017). Exploiting induced and natural epigenetic variation for crop improvement. Nat Rev Genet, 18, 563-575. [CrossRef] [PubMed] [Google Scholar]
- Taagen, E., Bogdanove, A.J., Sorrells, M.E. (2020). Counting on crossovers: controlled recombination for plant breeding. Trends Plant Sci, 25, 455-465. [CrossRef] [PubMed] [Google Scholar]
- Tronick, E., Hunter, R.G. (2016). Waddington, dynamic systems, and epigenetics. Front Behav Neurosci, 10, 107. [CrossRef] [PubMed] [Google Scholar]
- Tucker, M.R., Laux, T. (2007). Connecting the paths in plant stem cell regulation. Trends Cell Biol, 17, 403-410. [Google Scholar]
- Waddington, C.H., Organisers and genes, The Cambridge University Press, Cambridge, UK, 1940, 166 p. [Google Scholar]
- Waddington, C.H. (1942). Canalization of development and the inheritance of acquired characters. Nature, 150, 563-565. [Google Scholar]
- West-Eberhard, M.J., Developmental plasticity and evolution, Oxford University Press, 2003, 814 p. [CrossRef] [Google Scholar]
- Wójcikowska, B., Wójcik, A.M., Gaj, M.D. (2020). Epigenetic regulation of auxin-induced somatic embryogenesis in plants. Int J Mol Sci, 21, 2307. [Google Scholar]
- Wong, M.M., Chong, G.L., Verslues, P.E. (2017). Epigenetics and RNA processing: connections to drought, salt, and ABA? Methods Mol Biol, 1631, 3-21. [CrossRef] [PubMed] [Google Scholar]
- Yadav, R.K., Girke, T., Pasala, S., Xie, M., Reddy, G.V. (2009). Gene expression map of the Arabidopsis shoot apical meristem stem cell niche. PNAS, 106, 4941-4946. [CrossRef] [Google Scholar]
- Yakovlev, I.A., Asante, D.K.A., Fossdal, C.G., Junttila, O., Johnsen, Ø. (2011). Differential gene expression related to an epigenetic memory affecting climatic adaptation in Norway spruce. Plant Sci, 180, 132-139. [CrossRef] [PubMed] [Google Scholar]
- Yakovlev, I.A., Carneros, E., Lee, Y., Olsen, J.E., Fossdal, C.G. (2016). Transcriptional profiling of epigenetic regulators in somatic embryos during temperature induced formation of an epigenetic memory in Norway spruce. Planta, 243, 1237-1249. [CrossRef] [PubMed] [Google Scholar]
- Yamamuro, C., Zhu, J.-K., Yang, Z. (2016). Epigenetic modifications and plant hormone action. Mol Plant, 9, 57-70. [CrossRef] [PubMed] [Google Scholar]
- Yan, B., Lv, Y., Zhao, C., Wang, X. (2020). Knowing when to silence: roles of polycomb-group proteins in SAM maintenance, root development, and developmental phase transition. Int J Mol Sci, 21, 5871. [Google Scholar]
- Yang, X., Mackenzie, S.A. (2019). Many facets of dynamic plasticity in plants. Cold Spring Harb Perspect Biol, 11, 034629. [Google Scholar]
- Yona, A.H., Frumkin, I., Pilpel, Y. (2015). A relay race on the evolutionary adaptation spectrum. Cell, 163, 549-559. [CrossRef] [PubMed] [Google Scholar]
- Yu, X., Li, L., Li, L., Guo, M., Chory, J., Yin, Y. (2008). Modulation of brassinosteroid-regulated gene expression by jumonji domain-containing proteins ELF6 and REF6 in Arabidopsis. PNAS, 105, 7618-7623. [CrossRef] [Google Scholar]
- Zhang, H., Lang, Z., Zhu, J.-K. (2018). Dynamics and function of DNA methylation in plants. Nat Rev Mol Cell Biol, 19, 489-506. [CrossRef] [PubMed] [Google Scholar]
- Zhang, Y.-Y., Fischer, M., Colot, V., Bossdorf, O. (2013). Epigenetic variation creates potential for evolution of plant phenotypic plasticity. New Phytol, 197, 314-322. [CrossRef] [PubMed] [Google Scholar]
- Zhao, H., Winogradoff, D., Dalal, Y., Papoian, G.A. (2019). The Oligomerization Landscape of Histones. Biophys J, 116, 1845-1855. [CrossRef] [PubMed] [Google Scholar]
- Zheng, X., Hou, H., Zhang, H., Yue, M., Hu, Y., Li, L. (2018). Histone acetylation is involved in GA-mediated 45S rDNA decondensation in maize aleurone layers. Plant Cell Rep, 37, 115-123. [Google Scholar]
- Zhu, R., Shevchenko, O., Ma, C., Maury, S., Freitag, M., Strauss, S.H. (2013). Poplars with a PtDDM1-RNAi transgene have reduced DNA methylation and show aberrant post-dormancy morphology. Planta, 237, 1483-1493. [CrossRef] [PubMed] [Google Scholar]
- Zuo, J., Niu, Q.-W., Frugis, G., Chua, N.-H. (2002). The WUSCHEL gene promotes vegetative-to-embryonic transition in Arabidopsis. Plant J, 30, 349-359. [CrossRef] [PubMed] [Google Scholar]
Citation de l’article : Vigneaud, J. et Maury, S. (2020). La plasticité développementale chez les plantes : une interaction entre hormones et épigénétique dans les cellules souches méristématiques. Biologie Aujourd’hui, 214, 125-135
Liste des figures
 |
Figure 1 La réponse épigénétique aux changements environnementaux au niveau des méristèmes. Les signaux environnementaux sont perçus directement ou indirectement par les méristèmes et pourraient affecter la balance hormonale et l’état redox cellulaire, qui sont alors associés à des changements épigénétiques. Les principaux mécanismes épigénétiques impliqués dans la réponse aux changements environnementaux comprennent le remodelage de la chromatine, la méthylation de l’ADN, les mécanismes des ARN non codants et les modifications et variants d’histones. L’interaction complexe entre les processus de signalisation, l’épigénétique et la génétique se traduit par une variation de l’expression des gènes et/ou la mobilisation d’éléments transposables (TE). Cette interaction participe alors au choix de développement (robustesse vs plasticité) en contrôlant les gènes d’identité cellulaire dans les méristèmes, l’intégration de l’information de la balance hormonale ou encore la stabilisation par la chromatine de l’expression de gènes de réponse aux changements environnementaux. Une réponse physiologique est alors observée ainsi que d’éventuels changements phénotypiques qui permettent aux plantes de s’acclimater aux variations environnementales. La transmission héréditaire de ces changements épigénétiques a été observée dans certains cas chez les plantes (D’après Maury et al., 2019 et Amaral et al., 2020). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.


