| Numéro |
Biologie Aujourd’hui
Volume 215, Numéro 1-2, 2021
|
|
|---|---|---|
| Page(s) | 45 - 57 | |
| DOI | https://doi.org/10.1051/jbio/2021006 | |
| Publié en ligne | 16 août 2021 | |
Article
Contrôle des voies métaboliques par les enzymes E3 ligases : une opportunité de ciblage thérapeutique
Metabolic pathways controlled by E3 ligases: an opportunity for therapeutic targeting
Université Clermont Auvergne, INRAE, UNH, Unité de Nutrition Humaine,
63000
Clermont-Ferrand, France
* Auteur correspondant : daniel.taillandier@inrae.fr
Reçu :
4
Juin
2021
Depuis sa découverte, le Système Ubiquitine Protéasome (UPS) est reconnu pour son rôle majeur dans le contrôle de la plupart des voies métaboliques de la cellule. Outre son rôle primordial dans la dégradation des protéines, il intervient aussi dans l’adressage, la signalisation ou la réparation de l’ADN, ce qui en fait un acteur incontournable de l’homéostasie cellulaire. Bien que d’autres systèmes de contrôles existent dans la cellule, l’UPS est souvent considéré comme le chef d’orchestre. Au vu de son importance, toute dérégulation de l’UPS entraîne des désordres plus ou moins sévères pour la cellule et donc l’organisme. De fait, l’UPS est impliqué dans de nombreuses pathologies (cancer, maladie d’Alzheimer, de Huntington, etc.). L’UPS est composé de plus de 1000 protéines différentes dont les combinaisons permettent le ciblage fin de virtuellement toutes les protéines de l’organisme. L’UPS fait appel à une cascade enzymatique (E1, 2 isoformes ; E2 > 35 isoformes ; E3 > 800 isoformes) qui permet le transfert de l’ubiquitine, une petite protéine de 8,5 kDa, sur la protéine à cibler soit pour sa dégradation, soit pour modifier son activité. Ce signal d’ubiquitinylation est réversible et de nombreuses déubiquitinylases (DUB, ∼ 80 isoformes) jouent aussi un rôle important. Les enzymes E3 sont les plus nombreuses et leur fonction est de reconnaître la protéine cible, ce qui en fait des acteurs importants dans la spécificité d’action de l’UPS. La nature même des E3 et la complexité de leurs interactions avec différents partenaires offrent un champ d’investigation très large et donc des potentialités importantes pour le développement d’approches thérapeutiques. Sans être exhaustive, cette revue illustre les différentes stratégies ayant déjà été mises en œuvre pour lutter contre différentes pathologies (à l’exclusion des infections bactériennes ou virales).
Abstract
Since its discovery, the Ubiquitin Proteasome System (UPS) has been recognized for its major role in controlling most of the cell’s metabolic pathways. In addition to its essential role in the degradation of proteins, it is also involved in the addressing, signaling or repair of DNA, which makes it a key player in cellular homeostasis. Although other control systems exist in the cell, the UPS is often referred to as the conductor. In view of its importance, any dysregulation of the UPS leads to more or less severe disorders for the cell and therefore the body, which accounts for UPS implication in many pathologies (cancer, Alzheimer’s disease, Huntington’s disease, etc.). UPS is made up of more than 1000 different proteins, the combinations of which allow the fine targeting of virtually all proteins in the body. UPS uses an enzymatic cascade (E1, 2 members; E2 > 35; E3 > 800) which allows the transfer of ubiquitin, a small protein of 8.5 kDa onto the protein to be targeted either for its degradation or to modify its activity. This ubiquitinylation signal is reversible and many deubiquitinylases (DUB, ∼ 80 isoforms) also have an important role. E3 enzymes are the most numerous and their function is to recognize the target protein, which makes them important players in the specific action of UPS. The very nature of E3 and the complexity of their interactions with different partners offer a very broad field of investigation and therefore significant potential for the development of therapeutic approaches. Without being exhaustive, this review illustrates the different strategies that have already been implemented to fight against different pathologies (excluding bacterial or viral infections).
Mots clés : système ubiquitine-protéasome / ligases E3 / inhibiteurs / développements thérapeutiques / cancers
Key words: ubiquitin proteasome system / E3 ligases / inhibitors / therapeutic developments / cancers
© Société de Biologie, 2021
Introduction
Le Système Ubiquitine Protéasome (UPS) est le système protéolytique le plus complexe de l’organisme et son importance dans le contrôle du métabolisme cellulaire à l’état basal ou au cours de pathologies a transformé l’idée originelle d’une protéolyse servant uniquement de machine à éliminer les déchets en un système de contrôle précis de la vie, de la croissance et de la mort cellulaire (Glickman & Ciechanover, 2002). En effet, l’UPS est le chef d’orchestre de la plupart, si ce n’est de la totalité, des voies métaboliques, que ce soit directement ou indirectement. Le système UPS est pyramidal avec une enzyme E1 (une autre isoforme existe aussi mais son champ d’activité est restreint) qui active l’ubiquitine (Ub) et la transfère à une enzyme E2 (≥ 35 isoformes chez l’homme ; Michelle et al., 2009). L’enzyme E2 se combine ensuite à une E3 (> 800 isoformes) afin de transférer l’Ub sur la protéine cible. Les enzymes E3 ont pour rôle principal de cibler spécifiquement la protéine à modifier, mais souvent, elles ne possèdent aucune activité catalytique et c’est l’enzyme E2 qui effectue le transfert (Metzger et al., 2014 ; Zheng & Shabek, 2017). L’Ub est ainsi transférée sur le ɛ-NH2 d’un résidu lysine de la protéine cible. Cette ubiquitinylation peut ensuite donner lieu à la genèse d’une chaîne d’Ub qui utilisera l’une de ses 7 lysines et donnera lieu à une signalisation différente en fonction de la lysine utilisée. On distingue en effet les chaînes de type K6, K11, K27, K29, K33, K48 ou K63 qui ont des rôles différents (Polge et al., 2013 ; Park & Ryu, 2014). La complexité de cette signalisation est renforcée par la présence de chaînes mixtes (plusieurs résidus lysine de l’Ub sont utilisés dans une même chaîne) (Nakasone et al., 2013 ; French et al., 2017), voire la combinaison d’Ub avec d’autres protéines appelées Ub-like (Nie & Boddy, 2016). Cependant, la majorité du signal d’ubiquitinylation d’une cellule provient de protéines mono-ubiquitinylées (∼ 50 %) ou poly-ubiquitinylées avec des chaînes de type K48 (20 %) et K63 (20 %) utilisées principalement pour la dégradation de la cible par le protéasome et par le système Autophagie respectivement. Des variations importantes de leurs proportions relatives ont cependant été décrites en fonction de l’organe ou du type cellulaire considéré (Kaiser et al., 2011 ; Wagner et al., 2011).
De nombreuses déubiquitinylases (DUB, ≈ 100 isoformes) ont la capacité d’enlever tout ou partie du signal d’ubiquitinylation, ce qui permet le recyclage de l’Ub mais aussi un contrôle fin du devenir du substrat. Ainsi, l’implication des DUB dans l’UPS va bien au-delà du simple recyclage de l’Ub (Komander et al., 2009 ; Kim & Goldberg, 2017 ; Kuo & Goldberg, 2017) et l’expression de plusieurs DUB a été reliée au développement de différentes pathologies (pour une revue récente, voir Harrigan et al., 2018). Ces enzymes sont considérées comme des cibles thérapeutiques potentielles et plusieurs inhibiteurs de DUB sont en cours de développement, bien que la mise au point de ces inhibiteurs ait été confrontée à de nombreux écueils (Harrigan et al., 2018).
Les chaînes de type K48 entraînent généralement la protéine cible vers la dégradation par un complexe multi-catalytique, le protéasome 20S, lui-même soumis à une régulation fine par la présence de différents complexes régulateurs (PA700/19S RC, PA28/11S, PA200, PI31, CCRs) (Tomko & Hochstrasser, 2013 ; Bard et al., 2018). Cette dégradation rapide et hautement spécifique des protéines cibles permet de contrôler l’homéostasie protéique et donc le bon fonctionnement de la cellule et des organes. Par leur rôle essentiel dans la sélection des protéines à modifier, les enzymes E3 sont des acteurs majeurs du métabolisme cellulaire mais peuvent aussi être en cause dans la dérégulation des voies métaboliques (Rotin & Kumar, 2009 ; Esposito et al., 2017 ; Zheng & Shabek, 2017). La dégradation n’étant qu’un des aspects de l’ubiquitinylation, tous les signaux concernés (K11, chaînes mixtes, etc.) sont en réalité susceptibles de contrôler de nombreux évènements cellulaires importants justifiant pourquoi toutes les enzymes E3 peuvent potentiellement faire l’objet d’un ciblage thérapeutique.
Les enzymes E3, des cibles thérapeutiques de choix au sein de l’UPS
L’importance de l’UPS dans le contrôle de fonctions cellulaires majeures et dans le développement de nombreuses pathologies a très rapidement donné lieu à des stratégies pharmacologiques pour lutter notamment contre le développement de tumeurs. Le protéasome 20S possède les activités catalytiques nécessaires au clivage des protéines en peptides. Les premières molécules pharmacologiques visaient à inhiber le protéasome 20S à l’aide de petites molécules ciblant les résidus thréonine qui sont responsables de l’attaque de la liaison peptidique de la protéine à dégrader. Certaines drogues, comme le Velcade/Bortezomib, sont désormais utilisées chez l’Homme pour le traitement de myélomes (Einsele, 2014 ; Scott et al., 2016). Il est cependant facile d’imaginer ce que l’inhibition totale de l’UPS peut engendrer comme effets secondaires, puisque l’inhibition du protéasome entraîne des perturbations pour l’ensemble des protéines à dégrader dans tous les organes. De fait, de nombreux effets secondaires ont été rapportés après l’administration de Velcade : nausées, vomissements, diarrhée, constipation, faible nombre de plaquettes (thrombocytopénie), faible nombre de globules rouges (anémie), faible nombre de globules blancs (neutropénie), éruption cutanée, fièvre, neuropathie, fatigue, et perte d’appétit (Cengiz Seval & Beksac, 2018 ; Pancheri et al., 2020). De plus, l’emploi d’inhibiteurs du protéasome est désormais connu pour entraîner une neurotoxicité périphérique (Velasco et al., 2019). Il est donc nécessaire de développer des stratégies plus ciblées, impactant un nombre limité de fonctions cellulaires. Les enzymes de la cascade d’ubiquitinylation comme les E3 sont à la base de la spécificité de reconnaissance des substrats de l’UPS et représentent donc des cibles de choix pour de nouvelles générations de traitements thérapeutiques.
Des perturbations de l’ubiquitinylation des protéines cibles (ou substrats) liées à des enzymes E3 et impliquant différents signaux de l’UPS ont été observées dans de nombreuses pathologies (Zheng & Shabek, 2017 ; Taillandier & Polge, 2019 ; Wang et al., 2020). Le tableau 1 présente une liste non exhaustive des E3 liées à des dysfonctionnements métaboliques dont le nombre augmente régulièrement, et qui correspondent à un nombre croissant d’opportunités thérapeutiques, même si très peu de ces E3 ligases ont été étudiées en détail. En effet, leur activité et leur régulation sont, dans l’ensemble, largement inconnues. Il est important d’intégrer les E3 dans le contexte cellulaire où elles ont la capacité d’interagir avec différentes protéines (y compris d’autres E3) ou de subir des modifications post-traductionnelles. Le décryptage des interactions multiples que les E3 peuvent (ou doivent) mettre en place pour assurer leur fonction ralentit les avancées thérapeutiques mais ces études sont indispensables pour appréhender correctement leur fonction biologique et synthétiser des molécules à visée thérapeutique. À titre d’exemple, c’est la combinaison entre les E2 et les E3 qui définit le type de chaîne d’Ub et donc le devenir de la protéine cible (Soss et al., 2013 ; DiBello et al., 2016), de telle sorte que le mécanisme moléculaire régissant l’ubiquitinylation du substrat est donc souvent davantage lié au fonctionnement du couple E2-E3 qu’à l’enzyme E3 seule. Comme une E3 peut interagir avec plusieurs E2 et vice versa, les multiples combinaisons sont à la base de la spécificité de ciblage par l’UPS (Polge et al., 2015) et donc de la complexité du mécanisme. De plus, une protéine substrat peut être ciblée par différentes E3 en fonction du contexte et une E3 possède plusieurs cibles (Peris-Moreno et al., 2021). Par exemple, le suppresseur de tumeur p53 est ciblé par 17 E3 différentes en fonction du contexte (Mdm2/Hdm2, PirH2, ARF-BP1/Mule, Cop1, E6-AP, HUWE1, Topors, etc.) (Jain & Barton, 2010 ; Bang et al., 2019). Si cette multiplicité d’interactions potentielles augmente la spécificité du ciblage (y compris le ciblage thérapeutique), il engendre aussi un degré de complexité qui rend plus difficile la compréhension des mécanismes mis en jeu dans la cellule et donc la mise au point de molécules thérapeutiques efficaces.
De nombreuses pathologies sont liées à une surexpression, une dégradation accrue ou une mutation d’enzymes E3. Ce tableau n’est pas exhaustif car pour les seules maladies neuro-dégénératives, l’implication de 41 E3 différentes a été répertoriée (George et al., 2018).
Les différentes classes d’enzymes E3
On distingue classiquement trois classes d’enzymes E3 en fonction de leur mécanisme d’action : Les E3 à domaine RING (Really Interesting New Genes), les E3 à domaine HECT (Homologous to E6-AP Carboxyl Terminus) et les E3 de type RBR (Ring Between Ring) qui fonctionnent comme des hybrides de RING et HECT E3 (Figure 1) (Voir Zheng & Shabek, 2017 pour une revue détaillée). Une 4e classe a été décrite récemment, les RCR (Ring Cysteine Relay) (Pao et al., 2018), mais elle ne sera pas abordée dans cette revue.
Il existe 28 enzymes E3 à domaine HECT (Sluimer & Distel, 2018 ; Wang et al., 2020) et > 20 enzymes de type RBR (Dove & Klevit, 2017), qui reçoivent l’ubiquitine de l’enzyme E2 et la transfèrent au substrat. Ces E3 possèdent donc une activité catalytique intrinsèque mais, très souvent (notamment les E3 à domaine HECT), elles ont besoin de cofacteurs pour pouvoir se lier efficacement au substrat.
Les E3 à domaine RING sont de loin les plus nombreuses (> 600) et fonctionnent comme un hub accueillant la protéine ciblée et l’enzyme E2, cette dernière possédant l’activité catalytique (Deshaies & Joazeiro, 2009 ; Zheng & Shabek, 2017). Le domaine RING qui accueille l’enzyme E2 est stabilisé par 2 atomes de zinc mais il existe une version modifiée du domaine RING, appelée U-Box, où la stabilisation est assurée par des liaisons hydrophobes et hydrogène. Les RING E3 sont soit monomériques, soit formées par une association de plusieurs sous-unités (Deshaies & Joazeiro, 2009 ; Zheng & Shabek, 2017), ces dernières étant dénommées CRL (Cullin RING Ligase) (Figure 2). Dans le cas des E3 de type CRL, l’architecture comprend une protéine servant à l’échafaudage du complexe (Culline1-7), une protéine acceptant l’E2 (2 membres) et une protéine adaptatrice (4 membres) qui elle-même se lie à la protéine réceptrice du substrat dont il existe plus de 80 membres (Figure 2). Les combinaisons entre les différentes sous-unités sont spécifiques d’une E3 donnée et la nomenclature est basée sur le type de culline et sur le récepteur du substrat ; par exemple, l’enzyme CRL1FBXO32 est formée par la culline-1, FBXO32 est la protéine réceptrice des substrats, la protéine adaptatrice est obligatoirement Skp1 et Rbx1 est la protéine réceptrice de l’E2 (Voir Figure 2 pour les combinaisons en fonction de la culline). Pour les E3 de type CRL, les surfaces d’interaction entre sous-unités sont nombreuses, permettant de concevoir autant de stratégies potentiellement utilisables (Figure 3). Cependant, le ciblage de certaines interactions peut altérer la fonction de plusieurs E3 ; c’est le cas par exemple d’un inhibiteur empêchant la fixation d’une E2 sur une culline, qui entraînerait une inhibition de tous les complexes impliquant cette dernière. Dans certains cas, des cofacteurs peuvent intervenir pour faciliter ou stabiliser l’interaction entre l’E3 et la protéine cible. Les multiples interactions nécessaires au transfert de l’ubiquitine sur le substrat sont autant de surfaces pouvant être ciblées par des molécules pharmacologiques. Il est important de noter que de nombreuses E3 sont monomériques mais qu’elles ont la capacité, voire l’obligation, de former des homo-polymères afin d’assurer leur fonction dans la cellule (Koliopoulos et al., 2016 ; Balaji & Hoppe, 2020).
 |
Figure 1 Les protéines ciblées par l’UPS sont ubiquitinylées grâce à une cascade enzymatique E1 (1 membre, 2 isoformes), E2 (35 membres), E3 (> 800 membres). L’ubiquitine (Ub), une petite protéine de 8,5 kDa, est activée en présence d’ATP par l’enzyme E1 qui la transfère sur une enzyme E2. Le complexe est ensuite recruté par une enzyme E3 dont le rôle principal est de reconnaître la protéine cible à marquer par l’Ub. Il existe plusieurs familles d’enzyme E3 : les RING-E3 (mono sous-unité ou multi sous-unités) sont les plus nombreuses (> 600) et sont dépourvues d’activité catalytique ; les HECT-E3 reçoivent l’Ub de l’enzyme E2 et la transfèrent sur la protéine cible ; les RBR-E3 possèdent des caractéristiques hybrides grâce à la présence d’un deuxième domaine RING qui permet le transfert de l’Ub sur la protéine cible. |
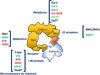 |
Figure 2 Les E3 de type SCF (Skip Cullin F-box Ligase) ou CRL (Cullin RING Ligase) sont des assemblages de plusieurs sous unités différentes dont la base est assurée par les cullines (Cul). Celles-ci interagissent avec une sous-unité acceptrice de l’enzyme E2 et une protéine adaptatrice qui porte la sous-unité réceptrice de la protéine cible (substrat). Le code couleur et les « * » indiquent les combinaisons entre les différentes sous-unités. Par exemple, Cul-1 se lie uniquement à l’adaptateur Skp1 qui porte une des nombreuses protéines de type F-box (par exemple FBXO32), et le récepteur de l’enzyme E2 est la protéine Rbx1/Roc1 ; la dénomination de cette E3 est donc CRL1FBXO32. La spécificité de reconnaissance de la protéine cible est donnée par les protéines réceptrices (> 80) qui sont classées en différentes familles selon le domaine interagissant avec l’adaptateur. La présence de cofacteurs peut être nécessaire pour stabiliser le complexe ou sa liaison avec la protéine cible. |
 |
Figure 3 Les E3 possèdent de nombreuses surfaces d’interaction avec leurs partenaires, chacune de ces surfaces pouvant faire l’objet d’une stratégie de ciblage pour empêcher l’ubiquitinylation de la protéine cible (substrat). |
Stratégies utilisées pour la découverte d’agents pharmacologiques inhibant les enzymes E3
De nombreuses molécules à visée thérapeutique ciblant différentes E3 ont été développées ces dernières années (Li et al., 2018b). Une multiplicité d’approches a été mise en œuvre pour identifier des molécules susceptibles d’inhiber l’activité des E3, mais sans spécificité vis à vis de ce type d’enzyme : criblage à haut débit de petites molécules, criblage in silico, synthèse basée sur la structure cristallographique, sélection de molécules basée sur les résidus intervenant dans les interactions au sein du complexe UPS, etc. L’identification de molécules efficaces pour inhiber ces interactions est quant à elle intimement liée à la fonction des E3 et concerne notamment l’activité d’ubiquitinylation, les niveaux d’un substrat endogène ou d’un substrat modèle dans des cellules en culture, la liaison entre l’E3 et sa cible (par mesure de FRET, Fluorescence Resonance Energy Transfer), etc.
On peut définir 5 grandes stratégies pour empêcher une enzyme E3 de modifier sa cible et inhiber sa dégradation :
-
Interférer avec la reconnaissance du substrat ;
-
Cibler des surfaces régulatrices spécifiques (impliquant un cofacteur ou la liaison avec l’enzyme E2) ;
-
Cibler la cystéine du site catalytique ;
-
Moduler l’état oligomérique (pour des RING E3 monomériques ou des HECT E3) ;
-
Bloquer le transfert de l’Ub (entre l’E2 et l’E3, entre l’E2 et le substrat ou entre l’E3 et le substrat).
Cette revue n’a pas pour objet de décrire de façon exhaustive tous les développements de molécules inhibitrices mais de donner un aperçu de la multiplicité de stratégies offertes aux futures recherches dans ce domaine encore naissant mais extrêmement prometteur.
Interférer avec la reconnaissance du substrat
Cette stratégie semble a priori la plus logique puisque la reconnaissance entre l’E3 et sa cible est l’étape clé pour la modification/dégradation de cette cible-substrat et que toute enzyme E3, quelle que soit sa structure, possède une surface d’interaction avec le substrat.
Cependant pour les E3 de type HECT, très peu de molécules interférant dans la reconnaissance entre l’E3 et sa cible ont été découvertes. Elles sont généralement peu efficaces et leur mécanisme d’inhibition n’est généralement pas connu (Watt et al., 2018). L’échec de cette stratégie est sans doute lié au peu d’affinité que les E3 de type HECT possèdent intrinsèquement pour leur cible (Weber et al., 2019). En effet, il faut généralement la présence d’une protéine adaptatrice qui joue le rôle d’un cofacteur, et c’est surtout le ciblage de la surface d’interaction entre l’enzyme E3 et la protéine adaptatrice qui est efficace (par exemple pour lutter contre des infections virales : Han et al., 2014 ; Malecka et al., 2014 ; Murayama et al., 2020 ; Offensperger et al., 2020).
Le ciblage de l’interaction E3-substrat est par contre beaucoup plus efficace pour les RING E3. Par exemple, l’enzyme CRL2VHL est connue pour sa capacité à cibler HIF1α qui sera ensuite dégradé par le protéasome (Cardote et al., 2017). HIF1α est un facteur de transcription qui régule environ 2 % des gènes de l’organisme et plus particulièrement ceux qui sont impliqués dans la détection de l’oxygène et dans la réponse à l’hypoxie. Une augmentation de la dégradation de HIF1α permet d’accroître la production d’érythropoïétine afin de traiter l’anémie chronique observée au cours de cancers ou de l’insuffisance rénale. La sous-unité VHL de CRL2VHL reconnaît HIF1 principalement par la présence d’une proline qui est hydroxylée en conditions normoxiques sous la dépendance d’oxygène. Les premières molécules testées ont donc ciblé précisément cette interaction en utilisant des analogues de l’hydroxyproline, comme le composé 15 (KD = 5 μM) (Bulatov & Ciulli, 2015)]. Grâce à la connaissance de la structure cristallographique, ces molécules ont pu être optimisées pour aboutir à des composés plus efficaces interagissant avec d’autres résidus proches de l’hydroxyproline, tel le composé 7 (KD = 185 nM) (Bulatov & Ciulli, 2015).
De nombreuses études ont été effectuées sur des protéines impliquées dans le développement de cancers et plus particulièrement p53, aussi connu comme le gardien du génome, car on estime que le développement de 50 % des cancers est dû à des mutations de ce facteur de transcription (Wang et al., 2017). L’anti-oncogène p53 est ciblé par plusieurs E3 (Jain & Barton, 2010 ; Bang et al., 2019) mais MDM2 est la principale et 7 % des cancers sont liés à une expression anormalement élevée de MDM2 (Hou et al., 2019). Dans le cas de p53, le but est donc de le protéger pour lutter contre le développement des cellules tumorales. MDM2 est une RING E3 de type monomérique, mais elle se lie à la protéine MDMX catalytiquement inactive pour former un hétérodimère capable de se lier à p53 (Wade et al., 2013). Comme en réalité à la fois MDM2 et MDMX peuvent se lier à p53, plusieurs stratégies permettent théoriquement de protéger p53 : empêcher la liaison p53-MDM2 ou la liaison p53-MDMX, ou les deux. Trois résidus hydrophobes de p53 (Phe19-Trp23-Leu26) sont particulièrement importants pour la liaison avec MDM2 ou MDMX (Vassilev et al., 2004 ; Grasberger et al., 2005). Toutes les molécules efficaces pour empêcher la liaison de ces E3 avec p53 sont des mimes de la structure formée par ces trois résidus. De nombreux composés sont particulièrement efficaces et inhibent MDM2 (nutlines, spirooxindoles et indoles, benzodiazépines), MDMX (imizadolyl-indole) ou les deux (imidazoles, isoquinolinones et piperidones, pyrrolidone et pyrrolidine), avec des IC50 de 1 à 90 nM (Wang et al., 2017 ; Lemos et al., 2019 ; Skalniak et al., 2019 ; Konopleva et al., 2020 ; Quartararo et al., 2020). Plusieurs de ces composés sont en essai clinique, dont 7 en phase I et 2 en phase II. Le composé RG7388/idasanutline (IC50 = 6–30 nM) est d’ores et déjà en essai de phase III pour des leucémies (Duffy et al., 2020 ; Montesinos et al., 2020).
Blocage du transfert d’Ub de l’enzyme E3 au substrat
SMURF1/2 sont des E3 ligases de type HECT qui régulent négativement aussi bien RhoB (suppresseur de tumeur) (Wang et al., 2014) que SMAD1 et SMAD2 (inhibiteurs de la prolifération cellulaire) (Seth et al., 2017 ; Fu et al., 2020), et une hyperactivité de SMURF1/2 est liée à des cancers métastatiques agressifs (Fu et al., 2020). Un criblage de 5000 molécules a permis d’identifier un composé (HS-152) qui prévient la dégradation de RhoB et de SMAD1 (IC50 = 2,1–4,4 μM) via l’inhibition spécifique de SMURF1/2 sans interagir avec d’autres enzymes de type HECT comme NEDD4-1/2 (Tian et al., 2019). L’analyse de son mécanisme d’action a permis d’établir (i) que la cible se liait à SMURF1/2, (ii) que l’Ub se liait efficacement à SMURF1/2 mais (iii) que le transfert vers le substrat était bloqué (Tian et al., 2019).
Ciblage de la cystéine du site catalytique de l’enzyme E3
Cette stratégie est limitée aux E3 possédant une activité catalytique intrinsèque (HECT, RBR). Elle a été utilisée pour identifier des inhibiteurs de SMURF2 et NEDD4 (Mund et al., 2014). Les auteurs ont utilisé la technique de phage display avec une banque de phages exprimant de petits peptides (14 acides aminés) comportant 3 résidus cystéine. Plusieurs peptides bi-cycliques interagissant avec les peptides riches en cystéine ont été sélectionnés pour leur capacité à se lier à SMURF2 et NEDD4. En particulier le composé Heclin (HECT ligase inhibitor) s’est révélé capable d’interagir avec la cystéine du site actif de SMURF2 et NEDD4 (IC50 = 2–3 μM) (Mund et al., 2014). Cependant, ce composé n’est pas spécifique de ces 2 E3 ligases car il inhibe aussi l’activité de toutes les enzymes HECT testées jusqu’à présent (MULE/HUWE1, UBE3C et WWP1). Il sera donc nécessaire dans des études futures d’intégrer d’autres résidus voisins de la cystéine du site catalytique pour obtenir des inhibiteurs plus spécifiques.
Une étude in silico destinée à évaluer les propriétés anti-prolifératives de l’indole-3-carbinol (I3C), une molécule végétale présente dans des crucifères (choux, brocolis), a montré que ce composé pouvait potentiellement se fixer sur le site actif de l’enzyme HECT NEDD4-1 (Quirit et al., 2017 ; Murayama et al., 2020). Plusieurs dérivés ont été obtenus et testés, et le 1-benzyl-indole-3-carbinol (1-benzyl-I3C) s’est montré le plus efficace (IC50 = 30 μM). Cependant, au lieu de se lier au niveau du site actif contenant la cystéine qui reçoit l’Ub, cette molécule vient se fixer sur l’exosite de l’Ub. Plusieurs enzymes d’ubiquitinylation possèdent en effet 2 sites de liaison à l’Ub, celui qui effectue la catalyse (présence d’une cystéine) permettant de transférer l’Ub sur la protéine cible, et l’exosite où vient interagir une seconde molécule d’Ub. La présence d’une molécule d’Ub sur l’exosite est nécessaire à l’élongation de la chaîne d’Ub, étape nécessaire pour la reconnaissance de la protéine substrat et sa dégradation par le protéasome 26S. Au final, I3C inhibe l’élongation de la chaîne d’ubiquitine mais sans interagir avec le site actif.
Blocage de la fonction de l’enzyme E2 : solution ou utopie ?
Parmi les 40 enzymes E2 répertoriées chez l’homme, 35 sont dédiées au transfert de l’Ub sur une protéine cible, la nomenclature actuelle étant UBE2x pour les distinguer (par exemple, UBE2A) (van Wijk & Timmers, 2010 ; Wenzel et al., 2011). Les autres ont une fonction similaire, mais utilisent des protéines Ub-like comme ISG15 (Zuo et al., 2016), SUMO (Namuduri et al., 2020) ou NEDD8 (Bulatov & Ciulli, 2015). Toutes ces enzymes E2 jouent des rôles importants dans la cellule. Ainsi, UBE2T est considérée comme un oncogène potentiel car sa surexpression est liée à une instabilité chromosomique et prédispose à la survenue de cancers (Zhu et al., 2020). Une surexpression de UBE2L3 est observée dans certaines maladies auto-immunes (Wang et al., 2012 ; Kim et al., 2020) et UBE2W est impliquée à la fois dans des désordres du système immunitaire et de la reproduction (Wang et al., 2016). Le nombre relativement limité de ces E2 par rapport aux E3 (> 800) implique que chaque E2 coopère avec de nombreuses E3 et chaque E2 peut donc être engagée dans différentes/nombreuses fonctions métaboliques. À première vue, cibler l’E2 pourrait avoir pour conséquence le blocage étendu du système UPS avec le risque de survenue d’effets secondaires. De plus, certaines E2 possèdent une homologie de séquence importante (96 % pour UBE2A et UBE2B) (Polge et al., 2015), ce qui rend théoriquement plus difficile la mise au point d’éventuels inhibiteurs spécifiques. Cependant, ces E2 ont développé des surfaces d’interactions multiples impliquant un nombre limité d’acides aminés, ce qui leur confère une grande spécificité dans la reconnaissance de leur partenaire. Cette caractéristique, qui est à la base de leurs fonctions cellulaires multiples, permet donc d’envisager le développement de molécules empêchant la reconnaissance spécifique d’une E3 donnée sans affecter la liaison avec les autres E3. Un exemple typique est celui d’UBE2B dont la phosphorylation sur un seul résidu (Ser120) lui permet de changer de partenaire E3 (RAD18 vs. UBR1) et de fonction (respectivement monoUb vs. polyUb sur la cible) (Sarcevic et al., 2002 ; Kumar et al., 2010).
Stabilisation de la liaison E2-E3
Un premier exemple de blocage de la fonction de l’E2 concerne la famille MYC (MYC/c-MYC, MYCL, MYCN), qui est impliquée dans la survenue d’environ 50 % de cancers, parmi les plus agressifs. De par sa structure et sa localisation, la protéine c-MYC est considérée comme ne pouvant pas être ciblée pharmacologiquement (Carabet et al., 2018). Il est donc apparu nécessaire de se focaliser sur des protéines interagissant avec MYC, comme son co-activateur Max ou son répresseur MAD1. MAD1 est ciblé pour sa dégradation par l’enzyme E3 cIAP1/BIRC2, une RING E3 monomérique. L’inhibition de la dégradation de MAD1 pourrait ainsi diminuer les niveaux cellulaires de c-MYC et donc empêcher la progression cancéreuse. Le criblage d’une banque de 50 000 composés a permis d’identifier des inhibiteurs de l’activité d’ubiquitinylation de cIAP1, notamment la molécule D19-14 qui stabilise la liaison entre cIAP1 et une de ses E2 (UBE2D ou UBE2V1) (Li et al., 2018a). En effet, si le processus d’ubiquitinylation implique nécessairement une liaison E2-E3, celle-ci doit être furtive pour le bon déroulé du processus. En d’autres termes, le transfert d’Ub de l’E2 sur la cible doit s’accompagner d’un relargage rapide de l’E2. Ainsi, en cas de stabilisation de la liaison E2-E3, le processus d’ubiquitinylation est inhibé, ce qui est le cas avec les molécules de type D19 qui sont capables d’inhiber la croissance de cellules en culture et la progression tumorale dans un modèle de cancer chez la souris. Elles représentent un espoir de développement thérapeutique pour lutter contre la progression de cancers MYC-dépendants. De façon plus générale, cette stratégie offre de nombreuses perspectives car les zones d’interaction entre E2 et E3 utilisent plusieurs surfaces sur chaque enzyme et les acides aminés impliqués sont très différents d’un couple à un autre.
Inhibition de l’interaction E2-E3
Cette stratégie est l’opposé de la précédente mais présente les mêmes avantages du fait du ciblage d’un couple particulier avec une grande spécificité. Un criblage à haut débit de petites molécules a été utilisé pour identifier des inhibiteurs du site récepteur de l’E2 (RBX1/ROC1) dans les enzymes E3 de type RING CRL (Wu et al., 2016). Le criblage a été effectué par une approche de FRET en suivant le transfert d’Ub de l’E2 à l’E3. Un composé s’est révélé particulièrement efficace pour inhiber l’interaction ROC1-UBE2R1, la suramine (IC50 = 0,22 μM) (Wu et al., 2016 ; Zhang et al., 2020b). La suramine et d’autres composés dérivés ciblent 6 acides aminés présents dans une poche de ROC1 qui interagissent avec UBE2R1. L’efficacité de ces composés est similaire pour des enzymes de type CRL1 et CRL3 qui utilisent ROC1 comme accepteur de l’Ub. Il est important de souligner que la suramine et ses dérivés sont capables de discriminer les enzymes E2 car ils sont beaucoup moins efficaces contre UBE2D et UBE2M, bien que la poche de reconnaissance sur ROC1 soit la même (Wu et al., 2016 ; Zhang et al., 2020b).
Bloquer la décharge de l’Ub sur la protéine cible
La protéine p27Kip1 joue un rôle important dans la progression du cycle cellulaire en phase G1. Une altération des niveaux de p27Kip1 induit une perturbation du cycle cellulaire et sa sous-expression ou sa perte de fonction due à des mutations entraîne le développement de nombreux cancers (Bencivenga et al., 2017). La protéine p27Kip1 est ciblée par l’enzyme E3 CRL1skp2 et le criblage d’inhibiteurs de l’activité d’ubiquitinylation de cette E3 a permis d’identifier le composé CC0651 qui inhibe efficacement l’ubiquitinylation de p27Kip1 (IC50 = 1,72 μM) (Ceccarelli et al., 2011). Par cristallographie, les auteurs ont montré que CC0651 se lie à l’enzyme E2 UBE2R1 dans une région distante du site catalytique et de la zone d’interaction avec l’E3. CC0651 conduit à une modification de la structure de l’E2 qui bloque le transfert de l’Ub. Le principe d’action de CC0651 fait que toutes les enzymes utilisant UBE2R1, comme CRL1β-TrCP, CRL1Fbw7 ou CRL1Cdc4, vont être inhibées, ce qui occasionne la stabilisation de nombreuses protéines et potentiellement des effets secondaires indésirables.
Conclusion et perspectives
Le système UPS contrôle la majorité des voies métaboliques et représente donc une cible de choix pour lutter contre de nombreuses pathologies. Bien que les premières études aient ciblé le protéasome pour lutter contre la progression tumorale, cette stratégie ne permet pas une modulation fine de l’expression de protéines spécifiques, comme par exemple des protéines pro-oncogéniques ou anti-tumorales. La spécificité d’action de l’UPS étant portée par la combinaison des enzymes E2 et E3, il est logique que de nombreux travaux se soient focalisés sur leur ciblage depuis près de 20 ans. Les études cliniques effectuées sur l’UPS illustrent clairement l’intérêt des chercheurs et cliniciens pour les enzymes d’ubiquitinylation car il y a eu, en 2020, plus de 21 composés pharmacologiques testés contre des E3 versus seulement 4 ciblés sur le protéasome (Zhang et al., 2020a). Il faut noter que plus de 25 composés dirigés contre des DUB ont aussi fait l’objet d’études cliniques, ce qui confirme le potentiel des enzymes responsables du marquage, ou du « démarquage », de la cible. Malheureusement, très peu de ces enzymes sont suffisamment connues (rôle, mécanisme d’action, structure, etc.) pour permettre le développement de molécules pharmacologiques efficaces et la majorité des études se sont focalisées sur un nombre restreint d’oncogènes.
Une des difficultés des études futures est liée à l’extrême diversité des processus d’ubiquitinylation, ce qui implique que chaque cible/stratégie doit être adaptée au cas par cas. Outre les différentes combinaisons entre E2 et E3, des modifications post-traductionnelles (phosphorylation, etc.) ou des combinaisons entre plusieurs E2, plusieurs E3 ou plusieurs E2 et E3 rendent nécessaires une parfaite connaissance des mécanismes afin d’optimiser la stratégie permettant l’obtention de molécules à la fois efficaces et spécifiques.
Un point important concerne le degré d’inhibition. En effet, chaque E2, comme chaque E3, exerce des fonctions pléiotropiques et l’inhibition totale d’une enzyme peut être délétère. À titre d’exemple, une hypoactivité de l’E3 ligase E6AP/UBE3A est responsable du syndrome d’Angelman, alors qu’une hyperactivité de cette même enzyme est associée à certaines formes d’autisme (Khatri & Man, 2019). Les nouvelles molécules pharmacologiques devront permettre une modulation de l’activité de leur cible plutôt qu’induire une inhibition totale. Cet objectif devrait pouvoir être atteint en jouant sur les posologies mais surtout en optimisant la structure chimique des composés à synthétiser.
Abréviations
FRET : Fluorescence Resonance Energy Transfer
HECT : Homologous to E6-AP Carboxyl Terminus
RING : Really Interesting New Genes
UPS : Système Ubiquitine Protéasome
Références
- Annunziata, C.M., Davis, R.E., Demchenko, Y., Bellamy, W., Gabrea, A., Zhan, F., Lenz, G., Hanamura, I., Wright, G., Xiao, W., Dave, S., Hurt, E.M., Tan, B., Zhao, H., Stephens, O., Santra, M., Williams, D.R., Dang, L., Barlogie, B., Shaughnessy, J.D. Jr, Kuehl, W.M., Staudt, L.M. (2007). Frequent engagement of the classical and alternative NF-kappaB pathways by diverse genetic abnormalities in multiple myeloma. Cancer Cell, 12, 115-130. [CrossRef] [PubMed] [Google Scholar]
- Babu, J.R., Geetha, T., Wooten, M.W. (2005). Sequestosome 1/p62 shuttles polyubiquitinated tau for proteasomal degradation. J Neurochem, 94, 192-203. [CrossRef] [PubMed] [Google Scholar]
- Balaji, V., Hoppe, T. (2020). Regulation of E3 ubiquitin ligases by homotypic and heterotypic assembly. F1000Res, 9. [Google Scholar]
- Bang, S., Kaur, S., Kurokawa, M. (2019). Regulation of the p53 family proteins by the ubiquitin proteasomal pathway. Int J Mol Sci, 21, https://doi.org/10.3390/ijms21010261. [CrossRef] [Google Scholar]
- Bard, J.A.M., Goodall, E.A., Greene, E.R., Jonsson, E., Dong, K.C., Martin, A. (2018). Structure and function of the 26S proteasome. Annu Rev Biochem, 87, 697-724. [CrossRef] [PubMed] [Google Scholar]
- Bencivenga, D., Caldarelli, I., Stampone, E., Mancini, F.P., Balestrieri, M.L., Della Ragione, F., Borriello, A. (2017). p27(Kip1) and human cancers: a reappraisal of a still enigmatic protein. Cancer Lett, 403, 354-365. [CrossRef] [PubMed] [Google Scholar]
- Boisson, B., Laplantine, E., Prando, C., Giliani, S., Israelsson, E., Xu, Z., Abhyankar, A., Israel, L., Trevejo-Nunez, G., Bogunovic, D., Cepika A.-M., MacDuff D., Chrabieh M., Hubeau M., Bajolle, F., Debré, M., Mazzolari, E., Vairo, D., Agou, F., Virgin, H.W., Bossuyt, X., Rambaud, C., Facchetti, F., Bonnet, D., Quartier, P., Fournet, J.-C., Pascual, V., Chaussabel, D., Notarangelo, L.D., Puel, A., Israël, A., Casanova, J.-L., Picard, C. (2012). Immunodeficiency, autoinflammation and amylopectinosis in humans with inherited HOIL-1 and LUBAC deficiency. Nat Immunol, 13, 1178-1186. [CrossRef] [PubMed] [Google Scholar]
- Bulatov, E., Ciulli, A. (2015). Targeting Cullin-RING E3 ubiquitin ligases for drug discovery: structure, assembly and small-molecule modulation. Biochem J, 467, 365-386. [CrossRef] [PubMed] [Google Scholar]
- Carabet, L.A., Rennie, P.S., Cherkasov, A. (2018). Therapeutic inhibition of Myc in cancer. Structural bases and computer-aided drug discovery approaches. Int J Mol Sci, 20, 120. [CrossRef] [Google Scholar]
- Cardote, T.A.F., Gadd, M.S., Ciulli, A. (2017). Crystal structure of the Cul2-Rbx1-EloBC-VHL ubiquitin ligase complex. Structure, 25, 901-911, e903. [CrossRef] [PubMed] [Google Scholar]
- Ceccarelli, D.F., Tang, X., Pelletier, B., Orlicky, S., Xie, W., Plantevin, V., Neculai, D., Chou, Y.C., Ogunjimi, A., Al-Hakim, A., Varelas, X., Koszela, J., Wasney, G.A., Vedadi, M., Dhe-Paganon, S., Cox, S., Xu, S., Lopez-Girona, A., Mercurio, F., Wrana, J., Durocher, D., Meloche, S., Webb, D.R., Tyers, M., Sicheri, F. (2011). An allosteric inhibitor of the human Cdc34 ubiquitin-conjugating enzyme. Cell, 145, 1075-1087. [CrossRef] [PubMed] [Google Scholar]
- Cengiz Seval, G., Beksac, M. (2018). The safety of bortezomib for the treatment of multiple myeloma. Expert Opin Drug Saf, 17, 953-962. [CrossRef] [PubMed] [Google Scholar]
- Clarke, B.A., Drujan, D., Willis, M.S., Murphy, L.O., Corpina, R.A., Burova, E., Rakhilin, S.V., Stitt, T.N., Patterson, C., Latres, E., Glass, D.J. (2007). The E3 Ligase MuRF1 degrades myosin heavy chain protein in dexamethasone-treated skeletal muscle. Cell Metab, 6, 376-385. [CrossRef] [PubMed] [Google Scholar]
- Cohen, P., Strickson, S. (2017). The role of hybrid ubiquitin chains in the MyD88 and other innate immune signalling pathways. Cell Death Differ, 24, 1153-1159. [CrossRef] [PubMed] [Google Scholar]
- Cohen, S., Zhai, B., Gygi, S.P., Goldberg, A.L. (2012). Ubiquitylation by Trim32 causes coupled loss of desmin, Z-bands, and thin filaments in muscle atrophy. J Cell Biol, 198, 575-589. [CrossRef] [PubMed] [Google Scholar]
- Deshaies, R.J., Joazeiro, C.A. (2009). RING domain E3 ubiquitin ligases. Annu Rev Biochem, 78, 399-434. [CrossRef] [PubMed] [Google Scholar]
- DiBello, A., Datta, A.B., Zhang, X., Wolberger, C. (2016). Role of E2-RING interactions in governing RNF4-mediated substrate ubiquitination. J Mol Biol, 428, 4639-4650. [CrossRef] [PubMed] [Google Scholar]
- Doss-Pepe, E.W., Chen, L., Madura, K. (2005). Alpha-synuclein and parkin contribute to the assembly of ubiquitin lysine 63-linked multiubiquitin chains. J Biol Chem, 280, 16619-16624. [CrossRef] [PubMed] [Google Scholar]
- Dove, K.K., Klevit, R.E. (2017). RING-between-RING E3 ligases: emerging themes amid the variations. J Mol Biol, 429, 3363-3375. [CrossRef] [PubMed] [Google Scholar]
- Duffy, M.J., Synnott, N.C., O’Grady, S., Crown, J. (2020). Targeting p53 for the treatment of cancer. Semin Cancer Biol, S1044-579X(20)30160-7. [Google Scholar]
- Einsele, H. (2014). Bortezomib. Recent results. Cancer Res, 201, 325–345. [Google Scholar]
- Esposito, D., Koliopoulos, M.G., Rittinger, K. (2017). Structural determinants of TRIM protein function. Biochem Soc Trans, 45, 183-191. [CrossRef] [PubMed] [Google Scholar]
- Fabris, L., Berton, S., Pellizzari, I., Segatto, I., D’Andrea, S., Armenia, J., Bomben, R., Schiappacassi, M., Gattei, V., Philips, M.R., Vecchione, A., Belletti, B., Baldassarre, G. (2015). p27kip1 controls H-Ras/MAPK activation and cell cycle entry via modulation of MT stability. Proc Natl Acad Sci USA, 112, 13916-13921. [CrossRef] [Google Scholar]
- French, M.E., Klosowiak, J.L., Aslanian, A., Reed, S.I., Yates, J.R., 3rd, Hunter, T. (2017). Mechanism of ubiquitin chain synthesis employed by a HECT domain ubiquitin ligase. J Biol Chem, 292, 10398-10413. [CrossRef] [PubMed] [Google Scholar]
- Fu, L., Cui, C.P., Zhang, X., Zhang, L. (2020). The functions and regulation of Smurfs in cancers. Semin Cancer Biol, 67, 102-116. [CrossRef] [PubMed] [Google Scholar]
- George, A.J., Hoffiz, Y.C., Charles, A.J., Zhu, Y., Mabb, A.M. (2018). A comprehensive atlas of E3 ubiquitin ligase mutations in neurological disorders. Front Genet, 9, 29. [CrossRef] [PubMed] [Google Scholar]
- Glickman, M.H., Ciechanover, A. (2002). The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiol Rev, 82, 373-428. [CrossRef] [PubMed] [Google Scholar]
- Grasberger, B.L., Lu, T., Schubert, C., Parks, D.J., Carver, T.E., Koblish, H.K., Cummings, M.D., LaFrance, L.V., Milkiewicz, K.L., Calvo, R.R., Maguire, D., Lattanze, J., Franks, C.F., Zhao, S., Ramachandren, K., Bylebyl, G.R., Zhang, M., Manthey, C.L., Petrella, E.C., Pantoliano, M.W., Deckman, I.C., Spurlino, J.C., Maroney, A.C., Tomczuk, B.E., Molloy, C.J., Bone, R.F. (2005). Discovery and cocrystal structure of benzodiazepinedione HDM2 antagonists that activate p53 in cells. J Med Chem, 48, 909-912. [CrossRef] [PubMed] [Google Scholar]
- Han, Z., Lu, J., Liu, Y., Davis, B., Lee, M.S., Olson, M.A., Ruthel, G., Freedman, B.D., Schnell, M.J., Wrobel, J.E., Reitz, A.B., Harty, R.N. (2014). Small-molecule probes targeting the viral PPxY-host Nedd4 interface block egress of a broad range of RNA viruses. J Virol, 88, 7294-7306. [CrossRef] [PubMed] [Google Scholar]
- Harrigan, J.A., Jacq, X., Martin, N.M., Jackson, S.P. (2018). Deubiquitylating enzymes and drug discovery: emerging opportunities. Nat Rev Drug Discov, 17, 57-78. [CrossRef] [PubMed] [Google Scholar]
- Hock, A.K., Vousden, K.H. (2014). The role of ubiquitin modification in the regulation of p53. Biochim Biophys Acta, 1843, 137-149. [CrossRef] [PubMed] [Google Scholar]
- Hou, H., Sun, D., Zhang, X. (2019). The role of MDM2 amplification and overexpression in therapeutic resistance of malignant tumors. Cancer Cell Int, 19, 216. [CrossRef] [PubMed] [Google Scholar]
- Jain, A.K., Barton, M.C. (2010). Making sense of ubiquitin ligases that regulate p53. Cancer Biol Ther, 10, 665-672. [CrossRef] [PubMed] [Google Scholar]
- Kaiser, S.E., Riley, B.E., Shaler, T.A., Trevino, R.S., Becker, C.H., Schulman, H., Kopito, R.R. (2011). Protein standard absolute quantification (PSAQ) method for the measurement of cellular ubiquitin pools. Nat Methods, 8, 691-696. [CrossRef] [PubMed] [Google Scholar]
- Karbowski, M., Youle, R.J. (2011). Regulating mitochondrial outer membrane proteins by ubiquitination and proteasomal degradation. Curr Opin Cell Biol, 23, 476-482. [CrossRef] [PubMed] [Google Scholar]
- Kedar, V., McDonough, H., Arya, R., Li, H.H., Rockman, H.A., Patterson, C. (2004). Muscle-specific RING finger 1 is a bona fide ubiquitin ligase that degrades cardiac troponin I. Proc Natl Acad Sci USA, 101, 18135-18140. [CrossRef] [Google Scholar]
- Khatri, N., Man, H.Y. (2019). The autism and Angelman syndrome protein Ube3A/E6AP: the gene, E3 ligase ubiquitination targets and neurobiological functions. Front Mol Neurosci, 12, 109. [CrossRef] [PubMed] [Google Scholar]
- Kim, H.T., Goldberg, A.L. (2017). The deubiquitinating enzyme Usp14 allosterically inhibits multiple proteasomal activities and ubiquitin-independent proteolysis. J Biol Chem, 292, 9830-9839. [CrossRef] [PubMed] [Google Scholar]
- Kim, T., Bae, S.C., Kang, C. (2020). Synergistic activation of NF-kappaB by TNFAIP3 (A20) reduction and UBE2L3 (UBCH7) augment that synergistically elevate lupus risk. Arthritis Res Ther, 22, 93. [CrossRef] [PubMed] [Google Scholar]
- Knipscheer, P., Raschle, M., Smogorzewska, A., Enoiu, M., Ho, T.V., Scharer, O.D., Elledge, S.J., Walter, J.C. (2009). The Fanconi anemia pathway promotes replication-dependent DNA interstrand cross-link repair. Science, 326, 1698-1701. [CrossRef] [PubMed] [Google Scholar]
- Koliopoulos, M.G., Esposito, D., Christodoulou, E., Taylor, I.A., Rittinger, K. (2016). Functional role of TRIM E3 ligase oligomerization and regulation of catalytic activity. EMBO J, 35, 1204-1218. [CrossRef] [PubMed] [Google Scholar]
- Komander, D., Clague, M.J., Urbe, S. (2009). Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol, 10, 550-563. [CrossRef] [PubMed] [Google Scholar]
- Konopleva, M., Martinelli, G., Daver, N., Papayannidis, C., Wei, A., Higgins, B., Ott, M., Mascarenhas, J., Andreeff, M. (2020). MDM2 inhibition: an important step forward in cancer therapy. Leukemia, 34, 2858-2874. [CrossRef] [PubMed] [Google Scholar]
- Kontaxi, C., Piccardo, P., Gill, A.C. (2017). Lysine-directed post-translational modifications of tau protein in Alzheimer’s disease and related tauopathies. Front Mol Biosci, 4, 56. [CrossRef] [PubMed] [Google Scholar]
- Kumar, B., Lecompte, K.G., Klein, J.M., Haas, A.L. (2010). Ser(120) of Ubc2/Rad6 regulates ubiquitin-dependent N-end rule targeting by E3{alpha}/Ubr1. J Biol Chem, 285, 41300-41309. [CrossRef] [PubMed] [Google Scholar]
- Kumar, D., Kumar, P. (2019). Integrated mechanism of lysine 351, PARK2, and STUB1 in AbetaPP ubiquitination. J Alzheimers Dis, 68, 1125-1150. [CrossRef] [PubMed] [Google Scholar]
- Kuo, C.L., Goldberg, A.L. (2017). Ubiquitinated proteins promote the association of proteasomes with the deubiquitinating enzyme Usp14 and the ubiquitin ligase Ube3c. Proc Natl Acad Sci USA, 114, E3404-E3413. [CrossRef] [Google Scholar]
- Lagirand-Cantaloube, J., Offner, N., Csibi, A., Leibovitch, M.P., Batonnet-Pichon, S., Tintignac, L.A., Segura, C.T., Leibovitch, S.A. (2008). The initiation factor eIF3-f is a major target for atrogin1/MAFbx function in skeletal muscle atrophy. EMBO J, 27, 1266-1276. [CrossRef] [PubMed] [Google Scholar]
- Lee, J.T., Gu, W. (2010). The multiple levels of regulation by p53 ubiquitination. Cell Death Differ, 17, 86-92. [CrossRef] [PubMed] [Google Scholar]
- Lee, S., Challa-Malladi, M., Bratton, S.B., Wright, C.W. (2014). Nuclear factor-kappaB-inducing kinase (NIK) contains an amino-terminal inhibitor of apoptosis (IAP)-binding motif (IBM) that potentiates NIK degradation by cellular IAP1 (c-IAP1). J Biol Chem, 289, 30680-30689. [CrossRef] [PubMed] [Google Scholar]
- Lehmann, A.R. (2003). DNA repair-deficient diseases, xeroderma pigmentosum, Cockayne syndrome and trichothiodystrophy. Biochimie, 85, 1101-1111. [CrossRef] [PubMed] [Google Scholar]
- Lemos, A., Gomes, A.S., Loureiro, J.B., Brandao, P., Palmeira, A., Pinto, M.M.M., Saraiva, L., Sousa, M.E. (2019). Synthesis, biological evaluation, and in silico studies of novel aminated xanthones as potential p53-activating agents. Molecules, 24, 1975. [CrossRef] [Google Scholar]
- Li, H., Fang, Y., Niu, C., Cao, H., Mi, T., Zhu, H., Yuan, J., Zhu, J. (2018a). Inhibition of cIAP1 as a strategy for targeting c-MYC-driven oncogenic activity. Proc Natl Acad Sci USA, 115, E9317-E9324. [CrossRef] [Google Scholar]
- Li, X., Elmira, E., Rohondia, S., Wang, J., Liu, J., Dou, Q.P. (2018b). A patent review of the ubiquitin ligase system: 2015-2018. Expert Opin Ther Pat, 28, 919-937. [CrossRef] [PubMed] [Google Scholar]
- Malecka, K.A., Fera, D., Schultz, D.C., Hodawadekar, S., Reichman, M., Donover, P.S., Murphy, M.E., Marmorstein, R. (2014). Identification and characterization of small molecule human papillomavirus E6 inhibitors. ACS Chem Biol, 9, 1603-1612. [CrossRef] [PubMed] [Google Scholar]
- Mattiroli, F., Vissers, J.H., van Dijk, W.J., Ikpa, P., Citterio, E., Vermeulen, W., Marteijn, J.A., Sixma, T.K. (2012). RNF168 ubiquitinates K13-15 on H2A/H2AX to drive DNA damage signaling. Cell, 150, 1182-1195. [CrossRef] [PubMed] [Google Scholar]
- Maxwell, P.H., Wiesener, M.S., Chang, G.W., Clifford, S.C., Vaux, E.C., Cockman, M.E., Wykoff, C.C., Pugh, C.W., Maher, E.R., Ratcliffe, P.J. (1999). The tumour suppressor protein VHL targets hypoxia-inducible factors for oxygen-dependent proteolysis. Nature, 399, 271-275. [CrossRef] [PubMed] [Google Scholar]
- Metzger, M.B., Pruneda, J.N., Klevit, R.E., Weissman, A.M. (2014). RING-type E3 ligases: master manipulators of E2 ubiquitin-conjugating enzymes and ubiquitination. Biochim Biophys Acta, 1843, 47-60. [CrossRef] [PubMed] [Google Scholar]
- Michelle, C., Vourc’h, P., Mignon, L., Andres, C.R. (2009). What was the set of ubiquitin and ubiquitin-like conjugating enzymes in the eukaryote common ancestor ? J Mol Evol, 68, 616-628. [CrossRef] [PubMed] [Google Scholar]
- Montesinos, P., Beckermann, B.M., Catalani, O., Esteve, J., Gamel, K., Konopleva, M.Y., Martinelli, G., Monnet, A., Papayannidis, C., Park, A., Récher, C., Rodríguez-Veiga, R., Röllig, C., Vey, N., Wei, A.H., Yoon, S.-S., Fenaux, P. (2020). MIRROS: a randomized, placebo-controlled, Phase III trial of cytarabine ± idasanutlin in relapsed or refractory acute myeloid leukemia. Future Oncol, 16, 807-815. [CrossRef] [PubMed] [Google Scholar]
- Mund, T., Lewis, M.J., Maslen, S., Pelham, H.R. (2014). Peptide and small molecule inhibitors of HECT-type ubiquitin ligases. Proc Natl Acad Sci USA, 111, 16736-16741. [CrossRef] [Google Scholar]
- Murayama, K., Kato-Murayama, M., Itoh, Y., Miyazono, K., Miyazawa, K., Shirouzu, M. (2020). Structural basis for inhibitory effects of Smad7 on TGF-beta family signaling. J Struct Biol, 212, 107661. [CrossRef] [PubMed] [Google Scholar]
- Nakasone, M.A., Livnat-Levanon, N., Glickman, M.H., Cohen, R.E., Fushman, D. (2013). Mixed-linkage ubiquitin chains send mixed messages. Structure, 21, 727-740. [CrossRef] [PubMed] [Google Scholar]
- Namuduri, A.V., Heras, G., Lauschke, V.M., Vitadello, M., Traini, L., Cacciani, N., Gorza, L., Gastaldello, S. (2020). Expression of SUMO enzymes is fiber type dependent in skeletal muscles and is dysregulated in muscle disuse. FASEB J, 34, 2269-2286. [CrossRef] [PubMed] [Google Scholar]
- Nie, M., Boddy, M.N. (2016). Cooperativity of the SUMO and ubiquitin pathways in genome stability. Biomolecules, 6, 14. [CrossRef] [PubMed] [Google Scholar]
- Offensperger, F., Muller, F., Jansen, J., Hammler, D., Gotz, K.H., Marx, A., Sirois, C.L., Chamberlain, S.J., Stengel, F., Scheffner, M. (2020). Identification of small-molecule activators of the ubiquitin ligase E6AP/UBE3A and Angelman syndrome-derived E6AP/UBE3A variants. Cell Chem Biol, 27, 1510-1520, e1516. [CrossRef] [PubMed] [Google Scholar]
- Olzmann, J.A., Chin, L.S. (2008). Parkin-mediated K63-linked polyubiquitination: a signal for targeting misfolded proteins to the aggresome-autophagy pathway. Autophagy, 4, 85-87. [CrossRef] [PubMed] [Google Scholar]
- Pancheri, E., Guglielmi, V., Wilczynski, G.M., Malatesta, M., Tonin, P., Tomelleri, G., Nowis, D., Vattemi, G. (2020). Non-hematologic toxicity of bortezomib in multiple myeloma: the neuromuscular and cardiovascular adverse effects. Cancers (Basel), 12, https://doi.org/10.3390/cancers12092540. [CrossRef] [Google Scholar]
- Pao, K.C., Wood, N.T., Knebel, A., Rafie, K., Stanley, M., Mabbitt, P.D., Sundaramoorthy, R., Hofmann, K., van Aalten, D.M.F., Virdee, S. (2018). Activity-based E3 ligase profiling uncovers an E3 ligase with esterification activity. Nature, 556, 381-385. [CrossRef] [PubMed] [Google Scholar]
- Park, C.W., Ryu, K.Y. (2014). Cellular ubiquitin pool dynamics and homeostasis. BMB Rep, 47, 475-482. [CrossRef] [PubMed] [Google Scholar]
- Peris-Moreno, D., Cussonneau, L., Combaret, L., Polge, C., Taillandier, D. (2021). Ubiquitin ligases at the heart of skeletal muscle atrophy control. Molecules, 26, 407. [CrossRef] [Google Scholar]
- Petrucelli, L., Dickson, D., Kehoe, K., Taylor, J., Snyder, H., Grover, A., De Lucia, M., McGowan, E., Lewis, J., Prihar, G., Kim, J., Dillmann, W.H., Browne, S.E., Hall, A., Voellmy, R., Tsuboi, Y., Dawson, T.M., Wolozin, B., Hardy, J., Hutton, M. (2004). CHIP and Hsp70 regulate tau ubiquitination, degradation and aggregation. Hum Mol Genet, 13, 703-714. [CrossRef] [PubMed] [Google Scholar]
- Pines, J. (2011). Cubism and the cell cycle: the many faces of the APC/C. Nat Rev Mol Cell Biol, 12, 427-438. [CrossRef] [PubMed] [Google Scholar]
- Polge, C., Attaix, D., Taillandier, D. (2015). Role of E2-Ub-conjugating enzymes during skeletal muscle atrophy. Front Physiol, 6, 59. [CrossRef] [PubMed] [Google Scholar]
- Polge, C., Heng, A.E., Jarzaguet, M., Ventadour, S., Claustre, A., Combaret, L., Bechet, D., Matondo, M., Uttenweiler-Joseph, S., Monsarrat, B., Attaix, D., Taillandier, D. (2011). Muscle actin is polyubiquitinylated in vitro and in vivo and targeted for breakdown by the E3 ligase MuRF1. FASEB J, 25, 3790-3802. [CrossRef] [PubMed] [Google Scholar]
- Polge, C., Uttenweiler-Joseph, S., Leulmi, R., Heng, A.E., Burlet-Schiltz, O., Attaix, D., Taillandier, D. (2013). Deciphering the ubiquitin proteome: limits and advantages of high throughput global affinity purification-mass spectrometry approaches. Int J Biochem Cell Biol, 45, 2136-2146. [CrossRef] [PubMed] [Google Scholar]
- Polge, C., Cabantous, S., Deval, C., Claustre, A., Hauvette, A., Bouchenot, C., Aniort, J., Bechet, D., Combaret, L., Attaix, D., Taillandier, D. (2018). A muscle-specific MuRF1-E2 network requires stabilization of MuRF1-E2 complexes by telethonin, a newly identified substrate. J Cachexia Sarcopenia Muscle, 9, 129-145. [CrossRef] [PubMed] [Google Scholar]
- Quartararo, A.J., Gates, Z.P., Somsen, B.A., Hartrampf, N., Ye, X., Shimada, A., Kajihara, Y., Ottmann, C., Pentelute, B.L. (2020). Ultra-large chemical libraries for the discovery of high-affinity peptide binders. Nat Commun, 11, 3183. [CrossRef] [PubMed] [Google Scholar]
- Quirit, J.G., Lavrenov, S.N., Poindexter, K., Xu, J., Kyauk, C., Durkin, K.A., Aronchik, I., Tomasiak, T., Solomatin, Y.A., Preobrazhenskaya, M.N., Firestone, G.L. (2017). Indole-3-carbinol (I3C) analogues are potent small molecule inhibitors of NEDD4-1 ubiquitin ligase activity that disrupt proliferation of human melanoma cells. Biochem Pharmacol, 127, 13-27. [CrossRef] [PubMed] [Google Scholar]
- Rizk, J., Kaplinsky, J., Agerholm, R., Kadekar, D., Ivars, F., Agace, W.W., Wong, W.W., Szucs, M.J., Myers, S.A., Carr, S.A., Waisman, A., Bekiaris, V. (2019). SMAC mimetics promote NIK-dependent inhibition of CD4(+) TH17 cell differentiation. Sci Signal, 12, https://doi.org/10.1126/scisignal.aaw3469. [CrossRef] [PubMed] [Google Scholar]
- Rotin, D., Kumar, S. (2009). Physiological functions of the HECT family of ubiquitin ligases. Nat Rev Mol Cell Biol, 10, 398-409. [CrossRef] [PubMed] [Google Scholar]
- Rubio, I., Rodriguez-Navarro, J.A., Tomas-Zapico, C., Ruiz, C., Casarejos, M.J., Perucho, J., Gomez, A., Rodal, I., Lucas, J.J., Mena, M.A., García de Yébenes, J. (2009). Effects of partial suppression of parkin on huntingtin mutant R6/1 mice. Brain Res, 1281, 91-100. [CrossRef] [PubMed] [Google Scholar]
- Rui, L., Yuan, M., Frantz, D., Shoelson, S., White, M.F. (2002). SOCS-1 and SOCS-3 block insulin signaling by ubiquitin-mediated degradation of IRS1 and IRS2. J Biol Chem, 277, 42394-42398. [CrossRef] [PubMed] [Google Scholar]
- Sarcevic, B., Mawson, A., Baker, R.T., Sutherland, R.L. (2002). Regulation of the ubiquitin-conjugating enzyme hHR6A by CDK-mediated phosphorylation. EMBO J, 21, 2009-2018. [CrossRef] [PubMed] [Google Scholar]
- Scott, K., Hayden, P.J., Will, A., Wheatley, K., Coyne, I. (2016). Bortezomib for the treatment of multiple myeloma. Cochrane Database Syst Rev, 4, CD010816. [PubMed] [Google Scholar]
- Seth, B., Yadav, A., Agarwal, S., Tiwari, S.K., Chaturvedi, R.K. (2017). Inhibition of the transforming growth factor-beta/SMAD cascade mitigates the anti-neurogenic effects of the carbamate pesticide carbofuran. J Biol Chem, 292, 19423-19440. [CrossRef] [PubMed] [Google Scholar]
- Sharma, A., Alswillah, T., Singh, K., Chatterjee, P., Willard, B., Venere, M., Summers, M.K., Almasan, A. (2018). USP14 regulates DNA damage repair by targeting RNF168-dependent ubiquitination. Autophagy, 14, 1976-1990. [CrossRef] [PubMed] [Google Scholar]
- Skalniak, L., Surmiak, E., Holak, T.A. (2019). A therapeutic patent overview of MDM2/X-targeted therapies (2014–2018). Expert Opin Ther Pat, 29, 151-170. [CrossRef] [PubMed] [Google Scholar]
- Sluimer, J., Distel, B. (2018). Regulating the human HECT E3 ligases. Cell Mol Life Sci, 75, 3121-3141. [CrossRef] [PubMed] [Google Scholar]
- Song, J.J., Szczepanski, M.J., Kim, S.Y., Kim, J.H., An, J.Y., Kwon, Y.T., Alcala, M.A. Jr., Bartlett, D.L., Lee, Y.J. (2010). c-Cbl-mediated degradation of TRAIL receptors is responsible for the development of the early phase of TRAIL resistance. Cell Signal, 22, 553-563. [CrossRef] [PubMed] [Google Scholar]
- Song, R., Peng, W., Zhang, Y., Lv, F., Wu, H.K., Guo, J., Cao, Y., Pi, Y., Zhang, X., Jin, L., Zhang, M, Jiang, P, Liu, F, Meng, S, Zhang, X, Jiang, P, Cao, CM, Xiao, RP. (2013). Central role of E3 ubiquitin ligase MG53 in insulin resistance and metabolic disorders. Nature, 494, 375-379. [CrossRef] [PubMed] [Google Scholar]
- Soss, S.E., Klevit, R.E., Chazin, W.J. (2013). Activation of UbcH5c∼Ub is the result of a shift in interdomain motions of the conjugate bound to U-box E3 ligase E4B. Biochemistry, 52, 2991-2999. [CrossRef] [PubMed] [Google Scholar]
- Spratt, D.E., Martinez-Torres, R.J., Noh, Y.J., Mercier, P., Manczyk, N., Barber, K.R., Aguirre, J.D., Burchell, L., Purkiss, A., Walden, H., Shaw, G.S. (2013). A molecular explanation for the recessive nature of parkin-linked Parkinson’s disease. Nat Commun, 4, 1983. https://doi.org/10.1038/ncomms2983. [CrossRef] [PubMed] [Google Scholar]
- Sugeno, N., Hasegawa, T., Tanaka, N., Fukuda, M., Wakabayashi, K., Oshima, R., Konno, M., Miura, E., Kikuchi, A., Baba, T., Anan, T., Nakao, M., Geisler, S., Aoki, M., Takeda, A. (2014). Lys-63-linked ubiquitination by E3 ubiquitin ligase Nedd4-1 facilitates endosomal sequestration of internalized alpha-synuclein. J Biol Chem, 289, 18137-18151. [CrossRef] [PubMed] [Google Scholar]
- Taillandier, D., Polge, C. (2019). Skeletal muscle atrogenes: From rodent models to human pathologies. Biochimie, 166, 251-269. [CrossRef] [PubMed] [Google Scholar]
- Takedachi, A., Saijo, M., Tanaka, K. (2010). DDB2 complex-mediated ubiquitylation around DNA damage is oppositely regulated by XPC and Ku and contributes to the recruitment of XPA. Mol Cell Biol, 30, 2708-2723. [CrossRef] [PubMed] [Google Scholar]
- Tan, W., van Twest, S., Leis, A., Bythell-Douglas, R., Murphy, V.J., Sharp, M., Parker, M.W., Crismani, W., Deans, A.J. (2020). Monoubiquitination by the human Fanconi anemia core complex clamps FANCI:FANCD2 on DNA in filamentous arrays. Elife, 9, https://doi.org/10.7554/eLife.54128. [PubMed] [Google Scholar]
- Tian, M., Zeng, T., Liu, M., Han, S., Lin, H., Lin, Q., Li, L., Jiang, T., Li, G., Lin, H., Jiang, T., Li, G., Lin, H., Zhang, T., Kang, Q., Deng, X., Wang, H.-R. (2019). A cell-based high-throughput screening method based on a ubiquitin-reference technique for identifying modulators of E3 ligases. J Biol Chem, 294, 2880-2891. [CrossRef] [PubMed] [Google Scholar]
- Tintignac, L.A., Lagirand, J., Batonnet, S., Sirri, V., Leibovitch, M.P., Leibovitch, S.A. (2005). Degradation of MyoD mediated by the SCF (MAFbx) ubiquitin ligase. J Biol Chem, 280, 2847-2856. [CrossRef] [PubMed] [Google Scholar]
- Tomko, R.J. Jr., Hochstrasser, M. (2013). Molecular architecture and assembly of the eukaryotic proteasome. Annu Rev Biochem, 82, 415-445. [CrossRef] [PubMed] [Google Scholar]
- Trotman, L.C., Wang, X., Alimonti, A., Chen, Z., Teruya-Feldstein, J., Yang, H., Pavletich, N.P., Carver, B.S., Cordon-Cardo, C., Erdjument-Bromage, H., Tempst, P, Chi, SG, Kim, HJ, Misteli, T, Jiang, X, Pandolfi, PP. (2007). Ubiquitination regulates PTEN nuclear import and tumor suppression. Cell, 128, 141-156. [CrossRef] [PubMed] [Google Scholar]
- van Wijk, S.J., Timmers, H.T. (2010). The family of ubiquitin-conjugating enzymes (E2s): deciding between life and death of proteins. FASEB J, 24, 981-993. [CrossRef] [PubMed] [Google Scholar]
- Vassilev, L.T., Vu, B.T., Graves, B., Carvajal, D., Podlaski, F., Filipovic, Z., Kong, N., Kammlott, U., Lukacs, C., Klein, C., Fotouhi, N., Liu, E.A. (2004). In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science, 303, 844-848. [CrossRef] [PubMed] [Google Scholar]
- Velasco, R., Alberti, P., Bruna, J., Psimaras, D., Argyriou, A.A. (2019). Bortezomib and other proteosome inhibitors-induced peripheral neurotoxicity: From pathogenesis to treatment. J Peripher Nerv Syst, 24(Suppl 2), S52-S62. [CrossRef] [PubMed] [Google Scholar]
- Wade, M., Li, Y.C., Wahl, G.M. (2013). MDM2, MDMX and p53 in oncogenesis and cancer therapy. Nat Rev Cancer, 13, 83-96. [CrossRef] [PubMed] [Google Scholar]
- Wagner, S.A., Beli, P., Weinert, B.T., Nielsen, M.L., Cox, J., Mann, M., Choudhary, C. (2011). A proteome-wide, quantitative survey of in vivo ubiquitylation sites reveals widespread regulatory roles. Mol Cell Proteomics, 10, M111 013284. [CrossRef] [PubMed] [Google Scholar]
- Walsh, M.C., Kim, G.K., Maurizio, P.L., Molnar, E.E., Choi, Y. (2008). TRAF6 autoubiquitinylation-independent activation of the NFkappaB and MAPK pathways in response to IL-1 and RANKL. PLoS One, 3, e4064. [CrossRef] [PubMed] [Google Scholar]
- Wang, B., Merillat, S.A., Vincent, M., Huber, A.K., Basrur, V., Mangelberger, D., Zeng, L., Elenitoba-Johnson, K., Miller, R.A., Irani, D.N., Andrzej A., Dlugosz, A.A., Schnell, S., Scaglione, K.M., Paulson, H.L. (2016). Loss of the ubiquitin-conjugating enzyme UBE2W results in susceptibility to early postnatal lethality and defects in skin, immune, and male reproductive systems. J Biol Chem, 291, 3030-3042. [CrossRef] [PubMed] [Google Scholar]
- Wang, M., Guo, L., Wu, Q., Zeng, T., Lin, Q., Qiao, Y., Wang, Q., Liu, M., Zhang, X., Ren, L, Zhang, S., Pei, Y., Yin, Z., Ding, F., Wang, H.R. (2014). ATR/Chk1/Smurf1 pathway determines cell fate after DNA damage by controlling RhoB abundance. Nat Commun, 5, 4901. [CrossRef] [PubMed] [Google Scholar]
- Wang, S., Adrianto, I., Wiley, G.B., Lessard, C.J., Kelly, J.A., Adler, A.J., Glenn, S.B., Williams, A.H., Ziegler, J.T., Comeau, M.E., Marion, M.C. Wakeland, B.E., Liang, C., Kaufman, K.M., Guthridge, J.M., Alarcón-Riquelme, M.E., BIOLUPUS and GENLES Networks ; Alarcón, G.S., Anaya, J.-M., Bae, S.-C., Kim, J.-H., Joo, Y.B., Boackle, S.A., Brown, E.E., Petri, M.A., Ramsey-Goldman, R., Reveille, J.D., Vilá, L.M., Criswell, L.A., Edberg, J.C., Freedman, B.I., Gilkeson, G.S., Jacob, C.O., James, J.A., Kamen, D.L., Kimberly, R.P., Martin, J., Merrill, J.T., Niewold, T.B., Pons-Estel, B.A., Scofield, R.H., Stevens, A.M., Tsao, B.P., Vyse, T.J., Langefeld, C.D., Harley, J.B., Wakeland, E.K., Moser, K.L., Montgomery, C.G., Gaffney, P.M. (2012). A functional haplotype of UBE2L3 confers risk for systemic lupus erythematosus. Genes Immun, 13, 380-387. [CrossRef] [PubMed] [Google Scholar]
- Wang, S., Zhao, Y., Aguilar, A., Bernard, D., Yang, C.Y. (2017). Targeting the MDM2-p53 protein-protein interaction for new cancer therapy: progress and challenges. Cold Spring Harb Perspect Med, 7(5): a026245. [CrossRef] [PubMed] [Google Scholar]
- Wang, Y., Argiles-Castillo, D., Kane, E.I., Zhou, A., Spratt, D.E. (2020). HECT E3 ubiquitin ligases – Emerging insights into their biological roles and disease relevance. J Cell Sci, 133, https://doi.org/10.1242/jcs.228072. [Google Scholar]
- Watt, J.E., Hughes, G.R., Walpole, S., Monaco, S., Stephenson, G.R., Bulman Page, P.C., Hemmings, A.M., Angulo, J., Chantry, A. (2018). Discovery of small molecule WWP2 ubiquitin ligase inhibitors. Chemistry, 24, 17677-17680. [CrossRef] [PubMed] [Google Scholar]
- Weber, J., Polo, S., Maspero, E. (2019). HECT E3 ligases: a tale with multiple facets. Front Physiol, 10, 370. [CrossRef] [PubMed] [Google Scholar]
- Wenzel, D.M., Stoll, K.E., Klevit, R.E. (2011). E2s: structurally economical and functionally replete. Biochem J, 433, 31-42. [CrossRef] [PubMed] [Google Scholar]
- Witt, A., Vucic, D. (2017). Diverse ubiquitin linkages regulate RIP kinases-mediated inflammatory and cell death signaling. Cell Death Differ, 24, 1160-1171. [CrossRef] [PubMed] [Google Scholar]
- Wu, K., Chong, R.A., Yu, Q., Bai, J., Spratt, D.E., Ching, K., Lee, C., Miao, H., Tappin, I., Hurwitz, J., Zheng, N., Shaw, G.S., Sun, Y., Felsenfeld, D.P., Sanchez, R., Jun-Zheng, N., Pan, Z.-Q. (2016). Suramin inhibits cullin-RING E3 ubiquitin ligases. Proc Natl Acad Sci USA, 113, E2011-2018. [CrossRef] [Google Scholar]
- Wu, W., Koike, A., Takeshita, T., Ohta, T. (2008). The ubiquitin E3 ligase activity of BRCA1 and its biological functions. Cell Div, 3, 1. [CrossRef] [PubMed] [Google Scholar]
- Xia, Y., Pao, G.M., Chen, H.W., Verma, I.M., Hunter, T. (2003). Enhancement of BRCA1 E3 ubiquitin ligase activity through direct interaction with the BARD1 protein. J Biol Chem, 278, 5255-5263. [CrossRef] [PubMed] [Google Scholar]
- Yang, W.L., Zhang, X., Lin, H.K. (2010). Emerging role of Lys-63 ubiquitination in protein kinase and phosphatase activation and cancer development. Oncogene, 29, 4493-4503. [CrossRef] [PubMed] [Google Scholar]
- Zhang, X., Linder, S., Bazzaro, M. (2020a). Drug development targeting the ubiquitin-proteasome system (UPS) for the treatment of human cancers. Cancers (Basel), 12, 902. [Google Scholar]
- Zhang, X., Shi, S., Su, Y., Yang, X., He, S., Yang, X., Wu, J., Zhang, J., Rao, F. (2020b). Suramin and NF449 are IP5K inhibitors that disrupt inositol hexakisphosphate-mediated regulation of cullin-RING ligase and sensitize cancer cells to MLN4924/pevonedistat. J Biol Chem, 295, 10281-10292. [PubMed] [Google Scholar]
- Zheng, N., Shabek, N. (2017). Ubiquitin ligases: structure, function, and regulation. Annu Rev Biochem, 86, 129-157. [PubMed] [Google Scholar]
- Zhu, X., Li, T., Niu, X., Chen, L., Ge, C. (2020). Identification of UBE2T as an independent prognostic biomarker for gallbladder cancer. Oncol Lett, 20, 44. [PubMed] [Google Scholar]
- Zinngrebe, J., Montinaro, A., Peltzer, N., Walczak, H. (2014). Ubiquitin in the immune system. EMBO Rep, 15, 28-45. [PubMed] [Google Scholar]
- Zucchelli, S., Marcuzzi, F., Codrich, M., Agostoni, E., Vilotti, S., Biagioli, M., Pinto, M., Carnemolla, A., Santoro, C., Gustincich, S., Persichetti, F. (2011). Tumor necrosis factor receptor-associated factor 6 (TRAF6) associates with huntingtin protein and promotes its atypical ubiquitination to enhance aggregate formation. J Biol Chem, 286, 25108-25117. [PubMed] [Google Scholar]
- Zuo, C., Sheng, X., Ma, M., Xia, M., Ouyang, L. (2016). ISG15 in the tumorigenesis and treatment of cancer: An emerging role in malignancies of the digestive system. Oncotarget, 7, 74393-74409. [PubMed] [Google Scholar]
Citation de l’article : Taillandier, D. (2021). Contrôle des voies métaboliques par les enzymes E3 ligases : une opportunité de ciblage thérapeutique. Biologie Aujourd’hui, 215, 45-57
Liste des tableaux
De nombreuses pathologies sont liées à une surexpression, une dégradation accrue ou une mutation d’enzymes E3. Ce tableau n’est pas exhaustif car pour les seules maladies neuro-dégénératives, l’implication de 41 E3 différentes a été répertoriée (George et al., 2018).
Liste des figures
 |
Figure 1 Les protéines ciblées par l’UPS sont ubiquitinylées grâce à une cascade enzymatique E1 (1 membre, 2 isoformes), E2 (35 membres), E3 (> 800 membres). L’ubiquitine (Ub), une petite protéine de 8,5 kDa, est activée en présence d’ATP par l’enzyme E1 qui la transfère sur une enzyme E2. Le complexe est ensuite recruté par une enzyme E3 dont le rôle principal est de reconnaître la protéine cible à marquer par l’Ub. Il existe plusieurs familles d’enzyme E3 : les RING-E3 (mono sous-unité ou multi sous-unités) sont les plus nombreuses (> 600) et sont dépourvues d’activité catalytique ; les HECT-E3 reçoivent l’Ub de l’enzyme E2 et la transfèrent sur la protéine cible ; les RBR-E3 possèdent des caractéristiques hybrides grâce à la présence d’un deuxième domaine RING qui permet le transfert de l’Ub sur la protéine cible. |
| Dans le texte | |
 |
Figure 2 Les E3 de type SCF (Skip Cullin F-box Ligase) ou CRL (Cullin RING Ligase) sont des assemblages de plusieurs sous unités différentes dont la base est assurée par les cullines (Cul). Celles-ci interagissent avec une sous-unité acceptrice de l’enzyme E2 et une protéine adaptatrice qui porte la sous-unité réceptrice de la protéine cible (substrat). Le code couleur et les « * » indiquent les combinaisons entre les différentes sous-unités. Par exemple, Cul-1 se lie uniquement à l’adaptateur Skp1 qui porte une des nombreuses protéines de type F-box (par exemple FBXO32), et le récepteur de l’enzyme E2 est la protéine Rbx1/Roc1 ; la dénomination de cette E3 est donc CRL1FBXO32. La spécificité de reconnaissance de la protéine cible est donnée par les protéines réceptrices (> 80) qui sont classées en différentes familles selon le domaine interagissant avec l’adaptateur. La présence de cofacteurs peut être nécessaire pour stabiliser le complexe ou sa liaison avec la protéine cible. |
| Dans le texte | |
 |
Figure 3 Les E3 possèdent de nombreuses surfaces d’interaction avec leurs partenaires, chacune de ces surfaces pouvant faire l’objet d’une stratégie de ciblage pour empêcher l’ubiquitinylation de la protéine cible (substrat). |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.


